서론
최근 한우의 유전자형을 이용한 혈통과 친자여부를 검증하는 사업이 전국적으로 활발하게 진행되고 있는 상황이다. 이는 한우 산업에서 과학적 검증 데이터가 지속적으로 생산되고 축적되고 있다는 것을 의미한다. 정확한 혈통 및 형질의 정보는 축적된 개량의 효과를 더 세밀하게 관찰 및 확인을 할 수 있게 되었다. 대부분의 한우 유전자형 검증 분석은 고능력 암소 축군 조성을 위한 혈통 확인 및 검증과 한우 가축시장에서 혈통에 대한 인증 자료로 활용되고 있다. 전라북도는 2016년부터 현재까지 ʻ고능력 암소 축군 조성 사업’을 시행하면서 우수한 암소 유전자원들의 혈통을 검증하고 관리하고 있으며, 가축거래 시장에서 한우의 유전자 분석을 통한 친자검증 및 혈통인증 절차를 장려하고 지원하고 있다. 특히 가축시장에서 혈통이 검증된 한우 송아지의 경우 그렇지 않은 송아지에 비해 비교적 높은 가격을 형성하고 있으며(Sun et al. 2021), 이는 혈통을 이용해 계산된 유전능력의 신뢰도에 대한 가치라고 할 수 있다.
다양한 사업을 통해 한우의 유전자형 분석 물량은 지속적으로 증가하고 과학적 검증 데이터 역시 지속적으로 축적되고 있지만, 이렇게 축적된 정보들에 대한 종합적인 분석과 보고는 상당히 미흡한 수준이다. 대부분의 정보들은 혈통에 대한 검증과 인증 이후에 더 이상 활용되지 못하고, 데이터베이스 상에 쌓여만 가고 있는 현실이다. 이러한 데이터를 활용하여 유전적 다양성 및 근친도 증가와 같은 이슈를 해결하기 위한 체계적인 개량 전략이 필요한 실정이다. 하여 본 연구에서는 전국한우협회 정읍시지부의 농가들을 대상으로 2018-2022년에 실시된 고능력 암소 축군 조성사업과 한우 가축시장 친자검증 사업에서 생산된 한우 유전자형 정보를 수집하여 유전적 변화 추세 분석과 유전자원의 소실에 대한 학문적 검증 및 개선방향에 대한 제시를 위한 연구를 수행하였다.
재료 및 방법
1. 공시재료
본 연구는 전국한우협회 정읍시지부의 한우농가를 대상으로 2018-2022년까지 분석된 한우의 유전자형 정보를 수집하여 분석에 활용하였다. 총 33,300두의 유전자형 정보를 수집하였으며, 수집된 정보는 두 그룹(Set1, Set2)으로 나누어 분석을 수행하였다. 송아지, 아비, 어미의 유전자형 정보가 모두 확보된 개체들을ʻSet1’으로 분류하였으며, 총 28,952두의 유전자형 정보를 확보하였다. ʻSet2’는 송아지와 아비의 유전자형 정보만을 이용해 분석된 그룹으로 총 4,348 두의 유전자형 정보를 확보하였다. 분석에 활용된 각 그룹별, 연도별 샘플 수에 대한 정보를 Table 1에 제시하였다.
|
Table 1. Number of samples in this study 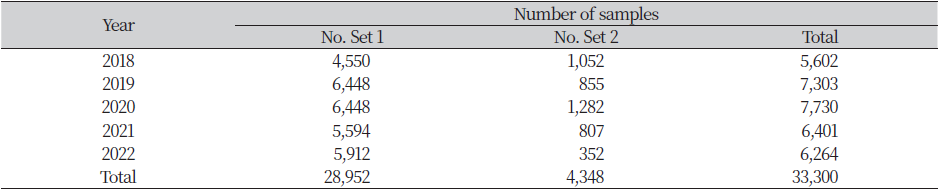
|
|
*Set1: calf and dam were analyzed together, Set2: Only calf individuals were ana-lyzed |
2. Genomic DNA 추출
DNA 추출을 위해 먼저 1.5 mL 튜브에 모근이 붙은 10~15개의 모발(약 0.5~1 cm)을 준비하였다. 준비된 시료에 E-Prep 50 ㎕와 Proteinase K 0.5 ㎕ (20 mg/mL)를 넣고 충분히 vortexing한 후 spin down하여 시약에 모근이 잠기도록 하였다. 이후 65℃에서 20분, 90℃에서 15분 동안 incubation을 실시하였고 실온에서 완전히 식힌 후 deep freezer에 2시간 이상 냉동하였다. 마지막으로 centrifuge를 이용하여 13,000 rpm에서 5분간 원심분리한 후 PCR 증폭용 몰드로 추출한 DNA를 -70℃에 보관하여 사용하였다.
3. MS marker의 정보
PCR 증폭 반응은 형광 염색된 MS marker의 색상과 크기에 따른 대립유전자의 분포를 고려하여 Multiplex PCR로 수행하였다. Multiplex PCR을 수행하기 위한 용액의 조성은 primer mix 8.25㎕, PCR reaction buffer 1.8 ㎕, 10 mM dNTP 1.2 ㎕, Taq DNA polymerase 0.6 ㎕, genomic DNA 1.5 ㎕, ddH2O를 사용하여 총 반응액의 volume을 15 ㎕로 하였다. PCR 반응에는Authorized Thermal Cycler (Eppendorf. Germany)를 사용하였고, PCR 조건의 경우 95℃에서 15분간 예열한 뒤, 94℃에서 1분 denaturation한 후, 56-58℃에서 1분 15초간 annealing, 72℃에서 1분간 extension하였다. 이를 39회 반복을 실시하고, 마지막으로 65℃에서 30초간 진행하여 종결하였다. PCR product를 적정량의 deionized water로 희석하고 DNA:formamide:size standard (500 liz)를 1 ㎕:9.25 ㎕:0.5 ㎕의 비율로 혼합하여 95℃에서 3분간 변성시킨 후 ABI3500xL Genetic Analyzer (Applied Biosystems, USA)와 Gene Mapper version 5.0 (Applied Biosystems. USA)를 이용하여 분석하였다.
2. 통계분석
Microsatellite toolkit s/w(Park. 2001)와 GeneAlEx ver6.5(Peak and Smouse. 2006)를 이용하여 분석 개체별, 집단별 및 연도별로 정리한 후 Cervus ver2.0 program(Marshall. 1998)을 이용하여 기대이형접합율(expected heterozygosity; Hex), 관측이형접합율(observed heterozygosity; Hob), 유전적 다형성 정보지수(polymorphism in formation content; PIC) 등을 계산 및 검정하였다.
결과 및 고찰
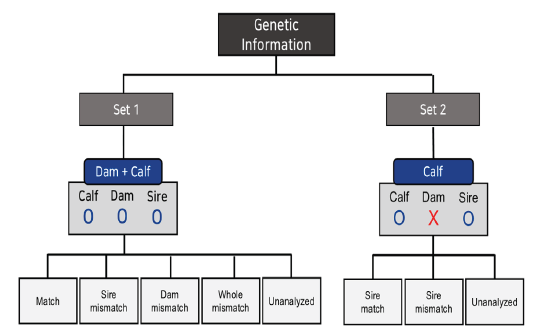
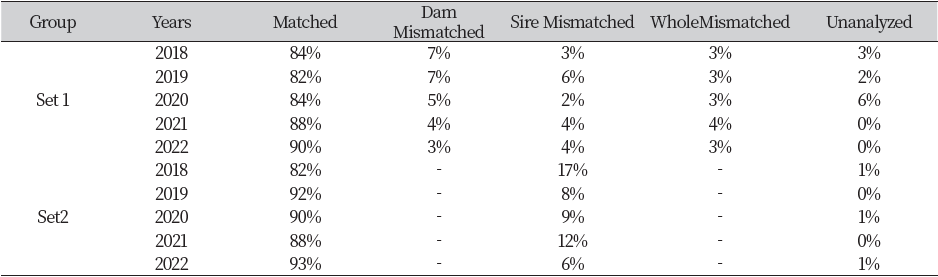
본 연구는 2018-2022년 사이에 전북 정읍지역에서 태어나고 사육된 한우 송아지를 대상으로 혈통확인 친자검증을 위해 분석된 유전자형 결과를 수집하여 분석에 활용하였으며, 각 그룹에 대한 분류 체계를 Fig 1을 통해 설명하였다. Set1에서는 송아지, 어미, 아비(KPN)의 유전자형을 분석했을 때의 결과, Set2에서는 어미의 유전자형 정보가 존재하지 않을 때 송아지와 아비(KPN)의 유전자형을 분석한 것이다. Set1에서 실험을 진행한 최종결과는 일치, 부부정, 모부정, 완전부정, 판단불가로 총 5가지이며, Set2에서는 일치, 부부정, 판단불가로 총 3가지이다. Table 2에서는 두 그룹의 일치율과 불일치율에 대한 정보를 제시하였다. Set1의 결과를 보면 2018년에서 2022년까지 친자일치율이 84%에서 90%로 증가하였고, 부정율이 13%에서 10%로 감소하였다. 전반적으로 꾸준히 친자검정 일치율이 증가하는 추세를 확인할 수 있으며, 판단불가 판정이 확실히 줄어들고 있음을 확인할 수 있었다. 이는 사업의 지속적인 추진을 통해 샘플링 상에서 발생했던 샘플의 오염 등의 사례가 감소한 결과로 추정된다.
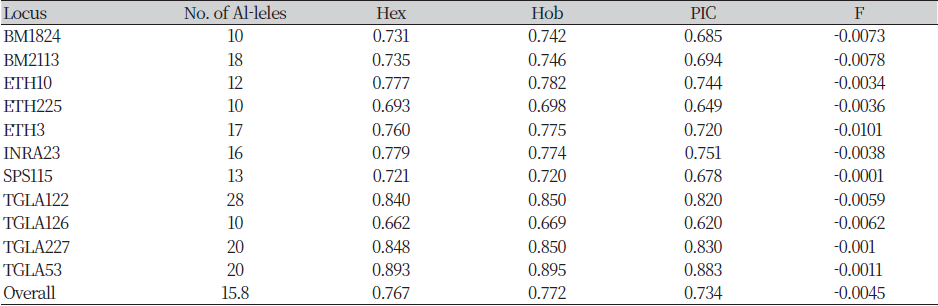
Set 2에서는 2018년 일치율이 82%로 가장 낮은 일치율을 보였고, 2022년도에는 93%의 일치율을 확인할 수 있다. Set1과 Set2 모두에서 일치율이 지속적으로 증가하는 추세를 확인할 수 있었다. 최근 개량을 위한 혈통관리의 중요성과 개량된 송아지들이 도축성적 또한 우수하게 나오고 있어, 유전자형을 이용한 친자검정에 대한 관심이 지속적으로 높아지고 있는 것이 반영된 결과로 추정된다. Table 3에는 총 33,300두의 한우에서 분석된 유전자형 정보를 이용해 유전자 다형성 분석을 실시한 결과를 제시하였다. 마커별 전체 대립유전자의 수, 기대이형접합율(Hex), 관측이형접합율(Hob), 다형정보지수(PIC) 및 F값을 계산하여 제시하였다. 11개의 MS 유전자좌위에서 총 174개의 대립유전자가 확인되었으며, 유전자좌위별 평균 대립유전자의 수는 15.81 개로 확인되었다. 가장 많은 대립유전자의 수는 TGLA12로 28개의 대립유전자가 확인되었다. 또한, BM1824, ETH225, TGLA126에서는 10개로 가장 적게 나타난 것으로 확인되었다. 그리고 집단의 유전적 다양성 분석에 활용되는Hex, Hob 그리고 PIC의 값들을 확인하였다. Hex은 유전적 평형 상태에서 기대이형접합율을 계산한 값이며, Hob은 실제 집단에서 관측이형접합율을 계산한 값이다. 그 결과 Hex의 평균 값은 0.767, Hob의 평균은 0.772로 확인되었다. Hardy-Weinberg Equilibrium에 따르면 Hex보다 Hob의 값이 낮으면 이형접합체 결손 현상이 발생했다고 할 수 있다. 국내에서 오랜 기간 동안 사육되고 개량된 한우의 경우 경제 가축으로써 우수한 종축을 선발하고, 선발된 종축을 이용하여 계획적인 교배가 이루어지기 때문에 Hob값이 Hex 값보다 작아지는 현상이 발생할 수 있다. 하지만 본 연구의 한우집단의 경우 이형접합체 결손 현상이 발생하지 않은 것으로 확인되었다. 이러한 결손 현상은 유전적 다양성 감소와 근친도 증가의 지표로 활용된다. 집단의 유전적 다양성 지표로 활용되고 있는 PIC의 평균은 0.734이며, 가장 낮은 TGLA126의 값은 0.620, 가장 높은 TGLA53의 값은 0.883로 확인되었다. 마지막으로, 분석집단의 F 평균 값은 -0.0045으로, 이는 유전적 다양성과 근친교배 위험이 비교적 잘 관리되고 있음을 의미한다.
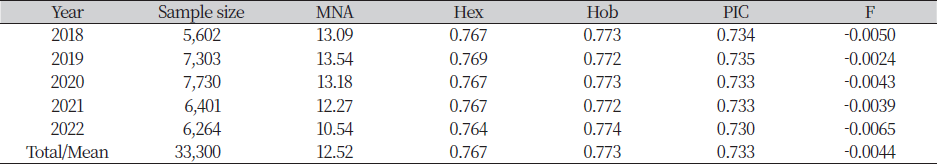
분석된 한우집단의 유전적 특성 및 다양성의 변화를 확인하기 위해 분석된 결과를 연도별(2018, 2019, 2020, 2021, 2022년)로 정리하여 Table 4에 제시하였다. 모든 연도에서 Hob의 값이 Hex 값보다 큰 것을 확인할 수 있었으며, 이는 집단의 유전적 다양성이 유지되고 있음을 의미한다. 하지만 가장 최근인 2022년 집단에서 Hex 값과 PIC 값이 다소 감소한 것이 확인되었으며, 이는 씨수소 집단의 유전적 다양성과 지역 개량사업에 있어 씨수소 쏠림현상 등에 대한 보다 심도 있는 분석이 필요할 것으로 사료된다.
MS marker을 이용한 DNA 검사방법은 혈통확인을 위한 친자검증 뿐만 아니라 개체식별을 위한 분석에도 널리 활용되고 있다(Oh et al. 2008, Shin et al. 2018). 총 11개의 유전자 좌위별 친자검정 일치율과 불일치율을 계산하여 Table 5에 제시하였다. 제시된 결과를 살펴보면 TGLA 53에서 불일치율이 가장 높은 것이 확인되었고, TGLA 126에서 불일치율이 가장 낮은 것이 확인되었다. Table 3에 제시된 결과를 살펴보면, TGLA 53의 이형접합율(Hex, Hob)과 PIC값은 가장 높은 것을 확인할 수 있으며, 반면 TGLA 126은 이형접합율(Hex, Hob)과 PIC값이 가장 낮은 것을 확인할 수 있다. 이는 보증씨수소 집단(Shin et al. 2018)과 칡소 집단(Yu et al. 2021)에서 분석된 결과와 비교하였을 때 가장 높은 다양성을 보이는 유전자 좌위와 가장 낮은 다양성을 보이는 유전자 좌위의 결과가 동일한 것으로 확인되었다. 각 유전자 좌위의 다양성 정보는 친자검정과 개체식별의 정확도에 큰 영향을 미치기 때문에(Barker, 1994) TGLA126의 유전적 다양성 감소에 대한 부분의 지속적인 확인이 필요할 것으로 사료된다.
Table 6은 부부정 판정을 받은 개체들을 대상으로 일치하는 아비의 유전자형을 조사한 결과를 정리하여 제시하였다. 분석된 결과에 따르면 아비와 어미의 유전자형을 보유한 Set1에서는 아비 KPN을 추정할 수 없는 개체가 총 94두(17%)로 확인이 되었고, 아비의 유전자형 정보만 가지고 있는 Set2에서는 24두(5%)로 확인되었다. 이는 자연종부 또는 농협 한우개량사업소에서 생산하는 KPN이 아닌 정액을 사용했을 가능성이 가장 높은 것으로 사료된다. 하나의 KPN을 아비로 추정하는 결과는 Set1에서 305두(58%), Set2에서 36두(7%)가 확인이 되었으며, 4마리 이상의 KPN을 추정한 결과는 Set1에서 44두(8%), Set2에서 275두(58%)로 확인되었다. 따라서 부부정 개체의 아비 추정을 통한 혈통정보 수정을 위해서는 어미의 유전자형 정보가 있을 경우 58% 이상은 하나의 KPN을 특정할 수 있으나, 어미의 유전자형 정보가 없을 경우는 58% 이상이 4개 이상의 KPN을 추정하여 혈통정보 수정이 불가능한 경우가 발생하는 것으로 확인되었다. 개량사업의 추진을 위한 혈통정보 확인이나 친자검정을 위한 분석의 경우, 어미의 유전자형을 가능한 포함하는 것이 혈통정보 오류 수정에 도움이 되며, 개량사업의 효율성 강화에 도움이 될 것으로 판단된다.
|
Table 6. Estimation of the number of KPN for paternal mismatch using microsatellite geno-typing data. 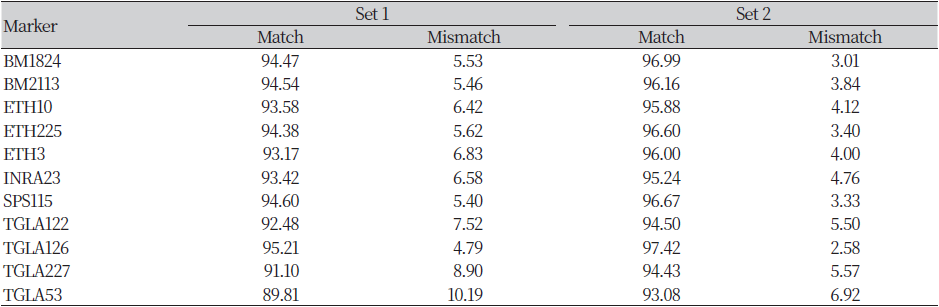
|
최근 한우 산업 축군의 개량을 위한 연구와 다양한 사업이 추진 중에 있다(Sun et al. 2010, Shin et al. 2018, Oh et al. 2021, Kim et al. 2022). 하지만 이러한 연구와 사업을 통해 생산된 데이터들이 다양하게 활용되지 못하고 사장되는 경우가 많은 것이 현실이다. 본 연구는 혈통정보 확인을 위한 친자검정 데이터를 이용해 한우 산업 축군의 유전적 다양성에 대한 모니터링을 실시하고, 개량사업 효율성 강화를 위한 자료를 제시하였다. 지속적으로 확대되고 있는 한우 친자검정 사업을 통해 생산되는 데이터를 이용해 한우 산업을 주기적으로 모니터링하는 연구가 필요할 것으로 판단된다.