INTRODUCTION
한우의 개량사업은 후대검정사업을 통해 보증씨수소를 선발하고, 선발된 보증씨수소의 정액을 전국 한우 농가에 공급하면서 한우산업 전반의 유전적 개량량을 증대시키며 농가 소득향상에 기여하고 있다. 특히 보증씨수소의 선발체계에 유전체를 이용한 유전능력 평가 방법을 활용하면서 평가 정확도가 상승하였고 유전적 개량량 증대에 기여하였다(Park et al. 2019). 농촌진흥청 축산과학원은 한우의 생산능력을 보다 정확하게 예측할 수 있는 한우 유전자칩(Hanwoo 50K SNP BeadChip_v1)을 개발하였다. 이 한우 유전자칩은 기존에 사용되었던 bovine 50K SNP chip에서 한우 품종에서 분석 효율성이 떨어지는 SNP를 제거하고, 한우 품종의 특성을 반영한 SNP와 유전질병에 대한 평가가 가능한 SNP 정보를 추가하여 개발되었다. 그리고 최근에는 도체형질 연관 SNP와 미토콘드리아 SNP 및 유전질병 연관 SNP를 보강한 한우 유전자칩2(Hanwoo 60K SNP BeadChip_v2)를 개발하여 산업현장에 보급 중에 있다. 현재 보증씨수소의 유전체 선발은 한우 유전자칩을 이용해 이루어지고 있다. 또한 보다 정확한 유전능력 평가를 위한 분석모델의 개발 연구와 다양한 축군에 적용을 통한 산업화 연구도 활발하게 진행되고 있다(Lee et al 2019; Lopez et al. 2020; Byun et al. 2021; Kim et al. 2022; Lee et al. 2022).
연관불편형(Linkage Disequilibrium: LD)은 염색세상에 두 대립유전자의 비임의적 연관(non-random association)으로 두개의 다른 대립유전자자가 기대치 이상으로 연관되어 나타나는 것을 의미하며, 양적형질 유전자좌위(QTL)의 위치를 탐색하는데 유용하게 활용되고 있다(Meuwissen & Goddard, 2000). 가축의 유전적 형질과 양적형질 유전자좌위(QTL) 등이 집단 내에서 재조합 될 가능성(유전자 이동, 선발, 돌연변이 등)을 추정하고, 연관불평형정도를 이용한 유전적 구조 및 특성의 분석 연구는 경제형질 개량의 효율성 강화를 위해 매우 중요한 자료를 제공하게 된다. 최근 산업축군에서도 한우 유전자칩을 이용한 유전능력 평가 및 선발을 위한 개량사업이 다양한 환경에서 시작되고 있다. 한우 산업축군의 유전체 선발은 시작단계에 있어 다양한 축군에 대한 분석정보가 아직 충분하지 못한 상태이다. 따라서 본연구는 전라북도의 한우 축군을 대상으로 한우 유전자칩 정보의 연관불평형(LD)을 추정하고 분석하여 지역 한우 축군의 개량을 위한 기초자료로 활용하고자 수행하였다.
MATERIALS AND METHODS
1. 공시재료
본 연구의 공시재료는 전라북도 지역에서 사육되고 있는 1,523두 한우의 꼬리털의 모근에서 채취한 DNA를 이용하였다.
2. 유전자형분석
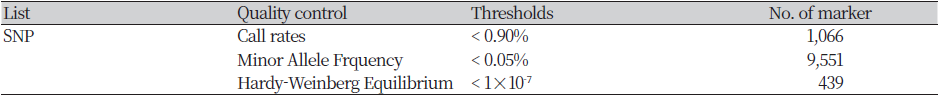
공시재료에서 추출한 DNA Sample은 농촌진흥청 축산과학원에서 개발된 한우유전자칩(Hanwoo 50K SNP BeadChip_v1)을 이용하여 유전자형 분석을 진행하였다. 유전자형 정보를 GenomeStudio 2.0 software (Illumina Inc, 2016)를 사용하여 Final report 파일을 출력하였고, Perl language를 이용하여 PLINK formatting 과정 후 ped, map file을 작성하였다. 유전자형 분석을 통하여 얻은 총 SNP 수는 52,195이며, Quality control (QC)를 진행하여 조건에 부합하지 않는 SNP를 제거하였다(Table 1). QC 기준은 SNP의 Call rate가 90% 미만인 SNP(1,066개), MAF(Minor Allele Frequency)가 1% 미만인 SNP (9,551개), HWE(Hardy-Weinberg Equilibrium)가 1×10-7 미만인 SNP (439개) 정보를 제거하여 최종적으로 41,605개의 SNP 정보를 사용하였다(Table 2).
3. 연관불편형 추정
연관불평형의 크기는 서로 다른 두 대립유전자가 서로 연관되어 유전되는 정도를 나타내는 값으로 먼저 D값을 구한 후에 이를 표준화를 진행한 D’ 또는 r2값을 이용하여 추정이 가능하다. 통계값 D를 이용한 연관불평형 추정은 집단의 크기가 작거나 대립유전자빈도가 작을 경우 과대추정이 일어날 수 있기 때문에 D 값을 이용하여 두 대립유전자 간의 상관을 보여주는 통계량인 r2값을 추정하였다. 두 개의 마커 간 연관불평형을 계산하기 위하여 Plink1.9 program(Purcell et al., 2007)을 이용하여 분석하였다. freq Ai, freq Aj, freq Bi, freq Bj는 두 마커의 대립유전자의 빈도를 나타낸다. D 값은 다음 같은 수식을 이용하여 계산하였다.
D = freq AiBi × freq AjBj− freq AiBj × freq AjBi
동일 염색체 상에 존재하는 두 마커 좌위 A와 B의 연관불평형 크기(r2)는 다음의 수식을 이용하여 계산하였다. 아래의 수식에서 freq AiBi, freq AjBj, freq AiBj, freq AjBi는 두 마커 사이에 대립유전자로 구성된 haplotype 빈도를 나타낸다. 각 분석으로 얻어진 연관불평형 자료는 R software 패키지(The R Project for Statistical Computing, ver.4.1.2; http://www.r-project.org)를 사용하여 수치와 그래프를 나타냈다.
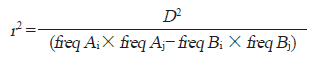
RESULTS AND DISCUSSION
본 연구는 농촌진흥청 축산과학원에서 개발한 한우 유전자칩을 이용해 전라북도에서 사육된 한우 1,523두의 DNA를 공시하여 SNP 유전자형 분석을 실시하였다. 총 52,195개의 SNP의 유전자형이 분석되었고, QC 과정을 통해 11,056개의 SNP가 제거되었으며, 총 41,139개의 SNP가 QC 조건에 부합하여 다음 분석에 이용되었다. 염색체별 SNP표지인자 정보에 대한 요약은 Table 3에 제시하였다. 29개 상염색체의 평균 MAF는 0.27으로 확인되었다. 분석된 결과에서 염색체상 SNP들 간의 거리가 가장 긴 것은 12번 염색체로 확인되었다. SNP간의 평균거리는 0.072Mb으로 확인되었으며 표준편차 역시 가장 큰 것으로 확인되었다(0.12). 가장 짧은 SNP 간의 거리를 가지고 있는 것은 14번 염색체로 확인되었다(0.046 Mb). 14번 염색체의 SNP간의 평균거리의 표준편차는 0.047으로 확인되었다. Lee 등(2021)의 경기 지역에서 사육되어진 한우를 대상으로 연관불평형분석을 수행하여 보고하였으며, 염색체 상에서의 SNP간의 거리 평균이 0.046-0.073 Mb인 것으로 확인하였다. Hong 등(2017)이 보고한 강원도 집단의 한우에서는 0.06-0.082Mb가 확인되었고, 후보종모우와 후대검정우를 대상으로한 Cho 등(2012)의 보고에서는 0.055-0.074Mb으로 확인되었다. 본 연구결과와 가장 유사한 결과는 경기도 지역의 한우 축군이었으며, 강원도 축군과 가장 큰 차이가 확인되었으며, 후보종모우 및 후대검정우 집단과도 차이가 확인되었다. 이러한 차이는 분석된 축군의 크기가 서로 다른 부분에서 기인한 것도 있지만, 가장 큰 이유는 분석에 이용된 유전자칩의 종류에서 기인한 것임을 확인할 수 있다. 축산과학원에서 개발한 한우 유전자칩은 2018년에 개발되어 보급되었기 때문에 그 이전의 연구에서는 Bovine 50K SNP chip을 이용하여 분석하였다.
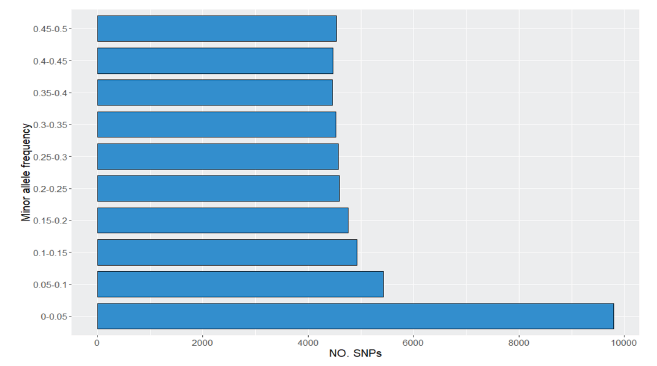
분석에 이용된 SNP의 MAF (Minor Allele Frequency)에 따라 분류하여 분포를 확인하였으며, MAF가 0.05 이하인 SNP를 포함한 총41,139개의 SNP의 분포를 Fig 1에 제시하였다. MAF가 0.05이하의 SNP를 제외하고 나머지 빈도에 분포하는 SNP의 숫자들은 비교적 유사한 것으로 확인되었다. 이는 대체로 SNP 표지인자들의 간격과 대립유전자의 빈도가 일정하게 분포하여 나타난 결과로 확인되어 진다. 최근 유전체 정보를 이용한 유전평가를 위해 특정 형질과 연관된 SNP의 가중치를 조성하여 분석에 활용하는 WGBLUP (weighted genomic best linear unBiased prediction)방법을 통해 평가 정확도를 향상시킨 연구들이 보고되고 있다(Lee et al., 2019; Teissier et al., 2019). 각 SNP 들의 기능적 측면에 대한 고려가 유전평가에 반영이 되면서, 분석의 효율성 강화를 위해 연관불편형 정보의 중요도는 더 높아질 것으로 사료된다.
|
Table 3. ber of markers, map distances and adjacent linkage disequilibrium on 29 autosomes 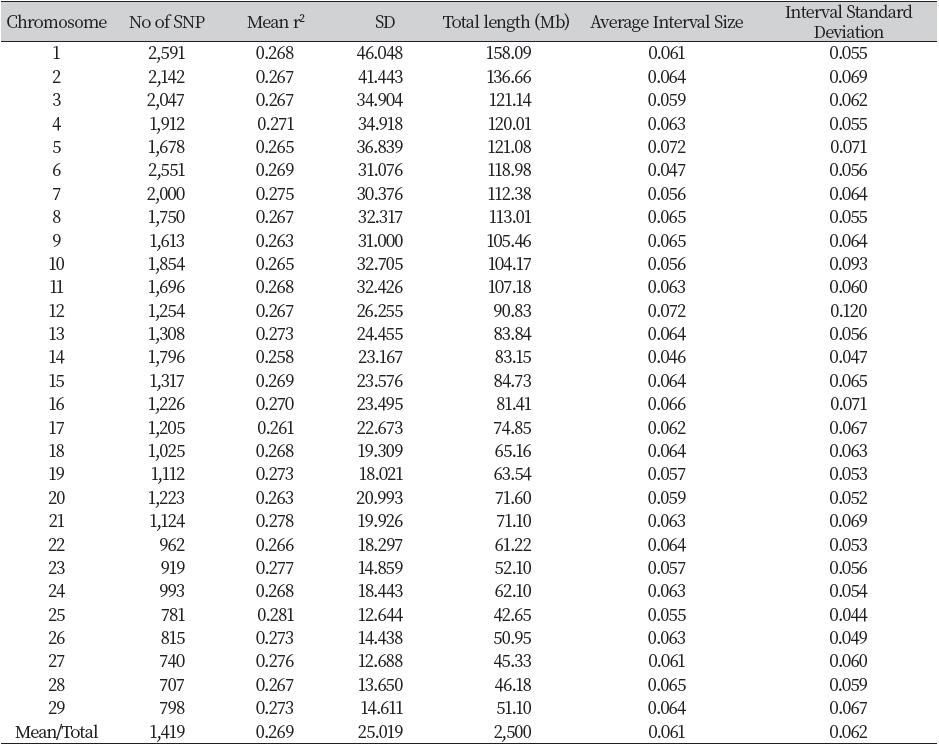
|
|
*SD: Standard deviation |
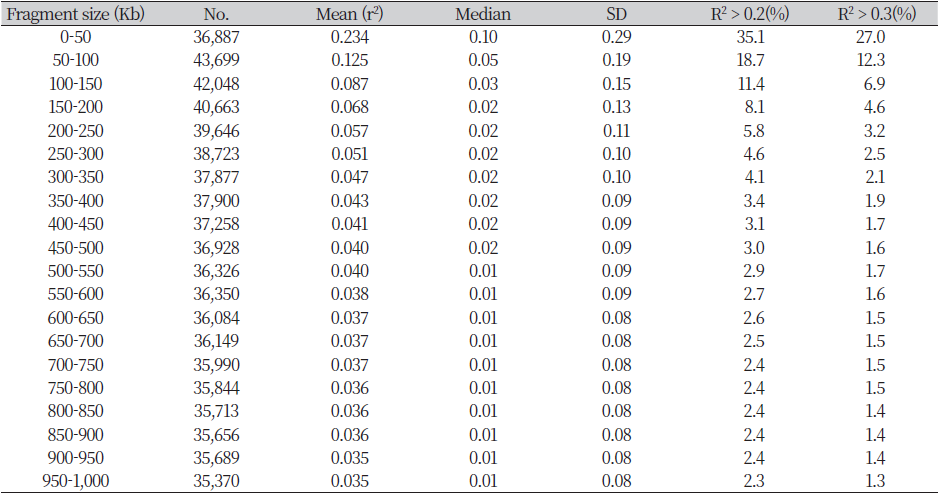
Table 4에서는 SNP 표지인자들을 50Kb 간격으로 나누어 r2 값에 대한 평균 및 표준편차 그리고 거리별 SNP의 개수를 확인하여 제시하였다. 총 750,800개의 SNP 쌍이 고르게 분포되어 있으며, SNP 사이의 거리가 가까울수록 r2 값이 높은 것이 확인되었다. 거리별SNP의 개수는 거리가 850-900 Kb일 때 35,656쌍으로 가장 적었고, 50~100 Kb일 때 43,699으로 가장 많았다. r2 값은 SNP 거리가 0~50 Kb인 경우 0.234, SNP 거리가 150~200 Kb인 경우 0.068로 연관불편형이 급격히 감소하는 현상을 확인하였으며, Fig 2에 그래프로 제시하였다. 서로 다른 한우 집단에서 연관불평형 분석을 보고한 Cho 등(2012), Hong 등(2017) 그리고 Lee 등(2021)의 보고에서도 200 Kb 미만에서 연관불편형이 급격히 감소하는 현상이 나타나고, r2의 값이 200Kb 이후에는 감소하는 것으로 확인되었다.
최근 유전체 정보를 이용한 유전능력 평가 정확도의 상승은 한우산업 전반의 개량량을 증대시키고 개량의 효율성을 강화하는데 큰 역할을 할 것으로 기대되고 있다. 지금까지 한우의 개량사업은 보증씨수소의 선발과 인공수정에 집중된 국가단위의 개량이 중심이 되어왔다. 이는 사육중인 암소의 유전능력을 정확히 평가할 수 있는 방법이 미흡했기 때문이며, 유전체 정보를 이용한 유전능력 평가 기술은 사육중인 암소들의 유전능력을 보다 정확히 평가하고 지역단위, 축군단위의 개량사업 추진을 지원하는 효과적인 기술이 될 것으로 기대된다. 효율적이고 다양한 개량사업의 추진을 위해서 다양한 축군과 환경에서 분석된 유전체 정보가 서로 공유되고 참고된다면, 보다 효율적인 개량 사업의 전략 수립에 큰 도움이 될 것으로 기대된다. 따라서 본 연구는 전라북도 지역의 한우 축군에 대한 유전적인 기초 자료를 제공하고 공유함으로 한우산업의 효율적인 개량 사업 추진에 도움이 되고자 수행하였다.