Introduction
국내에서 사슴은 주로 한약재로 이용 가능한 녹용을 생산하기 위해 엘크(Cervus canadensis; Elk), 붉은 사슴(Cervus elaphus; Red deer), 꽃사슴(Cervus nippon; Sika deer)의 종들이 사육되고 있다. 국산 녹용은 수입 녹용과 비교해 접근성과 가격 경쟁력이 떨어지는데 뉴질랜드, 러시아, 중국 등에서의 대량의 녹용 수입으로 인해 국내 사슴 산업은 위기를 직면했으며, 사슴사육두수는 2001년 156,076마리 이후 해마다 감소하여 2021년 28,464마리로 사슴 산업이 위축되고 있다. 국내에서는 녹용의 둔갑 판매 및 불법유통을 막기 위하여 녹용을 이용한 품종 식별 및 원산지 식별(Lee et al., 2008; Yoo et al., 2010; Kim et al., 2012)과 더불어 녹용의 진위 판별을 위한 연구가 진행되었으나 mitochondrial DNA (mtDNA)를 활용하는 것이 대부분이다(Kim et al., 2000; Shin et al., 2008; Kim et al., 2012).
사슴의 활용은 녹용 뿐만 아니라 사슴 고기도 가능한데, 사슴 고기는 지방이 적어 고급 육으로 인식되어 미국, 유럽 국가에서 활발한 소비가 이루어지고 있으나 국내에서는 사슴 고기를 활용한 연구가 미비하다(Kim et al., 2020; Lee et al., 2021). 따라서 사슴 산물의 다양한 활용 방안과 국내 사슴 산업의 침체를 해결하기 위해서는 국내 사슴의 체계적인 관리를 위한 시스템이 요구된다. Microsatellite (MS)는 1 ~ 6 bp의 짧은 염기서열이 반복되는 것으로 유전적 다형성과 분화율이 높아 계통분류학적 연구에 많이 활용되고 있으며(Ellegren, 2004; Vieira, 2016), 분자 마커 중에서 가장 많이 이용되고 있다(Grover and Sharma, 2016). 이미 한우(Lim et al., 2005), 돼지(Lim et al., 2009), 닭(Suh et al., 2013; Seo et al., 2015)등의 주요 가축에서 MS 마커는 높은 다형성과 재현성을 가지는 공우성 마커 탐색이 가능해 개체 식별, 친자 감정, 유전적 다양성 및 유연 관계 분석 등에 활용되고 있다.
국외에서는 이미 다양한 사슴 종의 식별, 유전적 거리 및 다양성, 계통지리분석(Meredith et al., 2007; Židek et al., 2008; Zsolnai et al., 2009)등에 MS가 활용되고 있다. 그러나 국내에 사육되고 있는 사슴 종을 판별하기에는 부족하다.
Multiplex Polymerase Chain Reaction (PCR)은 다수의 유전자를 한 번에 증폭시킬 수 있으며, MS 마커를 활용한 multiplex PCR 시스템은 적은 비용과 짧은 시간이 걸린다는 장점 때문에 분자유전학뿐만 아니라 법의학, 진단 의학 등 다양한 분야에서 활용되고 있다(Edwards and Gibbs, 1994).
현재 국내 사슴 산업의 실정에 맞는 개체 식별 및 친자 감정에 관한 연구는 미비하다. 따라서 본 연구는 사슴에 활용 가능한 MS 마커를 선발하여 multiplex PCR 시스템을 이용해 개체 식별 및 친자 감정에 활용할 수 있는 MS 마커 세트의 구축을 위해 실시하였다.
Materials and Methods
공시 동물과 DNA
공시 동물은 국립축산과학원 가축유전자원센터(경상남도 함양)에서 사육 및 보존 중인 엘크 51마리, 붉은 사슴 6마리, 꽃사슴 5마리의 혈액을 제공받아 사용하였다. 총 사슴 62마리의 혈액에서 Sucrose-Proteinase K (Birren et al., 1997)법으로 genomic DNA를 추출했으며, NanoDrop 2000 Spectrophotometer (Thermo Fisher Scientific, USA)로 추출한 genomic DNA의 농도 및 순도를 측정하였으며 100 ng/㎕를 분석에 이용하였다.
MS 마커 선발
본 연구에 사용된 MS 마커는 선행연구와 National Center for Biotechnology Information (NCBI; https://www.ncbi.nlm.nih.gov/)을 기초로 33개의 마커를 선발하였다.
MS 마커 세트 설정
33개의 MS 마커 중 single PCR을 수행하여 증폭 효율성이 떨어지는 11개의 마커를 제외하고 22개 마커의 대립유전자 개수, annealing temperature, 이형접합률(Heterozygosity)과 다형성정보지수(Polymorphism Information Content: PIC) 등의 기초 분석을 하였다. 기초 분석을 위한 PCR 조성은 template DNA 100 ng을 1 ㎕, Forward primer와 Reverse primer를 각각 1 ㎕, 10× buffer 1.5 ㎕, dNTP 10 mM을 1.5 ㎕, Hot-start Taq DNA Polymerase 0.1 Unit (Add Bio, Korea)에 증류수를 첨가하여 최종 부피 15 ㎕로 반응시켰다. PCR 조건은 95℃에서 10분간 예비 변성을 한 후 94℃에서 30초, 각 프라이머 조건 온도인 55 ~ 66.2℃에서 30초, 72℃에서 45초를 40회 반복한 후 72℃에서 5분간 추가로 신장시킨 후 8℃에서 종료하였다. PCR 산물은 ABI-3730xl 자동염기서열 분석 장치(Applied Biosystems, USA)를 이용해 전기 영동을 수행하였으며, GeneScan version 5 (Applied Biosystems, USA)를 이용하여 크기와 형광 표식자의 종류별로 분류하여 대립유전자형을 결정하였다. 기초 분석은 CERVUS Ver. 3.0.7 (Marshall et al., 1998)을 이용하였고 Brasher 등(1998)과 Kramer와 Coen (2001)에 따르면 PCR 수행 시 annealing temperature가 55 ~ 60℃일 때 효율적이므로 gradient PCR을 통해 55 ~ 60℃에서 증폭률이 높은 MS 마커를 우선으로 선발, 이형접합률과 다형성정보지수를 이용하여 마커의 다형성을 판단하였다. Multiplex PCR 시스템을 이용하기 위해 마커별 증폭 사이즈를 토대로 최종적으로 9개의 MS 마커를 선발하였으며, 이를 1개의 세트로 조합하여 MS 마커 세트를 설정하였다.
Multiplex PCR 조성 및 방법
Multiplex PCR은 template DNA 100 ng을 2 ㎕, 10× buffer 2.5 ㎕, dNTP 10 mM을 2 ㎕, MS marker primer mixture 8.25 ㎕, Hot-start Taq DNA Polymerase 0.4 Unit (Add Bio, Korea)에 증류수를 첨가하여 최종 부피 25 ㎕로 반응시켰다. Multiplex PCR 조건은 95℃에서 30분간 예비 변성 과정을 거쳐 94℃에서 60초, 59℃에서 60초, 72℃에서 60초를 5회 반복 후, 94℃에서 60초, 60℃에서 60초, 72℃에서 60초를 5회 반복하였다. 마지막으로 94℃에서 60초, 61℃에서 60초, 72℃에서 60초를 20회 반복한 후 65℃에서 30분간 추가로 신장시킨 후 8℃에서 종료하였다.
대립유전자형 분석
Multiplex PCR 산물을 크기별로 분류되도록 ABI-3730xl 자동염기서열 분석 장치(Applied Biosystems, USA)를 이용해 전기 영동을 수행하였으며, 크기와 형광 표식자의 종류별로 분류하기 위해 GeneScan version 5 (Applied Biosystems, USA)를 이용해 대립유전자형을 결정하였다. 분류한 자료를 Microsoft Excel (Microsoft, USA)을 이용해 취합하였다. GeneAlex 6.503 (Peakall and Smouse, 2006)을 이용해 마커별, 품종별로 대립유전자 출현 빈도 변화를 분석하였고, 이를 막대그래프로 도식화 하였다.
통계분석
취합한 자료를 CERVUS Ver. 3.0.7 (Marshall et al., 1998)을 이용해 대립유전자 개수, 기대 이형접합률(Expected heterozygosity: HExp), 관측 이형접합률(Observed heterozygosity: HObs), 다형성정보지수 등의 기초 분석을 하였다. 통계분석에 활용하기 위해 Microsatellite Analyzer (MSA) Ver. 4.05 (Dieringer and Schlötterer, 2003)로 자료의 형태를 변환했으며, GENEPOP Ver. 4.7.3 (Raymond and Rousset, 1995)으로 F-통계량을 추정하였다. 친자 감정 능력 분석을 위해 API-CALC Ver. 1.0 (Ayres and Overall, 2004)으로 무작위 교배 집단, 반형매 교배 집단의 동일개체출현확률을 추정하였고, 부권부정률(non-exclusion probability)은 CERVUS Ver. 3.0.7 (Marshall et al., 1998)의 결과를 이용하였다. MSA로 변환한 자료를 PHYLIP Ver. 3.698 package (Felsenstein, 2004)를 활용하여 Neighbor-Joining (NJ) 방법(Saitou and Nei, 1987)으로 개체별 계통수를 구축하였으며 이를 SplitsTree5 (Huson and Bryant, 2006)로 도식화 하였다. 집단 간의 유연 관계 분석을 위해 Genetix version 4.05 (Belkhir et al., 1994-2004)로 개체별 요인대응분석(Factorial correspondence Analysis: FCA)을 하였다.
Results and Discussion
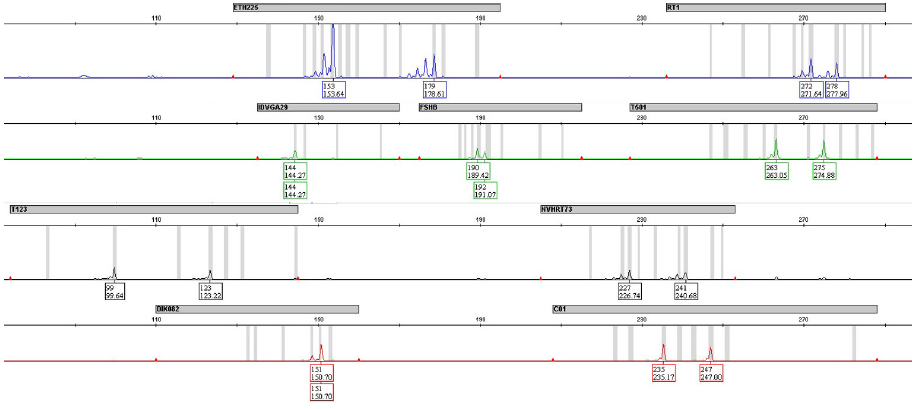
본 연구는 MS를 이용해 국내 사육 중인 사슴의 개체 식별과 친자 감정을 위한 MS 마커 세트 설정을 위해 시행되었다. 사슴의 선행연구와 NCBI를 토대로 33개의 MS 마커를 선발하였으며, 그중 증폭 효율성이 낮은 마커를 제외한 22개 MS 마커의 대립유전자 개수, 이형접합률과 다형성정보지수 등의 기초 분석 결과를 Table 1에 나타내었다. 기초 분석은 마커의 다형성을 평가하기 위한 것으로 이형접합률은 다른 대립유전자형 보유율을 나타내는데 이형접합률이 높을수록 유전적 다양성이 높고 낮을수록 유전적 다양성이 낮다고 판단하며, 다형성정보지수는 DNA 마커의 다형성 판별을 위한 지표로 활용한다. MS 마커 세트를 구성하기 위한 기초 분석 결과, 기대 이형접합률은 0.120 ~ 0.843, 다형성정보지수 값은 0.116 ~ 0.817의 범위이며, 대립유전자의 개수는 4 ~ 13개로 확인되었다. 시간 및 비용의 효율성을 높이기 위해 한 번에 여러 마커의 분석이 가능한 multiplex PCR 시스템에 활용하기 위한 MS 마커 세트 구성을 위해 55 ~ 60℃에서 증폭률이 높은 마커를 우선으로 선발하였다. 또한, 마커의 사이즈와 기대 이형접합률의 값이 0.6 이상, 다형성정보지수 값이 0.5 이상인 마커는 높은 다형성을 가지고 활용하기에 효율적인 마커로 보고되어 있고 다형성정보지수 값이 0.25 ~ 0.5 사이일 경우 활용하기에 적합한 마커라고 보고되어 있기에(Botstein et al., 1980) 기대 이형접합률, 다형성정보지수를 기준으로 하여 MS 마커를 선발하였다(Table 2). MS 마커 세트를 활용하여 사슴의 대립유전자를 결정한 결과를 Figure 1에 나타내었다.
9개 MS 마커의 대립유전자 개수, 이형접합률, 다형성정보지수의 분석 결과는 Table 3에 나타내었다. 62마리의 모든 MS 마커 좌위에서 증폭이 확인되었고 대립유전자의 수가 가장 많은 마커는 ETH225로 13개이고 기대 이형접합률과 다형성정보지수 값은 T501이 0.832, 0.805로 가장 높게 나타났으며, IDVGA29는 대립유전자의 수가 4개로 가장 적고 기대 이형접합률과 다형성정보지수 값이 0.306, 0.276으로 가장 낮은 것을 확인하였다. 9개 MS 마커의 평균 대립유전자 수, 관측 이형접합률, 기대 이형접합률, 다형성정보지수 값은 차례로 8.333, 0.525, 0.643, 0.602이며, 9개 MS 마커의 다형성정보지수는 모두 0.25 이상으로 활용에 적합한 마커로 판단된다.
|
Table 1. Basic analysis of 22 MS markers 
|
|
y HExp: Expected heterozygosity z PIC: Polymorphism Information Content |
|
Table 3. Basic analysis of 9 MS markers of deer utilized in this study 
|
|
x HObs: Observed heterozygosity y HExp: Expected heterozygosity z PIC: Polymorphism Information Content |
Takezaki와 Nei (1996)에 따르면 유전적 다양성 분석에 활용하기 위해서는 이형접합률이 0.3 ~ 0.8을 충족하여야 하는데, IDVGA29의 관측 이형접합률은 0.048, 기대 이형접합률은 0.306으로 앞서 보고된 범위를 벗어나 유전적 다양성 분석에 어려움이 있을 것으로 사료된다. 그러나 본 연구에 활용된 엘크 51마리는 IDVGA29 마커에 있어서 하나의 대립유전자형으로 고정되어 유전적 고착화로 인한 유전적 부동(genetic drift)이 있을 것으로 사료되므로 유전적으로 외부 유입이 있는 엘크 품종의 추가 분석이 된다면 유전적 다양성 분석에 MS 마커 세트의 활용이 가능할 것으로 판단된다.
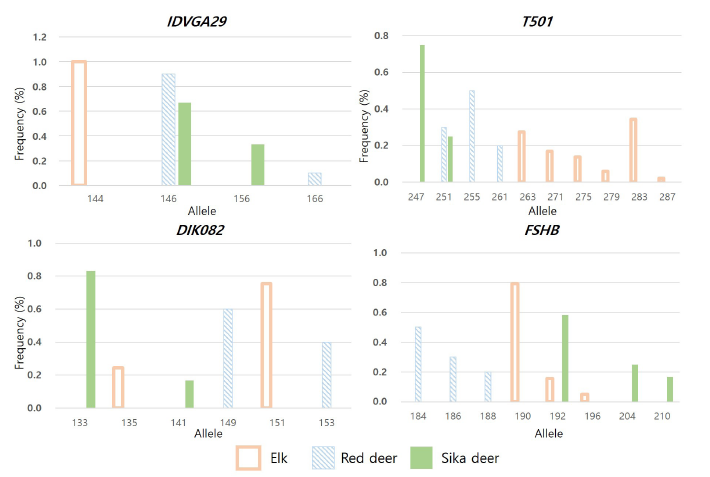
엘크, 붉은 사슴, 꽃사슴의 세 품종에 대하여 품종 식별 마커로서의 유용성 평가를 위해 마커별 대립유전자 빈도 변화를 분석하였으며, 품종별 특이적 대립유전자 출현 마커에 대하여 막대그래프로 도식화 하였다(Figure 2). 대립유전자 빈도 분석 결과, 4가지의 마커(IDVGA29, T501, DIK082, FSHB)에서 종 특이적 증폭을 관찰하였다. IDVGA29 마커에서 엘크 품종이 대립유전자 144 하나로 고정되어 있으며, T501 좌위의 대립유전자 263, 271, 275, 279, 283, 287의 좌위는 엘크 품종 특이성을 보여주는 표지 인자라고 판단된다. FSHB 좌위에서는 엘크 품종이 190, 192, 196에서 나타나며, 붉은 사슴은 184, 186, 188, 꽃사슴 품종은 192, 204, 210에서 대립유전자를 확인하였다. 붉은 사슴은 엘크와 꽃사슴과 비교하였을 때, 특이적 증폭을 보이며 엘크는 192 좌위에서 낮은 증폭을 보였다. 또한, DIK082 좌위의 대립유전자는 모든 품종에 있어서 특이적 대립유전자 출현을 보이는데 엘크 품종은 대립유전자 135와 151에서 확인되며, 붉은 사슴 품종은 대립유전자 149와 153의 좌위에서, 꽃사슴 품종은 대립유전자 133과 141의 좌위에서 대립유전자가 확인되어 분석 품종 모두에 있어 특이적 대립유전자 출현을 보여주어 품종 식별 표지인자로서 활용 가능할 것으로 판단된다.
집단 간 유전 변이와 집단 내 개체들의 근친도 파악을 위해 F-통계량을 추정하였다(Table 4). FST는 유전적 부동에 의한 부분 모집단의 이형접합체 감소 추이를 나타내는 고정 지수이며, FIT는 전체 집단에 대한 개체들의 모든 근친도 값을 내포하는 지수이다. 9개의 MS 마커 평균 FST는 0.616, 평균 FIT는 0.232를 나타내는데, F-통계량은 수치가 높을수록 개체 간의 상관관계가 높음을 의미하며 낮을수록 근친도가 낮음을 의미한다. FST와 FIT가 가장 낮은 마커는 T501로 차례로 0.506, 0.012의 수치를 확인했으며, 가장 높은 마커는 IDVGA29로 0.922, 0.843의 수치를 확인했다. 본 연구에 활용된 MS 마커 세트는 F-통계량 값이 높은 편으로 본 연구에서 활용한 사슴집단의 근친도가 높게 분석되었는데, 이는 IDVGA29 마커의 앞선 MS 마커의 기초 분석 결과와 마찬가지로 유전적 부동에 의한 이형접합체의 감소가 많이 일어난 것으로 엘크 품종에 있어서 추가 분석이 필요하다고 사료된다.
친자 감정 능력을 분석하기 위해 9개의 MS 마커 세트의 동일개체출현율과 부권부정률을 추정하여 나타내었다(Table 5). 그 결과 무작위 교배집단 동일개체출현율은 4.68 × 10-8, 반형매 교배집단 동일개체출현율은 3.23 × 10-6으로 나타났으며 NE-1P는 부모에 대한 정보가 전혀 없는 경우, NE-2P는 한쪽 부모에 대한 정보만 있는 경우를 의미하며, NE-1P는 0.0516621, NE-2P는 0.0045074로 분석되었다. 이는 현재 국내 사슴 사육 규모가 약 100배 증가하여도 활용 가능함을 의미한다.
국외에서 MS를 활용한 사슴 종의 개체 식별에 관한 연구와 비교해보면, 붉은 사슴과 다마 사슴(Fallow deer) 종에 대해 8개의 MS를 2개의 세트로 조합하여 친자 감정 능력을 분석했을 때 한쪽 부모에 대한 정보만 있는 경우의 부권부정률은 0.0071로 보고되어 있으며(Zsolnai et al., 2009), 싯카 검은꼬리사슴(Sitka black-tailed deer)의 개체 식별을 위해 7개의 MS를 활용한 연구에서는 무작위 교배 시 동일개체출현율 0.0003, 전형매 교배시 동일개체출현율 0.021로 보고되어 있다(Brinkman et al., 2010). Miller 등(2019)에 따르면 흰꼬리사슴(White-tailed deer)의 동일개체출현율은 2.915 × 10-17로 이전의 흰꼬리사슴과 노새 사슴(Mule deer)에 사용된 SNP 마커와 비교해도 MS 마커가 비슷한 힘을 가진다고 보고했다. 앞선 연구들에서는 MS 8 ~ 11개를 여러 개의 세트로 조합하여 연구를 수행하였는데, 본 연구에서는 1개의 세트로 조합하여 분석에 따른 비용 및 시간의 효율성을 높였다. 또한, 국내에서 녹용을 목적으로 사육 중인 엘크, 붉은 사슴, 꽃사슴의 품종을 한 번에 식별할 수 있는 연구는 없는 것으로 확인되며, 본 연구와 앞선 다른 사슴 종의 동일개체출현확률과 부권부정률을 비교했을 때, 국내에 사육 중인 사슴의 개체 식별 및 친자 감정에서 본 연구에서 설정한 MS 마커 세트의 활용 가능성이 크다고 판단된다.
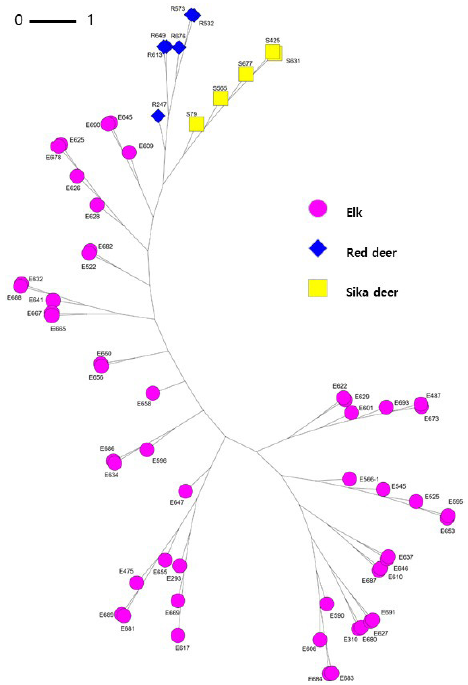
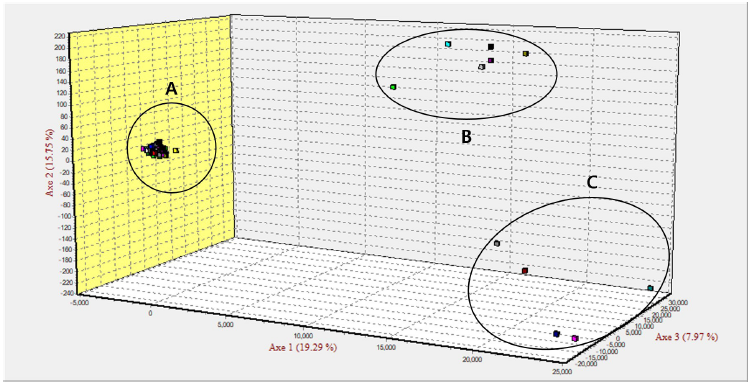
개체별 유연관계 분석을 위해 62마리의 개체별 계통수를 도식화하여 나타내었다(Figure 3). 계통수 분석 시 붉은 사슴과 꽃사슴의 유연관계가 가까운 것으로 보이는데 이는 Randi 등(2001)의 연구결과와 유사하며 Polziehn과 Strobeck (2002)은 붉은 사슴과 엘크의 유전적 거리가 가깝다고 보고했다. 또한, Figure 4에는 개체별 FCA 결과를 3차원으로 도식화 하였으며, 분석 결과, 제1분산치는 19.29%, 제2분산치는 15.75%, 제3분산치는 7.97%로 총 43.01%의 분산치를 나타냈다. 계통수와 FCA 분석을 통해, 엘크, 붉은 사슴, 꽃사슴이 집단별로 군집을 형성하는 것을 확인할 수 있었으나 엘크의 개체수가 붉은 사슴과 꽃사슴의 개체 수보다 압도적이므로 붉은 사슴과 꽃사슴의 개체를 추가적으로 확보해 사슴 유전자원의 다양성 및 유전적 거리를 추정해 볼 필요가 있을 것으로 사료된다.
본 연구에서 확립된 MS 마커 세트를 활용해 국내에 사육되고 있는 사슴의 개체 정보 시스템을 구축하고 친자 감정을 통한 생산이력 시스템의 바탕이 된다면 국산 녹용에 대한 소비자의 신뢰성 향상 및 소비 활성화가 기대되며, 개량을 위한 기초자료로서의 활용도 가능할 것으로 사료된다. 또한, 국내에 사육 중인 엘크, 붉은 사슴, 꽃사슴의 개체를 충분히 확보한다면 추가 분석을 통해 유전적 다양성 및 지리적 유연관계 분석에도 활용될 수 있을 것으로 기대된다.
Conclusion
사슴은 국내에서 한약재로 이용되는 녹용의 생산을 위해 주로 사육되고 있으나 국내 녹용의 자급률은 낮으며, 체계적인 시스템이 갖춰져 있지 않다. 이에 본 연구는 MS를 활용하여 국내에 주로 사육되고 있는 엘크, 붉은 사슴, 꽃사슴의 개체 식별 및 친자 감정을 위한 MS 마커 세트 설정을 위해 실시되었다. 마커의 사이즈와 이형접합률, 다형성정보지수 등 마커의 다형성을 토대로 총 9개의 MS 마커를 선발해 1개의 세트로 설정하였다. 본 연구에서 설정한 MS 마커 세트의 기초 분석 결과, 관측 이형접합률 0.525, 기대 이형접합률 0.643, 다형성정보지수 0.602로 충분히 활용하기에 적합한 마커로 판단된다. 친자 감정 능력 분석 결과, 9개의 MS 마커를 활용할 때 동일한 유전자형을 가진 개체가 출현할 확률은 무작위, 반형매 교배집단에서 각각 4.68 × 10-8, 3.23 × 10-6이며, 부권부정률은 부모에 대한 정보가 전혀 없는 경우, 한쪽 부모에 대한 정보만 있는 경우에 대해 각각 0.0516621, 0.0045074로 분석되어 현재 국내의 사슴 사육 규모가 증가하여도 개체 식별 및 친자 감정에 충분히 활용 가능할 것으로 판단된다. 따라서 개체 식별 및 친자 검정 시스템을 통한 생산이력 시스템 구축 또는 사슴 개량을 위한 기초자료로 활용한다면 국내 사슴 산업의 활성화가 기대된다.