Introduction
전 세계적으로 수산 양식업 생산량은 2001년부터 2018년까지 매년 평균 5.3% 증가하는 추세를 보였으며 (FAO, 2020) 우리나라의 수산 양식업 또한 이와 같이 2012년 약 4억 마리 대비 2020년 4억 4천만 마리로 10%가량 증가했다 (Korea, 2021; Jung et al., 2020). 국내 넙치의 생산량은 연어, 방어와 같은 수입 어종에 의한 소비 트렌드의 변화로 대략 2012년 6천7백만 마리에서 2020년 6천 2백만 마리로 다소 감소하는 경향을 보이고 있음에도, 2018년 전체 어류 생산량의 46.2%(37,240 metric ton)을 차지할 만큼 국내 양식업에서 주요한 어종이다 (Korea, 2021). 넙치 양식업에서 수산생물의 질병으로 인한 폐사피해는 양식 산업의 근간을 흔들 수 있는 원인이 된다 (Shim et al., 2019). 수입 어종과의 경쟁에서 국내 넙치 양식업을 유지하기 위해선 양식과정에서 발생하는 질병에 의한 폐사와 같은 생산량 저감 요인들을 방지해야 한다. 그 중에서도 특히 바이러스성 질병에 의한 감염은 전파속도가 빠르고 숙주 자체의 면역상태를 약화시켜 세균 감염 등 2차 피해를 야기할 수 있기 때문에 더욱 조심해야 한다 (Kang et al., 2012).
바이러스성 출혈성 패혈증 바이러스(Viral Hemorrhagic Septicemia Virus; VHSV)는 저수온 (8-15°C)에서 보다 오랜 기간 감염력을 유지하는 것으로 알려진 RNA 바이러스이다 (Jeong et al., 2019; Tadashi et al., 2001). 해당 바이러스로 인한 질병인 바이러스성 출혈성 패혈증 (viral hemorrhagic septicemia; VHS)은 성어 이전의 단계에서 특히 높은 치사율을 보이긴 하지만, 어류의 모든 연령 대에서 나타날 수 있는 질병이다 (Kim and Faisal, 2011). 급성 침투성 질병인 VHS는 종마다 정도는 다르지만 장기 내외부에서의 출혈을 증상으로 동반하여 심각한 경우 치사할 수 있다 (Tadashi et al., 2001). 국내에선 동해안 넙치 양식장에서 2001년에 처음으로 확인되었으며, 스쿠티카증, 여윔증과 함께 폐사의 주요 원인이다 (Kim et al., 2003; Shim et al., 2019). 이와 같이 국내에서 넙치의 피해를 최소화하기 위해선 바이러스성 질병 중에서도 VHSV에 따른 피해를 줄이는 것이 필수적이다.
한국, 일본, 중국 등 아시아 국가에서 경제적으로 중요한 어종인 넙치는 최근 몇 년 간 면역학과 질병에 관해 광범위하게 연구되었다 (Castaño-Sánchez et al., 2010). 특히 넙치의 VHS 발병에 따른 피해를 최소화하기 위한 선행연구가 진행되어 왔으나 환경에 따른 발병률의 변화, 다른 질병 감염 여부에 따른 치사율 감소 등을 확인한 연구들이 주를 이루어 넙치 자체의 직접적인 질병 저항성에 관한 정보는 아직까지 알려져 있지 않다 (Pakingking et al., 2004; Jeong et al., 2019).
넙치의 유전자 표지정보를 이용한 VHSV 저항성 관련 양적형질 유전 좌위(Quantitative Trait Loci; QTL) 탐색은 양적형질 좌위 내에 위치한 유전자를 탐색하고, 그 유전자로부터 바이러스 저항성과 연관된 유전변이를 검출함으로 마커도움 선발(Marker assisted selection, MAS)이 가능할 것이다. 이러한 바이러스 저항성과 관련된 유전마커를 이용하여 마커도움선발(Marker assisted selection)을 적용한다면, VHSV 저항성을 갖는 개체들을 선발 육종함으로 넙치의 생산량을 증가시킬 수 있다 (Zheng and Sun, 2011). 따라서 본 논문에서는 넙치 VHSV 저항성과 연관된 QTL을 검출하여 넙치 육종에 새로운 육종 방향성을 제시하고자 QTL 분석을 실시하였다.
Materials & Methods
VHSV 저항성 검사 및 가계 선발
VHSV 저항성 검사는 경상남도 거제시의 국립수산과학원 수산종자육종연구소 어류육종센터에서 수행되었다. VHSV에 대한 질병연관 유전자 지도 작성을 위해 1:1 full-sib 넙치 6가계가 생산되었다. 총 3,839 마리로 구성된 6 가계의 넙치를 병원성 바이러스에 감염시켜 54일간 생존율을 조사하였다. 최종 생존율이 50%에 근접한 가계를 선정하고, 그 중 생존기간을 기준으로 고르게 분포된 90마리를 공시재료로 표본추출하였다. 각 표본으로부터 채취된 시료는 96% 에탄올에 저장하였으며, QIAGEN의 DNeasy96 키트를 이용하여 DNA를 추출했다.
넙치 유전자 지도 작성을 위한 유전자형 분석
넙치의 유전체에서 검출된 총 473개의 MS 마커 중 다형성을 보이지 않은 monomorphic 마커, null allele 등 유전자형 분석이 불가능한 마커를 제외한 311개 마커에 대한 넙치 가계의 유전자형 분석을 진행하였다. 넙치 가계의 유전자형 분석에 사용된 넙치 Microsatellite (MS) 마커의 정보는 Table 1과 같다.
유전자 지도 작성
Segregation pattern 검정은 카이 제곱 검정을 기반으로 진행되었으며, 다중 검정에 대한 교정은 본페로니 교정을 사용하였다. Linkage 분석은 Hidden Markov Models (Wu et al., 2002)을 적용하여 multipoint technology를 가능하게 하는 R package, onemap_version2.8.2 (Margarido et al., 2007)를 통해 수행되었다. Linkage group은 LOD score (3) 와 recombination fraction (0.4)를 기준으로 형성되었다. 마커의 위치는 효율적인 결과 도출을 위해 각 그룹이 가진 마커 수가 10개 미만인 경우에는 compare (Lander et al., 1987), 10개 이상인 경우에는 order 알고리즘 (Mollinari et al., 2009)을 각각 이용했다. 마커 사이 재조합 비율은 kosambi mapping function을 이용하여 centiMorgan으로 변환 후 유전자 지도를 작성하였다 (Kosambi, 1943).
QTL 분석
넙치 VHSV 저항성에 대한 QTL mapping은 전형매 가계를 위한 Composite Interval Mapping(CIM)을 적용하여 수행되었다 (Gazaffi et al., 2014). CIM 모델에서, 15 cM의 window size를 고려하여 1 cM 간격에 대해 QTL 유전자형 확률을 얻었다. 각 그룹의 보조인자(cofactor)는 akaike information criterion (AIC) (Akaike, 1974)을 기반으로 다중선형회귀의 stepwise를 통해 구했으며, super-parameterization를 방지하기 위해 파라미터 최대 수를 2√n (n은 개체 수)으로 제한했다. QTL은 LOD 임계값 3을 기준으로 정의되었으며 분석은 R package인 fullsibQTL을 이용하여 수행되었다 (Gazaffi et al., 2014).
Results
넙치 병원성 바이러스(VHSV)에 대한 넙치 가계 생산
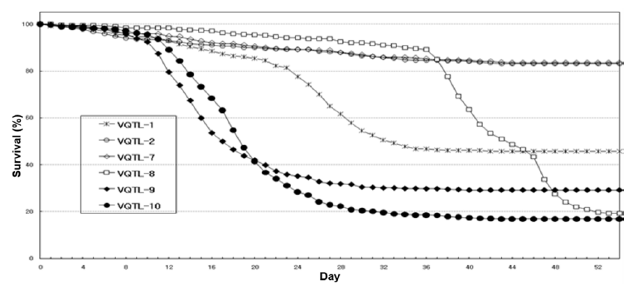
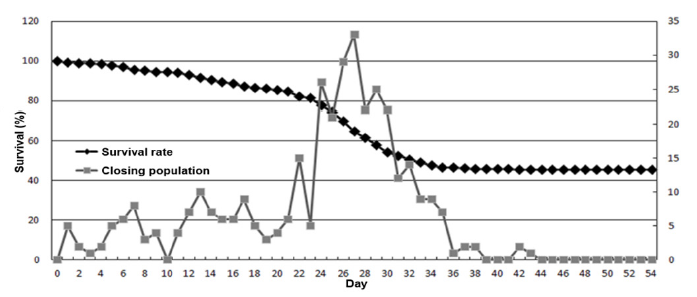
VHSV의 질병 연관 유전자 지도 작성을 위해 넙치 6가계(1:1 전형매)를 생산했다. 총 3,839마리를 병원성 바이러스에 감염시켜 54일 간 생존율을 조사한 결과, 평균 생존일령은 22.8일, 최종 생존 개체 수는 1,633마리로 확인되었으며 42.5%의 생존율을 보였다(Fig. 1). 6 가계 중 VQTL-1 가계 (총 662마리)는 평균 생존일령은 23.5일, 최종 생존 개체 수는 302마리로 확인되었다. 이에 VQTL-1이 최종 생존율 45.6%로 6가계 모두를 가장 대표하기 좋은 가계라고 판단하여 해당 가계를 최종 유전자 지도 작성용 가계로 선정했다(Fig. 2).
유전자 지도 작성
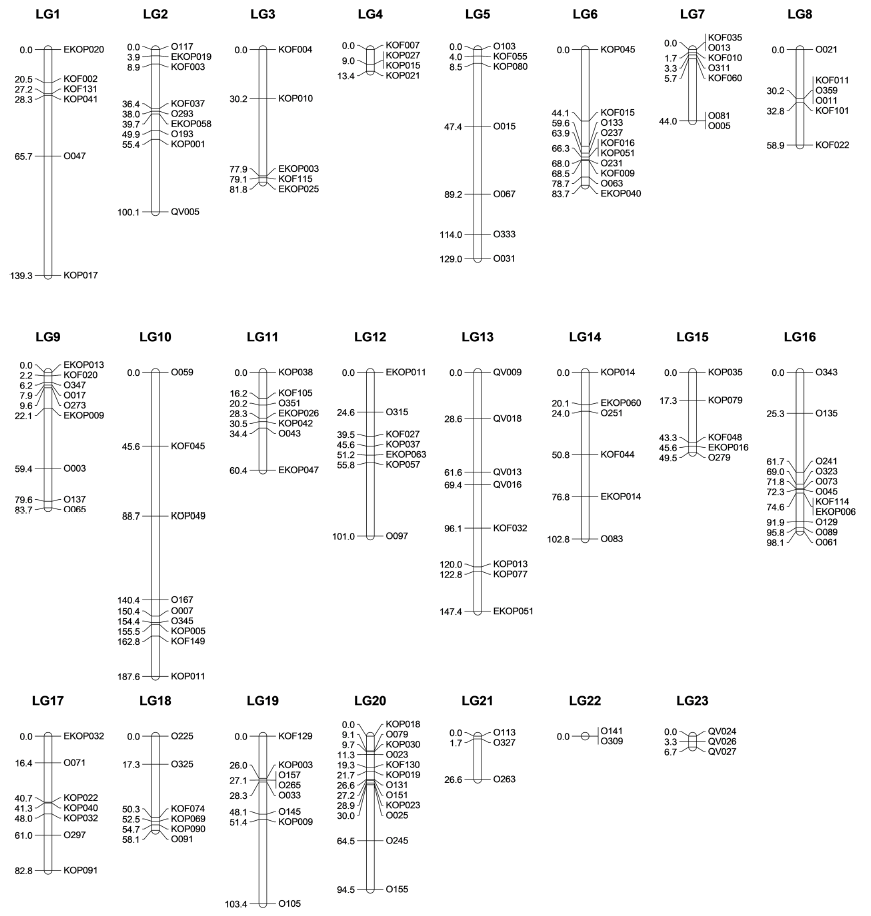
연관 분석은 311개의 MS 마커 중 재조합이 일어나지 않은 55개 마커, segeregation test에서 통계적으로 유의한 6종의 마커를 제외한 총 250개 마커가 linkage group (LG)을 형성하는데 사용되었다. 그 결과, 157개의 MS 마커가 23개의 linkage group을 구성하였다 (Fig. 3). 넙치의 염색체 수는 2n=48로 추정된 바 있고 (Kim et al., 1994), linkage group이 이에 상당히 근사하게 형성되었기 때문에 신뢰할 만한 결과라고 판단하였다. Linkage map의 총 거리는 1852.55 cM, 전체간격평균은 11.53 cM이었다. Genetic distance가 0인 LG22를 제외하면, 각 linkage group의 크기는 6.67 cM (LG23)에서 187.64 cM(LG10)로 다양했으며 LG20은 가장 높은 마커 수(12개)를 보였다. 그룹당 평균 길이는 80.55 cM이었으며 평균 마커 수는 6.74개였다(Table 2).
QTL mapping
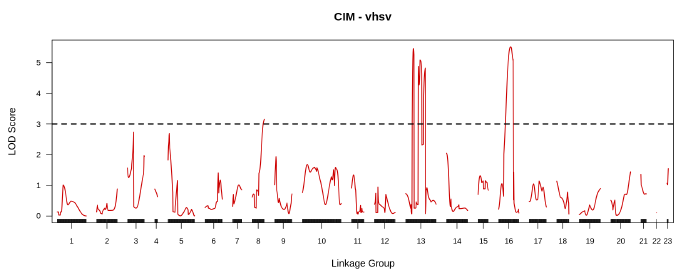
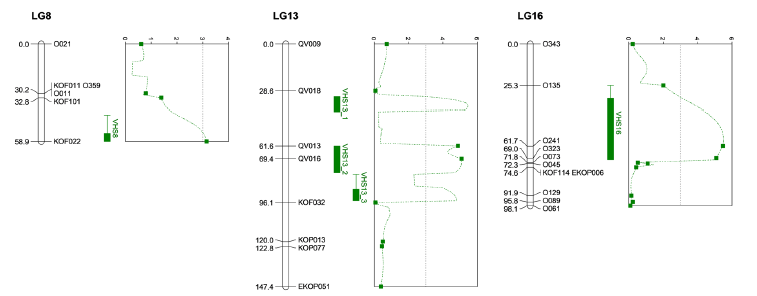
Linkage group을 활용하여 기존 마커 정보를 기반으로 pseudo marker 형성 및 넙치의 VHSV 저항성에 관여하는 QTL을 도출하였다 (Fig. 4,5). 발견된 5개의 QTL은 1:1:1:1, 1:2:1 및 1:1의 비율로 분리되었다. LG8에서 발견된 VHS8은 LOD 값 3.15, 설명력은 전체 표현형 분산의 7.68%를 차지했다. LG13에서는 VHS13_1, VHS13_2 및 VHS13_3이 QTL로 검출되었다. LOD 값은 각각 5.47, 5.09, 4.82 이었으며 전체 표현형 분산의 2.22%, 1.80%, 1.03%를 차지했다. 마지막으로 LG16에서 VHS16이 발견되었고, LOD 값은 5.51, 설명력은 전체 표현형 분산의 8.97%로 확인되었다. 어미에 대한 상가적 효과는 VHS16에서 가장 높았고(0.18), 아비에 대한 상가적 효과는 VHS13_3에서 가장 높았다(0.19). VHS13_1은 부모에 대한 상가적 효과는 미미하지만 가장 높은 우성효과를 보였다. 각 마커의 VHSV에 대한 자세한 QTL 정보는 Table 3에서 확인할 수 있다. Linkage group을 형성하는데 활용된 마커 중 전체 표현형의 8.97%를 설명하고 가장 높은 LOD 값 (5.51)을 나타낸 LG16의 VHS16에 가장 근접한 O135, O241은 앞으로의 육종에 있어 특히 중요하다고 사료된다. 이외에도 Table 3에 제시된 pseudo QTL marker에 인접한 KOF022, QV018, QV013, QV016, KOF032마커는 향후 육종 선발에 있어서 종묘 생산의 기준이 되기에 적합할 것으로 사료된다.
해당 논문에 활용된 VHSV 저항성을 갖는 개체의 선발은 생존/사망을 이산형 표현형 데이터로 활용하여 진행되었다. 많은 논문에서 확인되었듯, 표현형에 영향을 미치는 요인은 유전적 요인 뿐만 아니라 다양한 환경적 요인이 작용한다 (Gienapp et al., 2017). 따라서 추가 분석을 통해 앞서 제시한 QTL 마커들을 기반으로 다양한 환경적 요인을 추가로 보정해준 후 각 마커들이 갖는 effect값을 재 조정해주고 육종에 활용할 필요가 있는 것으로 사료된다.
Conclusion
QTL 분석 결과, 5개의 pseudo marker가 추정된 연관지도의 8번, 13번, 16번 linkage map에서 QTL로 확인되었다. 확인된 pseudo marker VHS8, VHS13_1, VHS13_2, VHS13_3, VHS16은 차례대로 각각 7.68%, 2.22%, 1.8%, 1.03%, 8.97%만큼의 설명력을 갖는 것으로 확인되었다. 이와 가장 근접하게 위치한 7개의 마커(KOF022, QV018, QV013, QV016, KOF032, O135)를 넙치의 VHSV 저항성 유전자 좌위로 제시될 수 있다. 다만 해당 논문에 활용된 표현형 데이터는 생존/사망의 이산형 데이터이기에 표현형 분산을 보다 정확하게 예측하는 QTL 마커를 규명하기 위해선 다양한 환경 요인을 고려하면 더 높은 신뢰를 갖는 표현형 분산을 도출해낼 수 있을 것으로 사료된다.