서론
인공지능(Artificial intelligence, AI)은 기계학습(Machine learning)과 딥러닝(Deep learning) 기술의 발달로 인해 다양한 분야로의 접목이 가능해지게 되었다(Fig. 1). 먼저 인공지능은 인간의 지능이 할 수 있는 사고, 학습, 개발 등을 컴퓨터가 할 수 있도록 연구하는 정보기술 분야로 이러한 인공지능을 구현하는 구체적인 접근방식으로 기계학습이 등장했다(Winston and Patrick Henry, 1992). 기계학습이란 기본적으로 알고리즘을 이용한 데이터 분석, 분석을 이용한 학습, 학습으로 얻어진 정보를 기반으로 판단이나 예측을 하는 것이다(Jordan et al., 2015). 즉, 대량의 데이터와 알고리즘을 통해 컴퓨터를 “학습” 시켜 작업 수행 방식을 익히는 것을 말하며 기초 데이터를 주지 않아도 스스로 학습을 통해 결과값을 만드는 원리는 알아가는 것을 말한다. 이후 완전한 기계학습 실현 기술로써 심층학습인 딥러닝이 등장하게 되었다(LeCun et al., 2015). 딥러닝은 컴퓨터가 특정 업무를 수행할 때 정형화된 데이터를 받지 않고 스스로 필요한 데이터를 수집하고 분석하여 빠르게 처리할 수 있도록 한다. 즉, 사람이 개입하던 과정이 필요 없어진 것이다. 기계학습은 많은 데이터를 기계학습화 하여 결과를 나타내는 것이고 이러한 결과에서 사람 개입 없이 기계 스스로 데이터를 분석하고 분류하는 것이 딥러닝이다(Deng et al., 2014). 딥러닝은 기계학습의 한 분야이지만 한 단계 발전한 형태라고 볼 수 있다.
기계학습방법으로는 지도학습(supervised learning), 비지도학습(unsupervised learning), 준지도학습(semi-supervised learning)이라는 다양한 접근방식이 존재하며 각 학습마다 적용하는 전략이 다르다(Sathya et al., 2013). 더 나아가 딥러닝은 데이터의 특성에 따라 합성곱신경망(Convolution Neural Network, CNN), 순환신경망(Recurrent Neural Network, RNN) 등 다양한 접근 방법이 존재한다(Ciaburro et al., 2017). 데이터의 종류와 상황에 따라 접근방식이 다르기 때문에 어떤 전략과 방법이 최적의 결과물을 출력하는지 고려하는 것이 중요하다.
표현형(Phenotype)에 대한 기계학습의 적용은 동물의 행동학적인 관찰에서 이루어지고 있다(Arac and Ahmet et al., 2019). 다층구조를 가지고 심층학습이 가능한 딥러닝의 등장은 다양한 분야로의 접목을 가능하게 하였다. 현재 국내 농·축산업 중에서 가장 규모가 큰 산업인 양돈산업에서 도체 등급판정이 많이 이루어지고 있다(Kyriazakis et al., 2008). 도체 등급판정은 몸통피부근과 넓은등근의 발달정도 즉, 비율로 결정이 된다(Lee et al., 2018). 사진 이미지의 경우 단면적을 수많은 픽셀로 나누어 각 픽셀에 가중치와 알고리즘을 이용해 기계학습 적용이 이루어지고 있다(Arganda-Carreras et al., 2017). 마찬가지로 도체 등급판정 시 도체의 단면적을 여러 픽셀로 나누어 전체 면적 중 몸통피부근과 넓은등근의 비율을 딥러닝을 통해 학습시키면 사람이 육안으로 등급판정을 하는 것보다 더 정확하고 빠르게 등급판정이 가능할 것으로 보인다. 표현형뿐만 아니라 유전체 분석을 통해 관련 형질 유전자 마커를 찾아 기계학습에 적용시킬 수 있다. 차세대염기서열분석(Next-generation sequencing, NGS)기술의 발달로 인한 전장유전체연관분석법(Genome-Wide Association Study, GWAS)는 관련형질에 대한 마커를 발굴할 수 있는 좋은 기법 중 하나로, 원하는 형질을 발현시키는 유전자를 찾을 수 있다(Wang et al., 2016). 기계학습에 유전체를 적용시키면 유전능력 예측이 가능하여 형질별 우수 축종에 대한 원하는 품종 개량 연구도 가능하다. 기계는 유전체의 전사시작위치, 전사종결위치, deoxyribo nucleic acid(DNA)서열 인식 등을 스스로 학습하여 가능하게한다.
기계학습 단계 및 방법
1. 기계학습의 단계
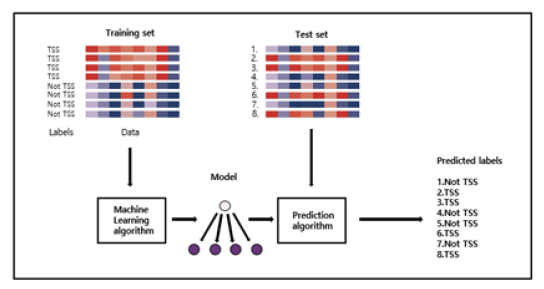
일반적으로 기계학습은 훈련 세트(Training set)와 테스트 세트(Test set)를 통해 ‘디자인-학습-테스트’라는 세 단계 과정을 거친다(Jones, David T 2019). 기계학습을 통해 전체유전자서열(Whole Genome Sequence)에서 전사시작위치(Transcription Start Site, TSS)를 식별하는 프로그램을 적용시킬 수 있다(Libbrecht, Maxwell W., and William Stafford Noble, 2015). 첫 단계는 알고리즘의 개발, 디자인이다(Fig. 2). 이후, 두 번째 단계를 진행하기 위해 알고리즘에 대규모의 TSS 서열과 TSS가 아닌 서열이 제공이 되고, TSS 여부를 나타내는 label이라는 주석을 달아준다. 알고리즘은 label이 지정된 서열을 식별, 처리하기 위해 splice site, promoter, enhancer, positioned nucleosome에 대해 훈련되고 모델을 저장한다. 마지막 단계에서, label이 지정되지 않은 서열들(Test set)이 알고리즘에 제공되고 훈련된 모델을 사용하여 제공된 서열에 대해 TSS 서열인지 혹은 TSS 서열이 아닌지 예측한다. 학습이 성공적이면 대부분의 예측된 label은 정확할 것이다. 이것은 지도학습이라는 기계학습의 하위 유형의 예시이다.
2. 기계학습의 방법
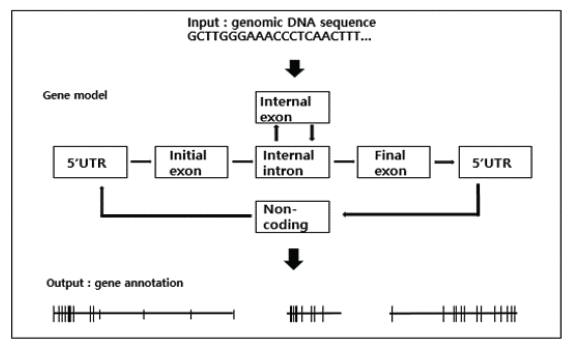
기계학습 방법은 크게 지도학습, 비지도학습이라는 두 가지 주요 범주로 나뉜다(Sathya et al., 2013). 지도학습은 label이 지정된 예제에 대해 학습한 후 label이 없는 예제에 대한 예측을 하는데 사용된다(Bzdok, Danilo, Martin Krzywinski, and Naomi Altman, 2018). 반면에 비지도학습은 label을 사용하지 않고 스스로 학습하여 예측하는데 사용된다(Ghahramani, Zoubin, 2003). 염색체의 DNA서열을 입력 데이터(Input data)로 사용하여, 염색체상에 존재하는 단백질 암호화 유전자의 위치와 인트론-엑손 구조를 예측하는 ‘gene-finding’ 알고리즘을 통해 두 접근방식의 차이를 설명할 수 있다(Schweikert, Gabriele, et al., 2009). 예측모델 설계의 가장 간단한 방법은 이미 알려진 유전체(Genome)를 사용하여 만드는 것이다(Fig. 3). 지도학습의 경우 입력 데이터는 유전자의 시작(전사시작)과 끝(전사종결)의 위치를 지정하는 label된 DNA서열을 요구한다. 기계는 두 위치 사이의 splice 위치뿐만 아니라 splice 부위 근처에서 전형적으로 발생하는 DNA 서열 패턴과 같은 유전자의 일반적인 특성을 학습한다. 학습된 모델은 학습된 특성을 사용하여 training set의 유전자와 유사한 추가적인 유전자를 식별할 수 있다. Label이 지정된 훈련 데이터를 사용할 수 없는 경우 비지도학습이 필요하다. 데이터에 사전 정의된 label을 강요하기보다는 데이터를 가장 잘 설명하는 label 유형을 찾는데 관심이 있다면 지도학습보다는 비지도학습을 사용해야 한다. 비지도학습의 알고리즘은 label이 없는 데이터와 원하는 수의 다른 label만 사용하여 입력 데이터로 지정한다. 그 후, genome을 segment로 분할하고 비슷한 데이터를 가진 segment에 동일한 label 지정을 목적으로 각 segment에 label을 지정한다. 비지도학습 접근방식은 각 label에 segment를 수동으로 지정해 주어야 하는 추가 단계가 필요하지만 label이 있는 데이터를 사용할 수 없는 경우 훈련을 할 수 있는 이점을 제공하고 잠재적으로 새로운 유형의 genome 요소를 식별할 수 있다.
준지도학습은 지도학습과 비지도학습의 중간 학습 방법이다(Zhu, Xiaojin Jerry, 2005). 지도학습에서 알고리즘은 label과 관련 있는 데이터를 입력으로 수신한다. 반면에 비지도학습에서 알고리즘은 데이터를 수신하지만 label은 받지 않는다. 준지도학습은 이 두 가지 접근방식의 혼합이다. 알고리즘은 데이터를 수신하지만 일부에만 연관된 label이 있다. 실제로 ‘gene-finding’ 시스템은 준지도학습 접근방식을 사용하여 훈련되기도 한다. 여기서 입력 데이터는 주석이 달린 유전자와 label 되지 않은 전체유전자서열이다. 학습절차는 훈련 데이터의 일부 label에 기초하여 초기 ‘gene-finding’ 모델을 제작함으로써 시작된다. 다음으로 모델은 genome을 스캔하기 위해 사용되고 잠정적인 label은 genome을 통해 지정된다. 이런 잠정적인 label은 학습된 모델을 개선하기 위해 사용될 수 있고 절차는 새로운 유전자가 발견되지 않을 때까지 반복된다. 모델은 높은 신뢰도를 가진 것으로 확인된 일부 유전자가 아닌 genome 안의 모든 유전자에서 학습할 수 있기 때문에 준지도학습은 지도학습보다 훨씬 더 잘 작동할 수 있다.
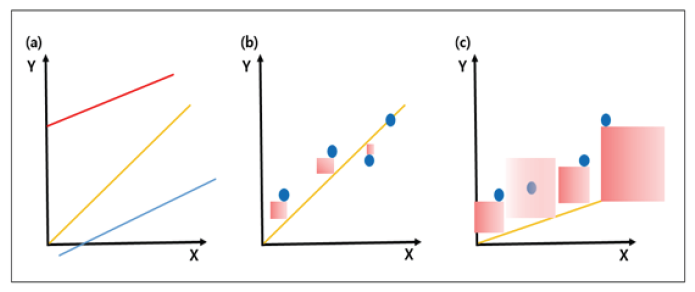
지도학습의 유형은 학습결과를 바탕으로, 미래의 무엇을 예측하느냐에 따라 회귀(Regression)문제, 분류(Classification)문제로 구분할 수 있다(Criminisi, Antonio, Jamie Shotton, and Ender Konukoglu, 2012). 회귀문제는 훈련 데이터를 이용하여 연속적인 값을 예측하는 것을 말하며 대표적인 예로 공부시간과 시험성적간의 관계를 들 수 있다. 회귀모델 중 선형회귀는 직선, 즉 일차함수의 개념인 Y= ax + b 직선을 임의로 그려놓고, 그 직선을 바탕으로 예측하는 것이다(Fig. 4. a). 함수를 수식으로 표현하면 H(x)= Wx + b이다. H(x)는 우리가 가정한 가설(Hypothesis)을 의미하고 W는 가중치(Weight)의 값 그리고 b는 편차(bias)를 의미하며 수치에 따라 선의 모양이 달라진다. 결과적으로 이 회귀분석을 통하여 그래프 상에서 선을 그렸을 때 W값과 미세하게 조절되는 b의 값을 찾아 가설과 실제 데이터의 차가 가장 작은 최소값을 찾는 것이 기계학습에서의 선형회귀의 학습이라고 볼 수 있다. 우리가 예측하기 위해 만든 모델인 Y= ax + b 직선과 실제 데이터를 찍어놓은 점들의 Y값 차이를 error라고 한다. Error가 실제 데이터의 Y값과 예측 직선모델의 Y값의 차이라고 하면 square error는 실제 데이터의 Y값과 예측 직선모델의 Y값의 차이를 제곱해서 넓이로 보는 것이다.
Error를 제곱해서 넓이로 보는 이유는 육안으로 보기 쉽고 수학적으로 볼 때, error가 조금이라도 있다면 값이 증폭되어 큰 값과 작은 값의 비교를 쉽게 할 수 있다. 또한 기계학습의 알고리즘에서 계산이 용이하게 편미분된다. 선형회귀에서 어떤 가설 모델이 더 적합한지 확인하려면 square error의 측면에서 확인해야 한다(Niculescu-Mizil, Alexandru, and Rich Caruana, 2005). Square error를 구하고 평균인 mean square error를 통해 적합한 선형회귀모델을 확인할 수 있다(Fig. 4).
분류문제는 훈련 데이터를 이용하여 주어진 입력 값이 어떤 종류의 값인지 구별하는 것을 지칭한다(Kotsiantis, Sotiris B., Ioannis Zaharakis, and P. Pintelas, 2007). 분류문제는 크게 binary classification과 multi-label classification 두 가지로 나뉜다. Binary classification은 ‘합격/불합격’ 또는 암의 경우 ‘악성종양/아닌지’ 등 ‘맞다/아니다’로 구분되는 문제를 말한다. 분류문제가 모두 ‘맞다/아니다’로 구분되는 것은 아니다. 예를 들어, 공부시간에 따른 ‘합격/불합격’을 예측한다고 하면 이는 binary classification으로 볼 수 있다. 반면에, 공부시간에 따른 학점을 ‘A/B/C/D/F’로 예측하는 분류문제를 multi-label classification이라고 한다. 회귀문제는 출력 값이 연속성을 가지고 있어 확률을 예측하는 유형이 아니지만 분류문제는 확률을 통해 예측을 하는 유형이다.
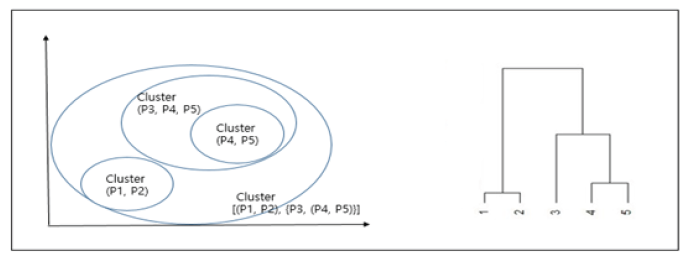
비지도학습은 훈련 데이터에 정답은 없고 입력 데이터만 있기 때문에, 입력에 대한 정답을 찾는 것이 아닌 입력 데이터의 패턴, 특성 등을 학습을 통해 발견하는 학습이다(Chen, Chien-Chang, et al., 2018). 예측이 아닌 데이터에서 의미를 파악하고 기준을 만드는데 사용하는 비지도학습에서 가장 대표적인 것이 군집화(Clustering)이다(Fig. 5). 즉, 군집화는 아무런 정보가 없는 상태에서 비슷한 성질을 가지고 있는지 파악하여 비슷한 데이터들끼리 군집으로 묶어 주는 분석방법이다(Greene, Derek, Pádraig Cunningham, and Rudolf Mayer, 2008). 군집분석 알고리즘에는 계층적 군집분석과 비계층적 군집분석이 존재하며 용도에 따라 다르게 활용된다. 먼저 계층적 군집분석은 한 군집이 다른 군집을 포함할 수 있는 구조로 군집을 만드는 기법이다. 상위항목에서 아래로 세분화되며 계층화되는 형태로 데이터를 군집화한다. 계층적 군집분석은 가지형태로 파생되는 형태로 결과가 얻어지는데 이를 계통도(Dendrogram)라고 한다. 반면 비계층적 군집분석은 군집끼리 포함관계를 이루지 않고 서로 독립적인 한 군집으로 만드는 기법이다. 비계층적 군집분석은 거리를 기반으로 군집화하는 방법(K-means)과 밀도를 기반으로 군집화하는 방법(DB-SCAN)이 있으며 데이터가 분포한 특징에 따라 원하는 방법을 선택해서 사용할 수 있다(Chen, Shouhong, et al., 2019). K-means 군집화는 n개의 중심점을 찍은 후에, 이 중심점에서 각 점간의 거리의 합이 가장 최소가 되는 중심점 n의 위치를 찾고, 이 중심점에서 가까운 점들을 중심점을 기준으로 묶는 클러스터링 방법이다. 이 중심점은 결국 각 군집의 데이터의 평균값을 위치로 가지게 되는데 이런 이유로 평균(means)값 방법이라고 한다. DB-SCAN은 점이 세밀하게 몰려 있어서 밀도가 높은 부분을 군집화하는 방법이다. 즉, 한 점을 기준으로 반경 X내에 점이 n개 이상 있으면 하나의 군집으로 인식하는 방법이다. K-means 군집화 방법은 중심점을 기준으로 가까운 데이터들을 군집에 포함시키기 때문에 원의 형태로 군집화가 된다. 반면에 DB-SCAN은 서로 이웃한 데이터들을 같은 군집에 포함시키기 때문에 불특정한 모양의 군집화가 된다. DB-SCAN은 K-means와 다르게 군집의 수를 지정할 필요가 없으며, 알고리즘이 자동으로 군집의 수를 찾는다. 더 나아가 원모양의 군집뿐만 아니라, 불특정한 모양의 군집도 찾을 수 있다. 하지만 데이터가 입력되는 순서와 알고리즘이 이용하는 거리 측정 방법에 따라 군집의 결과가 변한다.
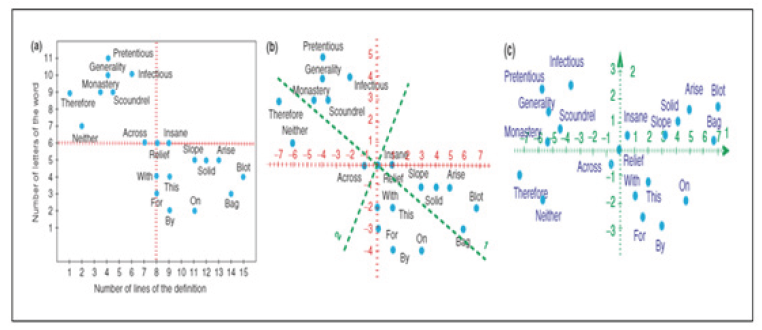
비지도학습에서 사용되는 주성분분석(Principle Component Analysis, PCA)은 고차원의 데이터를 저차원의 데이터로 축소시키는 차원축소방법 중 하나이다(Valpola, Harri, 2015). 주성분 분석은 특성들이 통계적으로 상관관계가 없도록 데이터 세트를 회전시키는 기술로 서로 연관 가능성이 있는 고차원의 데이터를 연관성이 없는 저차원으로 변환시키기 위해 직교변환을 사용한다(Fig. 6). 직교변환을 위해 먼저 알고리즘은 데이터에서 분산이 가장 큰 방향을 찾는다. 분산이 큰 방향은 특성들의 상관관계가 가장 크고 많은 정보를 담고 있는 방향이다. 가장 큰 분산을 갖는 방향을 찾고, 해당 방향과 직각이면서 두 번째로 분산이 큰 방향을 찾는다. 2차원 그래프에서는 방향이 한정적이기 때문에 직각 방향이 하나이지만, 고차원 그래프에서는 수많은 직각 방향이 존재한다. 일반적으로 특성의 개수만큼 주성분이 존재하고 선형모델과 유사하게 데이터에서 의미 있는 축을 찾는 과정이다. 데이터의 범위를 재조정하고 데이터 평균을 0으로 맞춰줌으로써 주성분 분석은 고차원의 데이터 중에서 중요한 차원을 골라준다. 기계학습을 할 때 훈련 데이터의 특성(feature)이 많은 경우가 있지만 모든 특성이 결과에 영향을 끼치는 것은 아니다. 주성분 분석을 통해 가장 중요한 특성 몇 개만을 선택할 수 있다. 10개의 특성이 존재한다고 할 때, 10개의 특성과 1개의 label을 mapping시키는 그래프를 그리려면 10차원의 그래프가 필요하다. 이때 중요한 2개의 특성만 선택해서 그래프를 그리면 2차원의 그래프가 될 것이다. 즉, 10개 중 2개의 특성만 뽑아서 10차원을 2차원으로 차원을 축소 시킬 수 있다. 주성분 분석의 단점은 그래프의 두 축을 해석하기가 어렵다는 것이다. 원본 데이터에 있는 어떤 방향에 대응하는 여러 특성이 같이 조합된 형태이고 거의 대부분의 특성이 섞여 있기 때문에 축이 가지는 의미를 설명하기가 어렵다. 그럼에도 불구하고 차원축소를 하는 가장 큰 이유는 시각화이다. 3차원이 넘어간 시각화는 우리 눈으로 볼 수 없기 때문에 시각화를 통해 데이터 패턴을 쉽게 인지할 수 있다. 불필요한 특성들을 제거해 출력된 데이터는 다른 학습 시에 수렴 속도와 성능을 향상 시킬 수 있다.
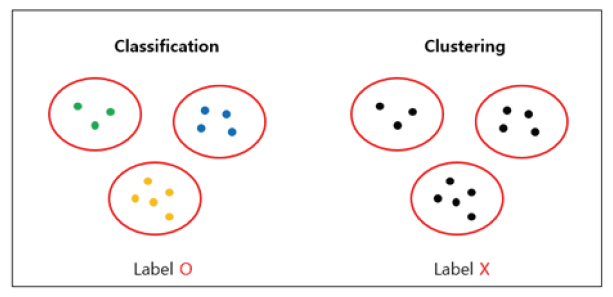
데이터를 비슷한 집단으로 묶는 방법이라는 점에서 분류와 군집화는 비슷한 특징을 갖지만, 분류는 label이 있는 데이터를 나누는 방법으로 지도학습의 일종이며 군집화는 label이 없는 데이터를 나누는 방법으로 비지도학습의 일종이다(Fig. 7).
3. 딥러닝 접근 방식
기계학습의 종류로 인공신경망 알고리즘인 딥러닝이 등장했다. 심층학습이라고 불리는 딥러닝은 인간의 신경망처럼 무수히 많은 인공신경망(Artificial neural network, ANN)을 통해 기계가 다층구조를 통해 학습하도록 만든 기술이다(Schmidhuber, Jürgen, 2015). 딥러닝 기술은 음성인식, 신호처리, 이미지 분석 등 다양한 분야에 적용되어 최첨단의 결과들을 보여주고 있다. 크게 CNN과 RNN이라는 두 가지 딥러닝 방식이 사용되고 있다.
먼저 CNN은 필터링 기법을 인공신경망에 적용함으로써 이미지 데이터를 학습하고 인식하는데 특화된 알고리즘이다(Albawi, Saad, Tareq Abed Mohammed, and Saad Al-Zawi, 2017). CNN은 neural network 앞에 여러 계층의 convolutional layer를 붙이는 방식으로, convolutional layer를 통해 입력 받은 이미지에 대한 특징(feature)을 추출한다. 이렇게 추출된 특징을 기반으로 기존의 neural network를 이용하여 분류해낸다. Convolutional layer는 특징을 추출하는 기능을 하는 필터와 이 필터의 값을 비선형 값으로 바꾸어주는 activation 함수(sigmoid)로 이루어진다. CNN은 데이터의 크기도 줄이면서 이미지의 특징도 추출할 수 있다는 장점을 가지고 있다.
RNN은 주가, 실시간 필기, 음성같이 시간에 따라 변화하면서 입력되는 데이터에 특화된 알고리즘으로 시간 차이가 근접할수록 서로 비슷한 특징을 갖는다(Yin, Chuanlong, et al., 2017). 시간에 따라 변화하는 데이터의 경우 바로 전 상황과 밀접한 관계가 있으므로 신경망은 지금 들어온 데이터와 전에 들어온 데이터가 연관이 있는지 인지해야 한다. 즉, 이전에 입력받은 데이터를 잊어버리지 않도록 새로운 데이터를 입력받았을 때 이전 데이터를 다시 입력시키는 방법인 순환 신경망이 개발되었다. RNN을 통해 기계가 음성을 합성하여 사람처럼 말하는 인공지능이 개발되었다.
4. 유전체 분석 및 적용
1) 유전체 분석 방법
유전체 데이터의 생산은 조직에서 단일 세포를 분리함으로써 이루어진다. 하나의 세포에서 얻을 수 있는 DNA의 양은 매우 적기 때문에, 증폭 과정을 통해 서열분석(sequencing)이 가능할 정도로 만들어준다. 이를 위해 각 세포에서 DNA를 추출하여 중합효소연쇄반응(Polymerase Chain Reaction, PCR)을 통해 증폭된 DNA를 얻고 sequencing 라이브러리를 제작하는 과정을 진행한다. 유전체 데이터 생산을 위한 분석기법은 다음과 같다(Edwards, K., C. Johnstone, and C1 Thompson, 1991).
Sanger sequencing은 DNA sequencing의 한 방법으로, 하나의 DNA 주형가닥에 프라이머(Primer)가 결합한 후 deoxynucleotide triphosphate (dNTP)에 의한 정상적인 합성이 일어나다가 dideoxynucleotide triphosphate (ddNTP)에 의한 합성 종결을 유도하는 분석기법이다(Men, Artem E., et al., 2008). ddNTP는 3번탄소에 OH가 아닌 H가 위치하여 Phosphodiester bond를 형성하지 못해 합성을 종료시키고 형광물질이 표기되어 서열을 감지할 수 있다. 짧은 염기서열을 분석할 경우 높은 정확도를 가지고 있지만 시간과 비용의 한계를 갖는다.
차세대 염기서열 분석법(Next-Generation Sequencing, NGS)은 대량으로 한꺼번에 유전체의 염기서열 정보를 얻는 방법으로, 방대한 유전체 정보를 빠르게 해독하는 분석기법이다(Behjati, Sam, and Patrick S. Tarpey, 2013). Sanger sequencing 등 기존의 염기서열분석 기법을 대체하는 기술이며 전사체와 후성유전체에 적용하여 새로운 영역을 발견할 수 있게 해주는 기술로 활용된다. 개별개체의 염기서열 정보를 중심으로 진행되며 진화, 환경적 요인의 차이에 의한 유전정보 분석을 가능하게 한다. 한 번에 대량의 염기서열을 생산해냄으로써, 시간과 비용을 획기적으로 줄일 수 있는 것이 핵심기술이다. Sanger sequencing에서 탐지할 수 없는 다양한 유전체 변이를 NGS는 정확하게 탐지해 낼 수 있고 탐지할 수 있는 변이의 민감도도 더 좋다.
2) 유전체 분석 적용
NGS 기술의 발달로 인한 GWAS는 관련형질에 대한 마커를 발굴할 수 있는 좋은 기법 중 하나로, 원하는 형질을 발현시키는 유전자에 대한 정보를 제공한다(Black, Michael, Wenzhi Wang, and Wei Wang, 2015). DNA가 가지고 있는 유전정보에 의하여 세포, 생물, 개체 등에서 발현된 유전적 특징을 유전형(Genotype)이라 하며, 생물체의 발생과 성장 과정에서 자연환경과 상호작용하여 나타나는 특징을 표현형(Phenotype)이라 한다(Chanock, Stephen J., et al., 2007). GWAS 분석은 크게 유전형과 표현형 2가지 요인에 의해서 좌우되고, 유전형과 표현형의 연관 관계를 찾는 것이 가장 큰 분석의 목적이라 할 수 있다. 다수 개체의 유전형 정보와 표현형 정보를 알고 있다면, 해당 표현형을 나타나게 하는 유전 좌위가 어느 곳인지 통계적 방법을 통해 확인할 수 있다. 이를 통해 유전체가 어떻게 기능하는지 이해할 수 있고, 다양한 응용을 가능하게 한다. 유전형 데이터 생성을 위해 Simple Sequence Repeat (SSR), Restriction Fragment Length Polymorphism(RFLP), 기본적인 분자 마커 등 다형성을 보이는 유전자형 마커가 사용되었지만, NGS 기술의 발달로 단일염기다형성(Single Nucleotide Polymorphism, SNP)이 많이 활용되고 있다(Nielsen, Rasmus, et al., 2011). SNP는 돌연변이에 의해 DNA 염기서열 중 하나의 서열이 다른 서열로 치환되고 이후 집단 내에서 일정한 빈도로 존재하는 유전변이이다. GWAS는 많은 SNP를 microarray 분석을 통해 각 개인의 SNP 유전자형들을 결정하고, 결정된 유전자형들에서 질병이나 특정 표현형과 동시에 존재하는 확률을 계산하여 가장 유의성이 높은 유전자형-표현형의 관련성을 나타내는 SNP를 발굴하는 분석이다(Yang, Jian, et al., 2012). GWAS를 통해서 발굴되는 SNP는 그 자체로써 표현형에 영향을 미치는 원인유전변이인 경우도 있고, 다른 유전변이들에 대한 마커로서의 역할을 할 수 있다. 마커로서 역할을 하는 유전변이의 경우는 실제적인 원인유전변이와 높은 연관불평형관계(Linkage Disequilibrium, LD)상태에 있는 것으로 해석된다(Corradin, Olivia, et al., 2014). 연관불평형이란 두 개의 SNP들의 유전체상에서의 위치를 고려하면, 생식세포의 분열 시에 둘 사이에 교차(Cross Over)가 일어나서 각각 유전될 수 있지만 같이 유전되는 확률이 높은 경우를 말한다. 즉, GWAS를 통해 관련 형질에 대한 마커를 발굴할 수 있고 원하는 형질을 발현시키는 유전자에 대한 정보를 얻을 수 있다.
최근 SNP 마커를 이용하여 가축 품종의 유전능력을 추정하는 연구가 진행되고 있다. 유전체선발(Genomic Selection, GS)은 기존 통계육종 방법에 비해 육종가 추정의 정확도를 높일 수 있으며, 세대 간격을 줄이고, 혈통 오류를 감소시키며, 유전력이 낮은 형질이나 측정하기 어려운 형질에 대해서도 육종가를 추정할 수 있는 장점이 있다(Goddard, M. E., and B. J. Hayes., 2007). 가축에서는 현재 딥러닝 수준에서 행동학적인 관찰이나, 질병 진단수준에서만 시도되고 있다. 하지만 동물분자육종분야에서, 복잡한 경제 형질에 대해 초기의 기계학습 알고리즘에 베이지언 추론 및 회귀 분석법을 접목하여 유전체유전능력예측 연구가 가능할 것으로 보인다. 기계학습을 이용하여 유전체 육종가를 추정하기 위한 예측모델을 구현할 때 유전체 선발 시 소요되는 데이터를 training data set (유전형, 표현형 모두 보유)과 testing data set (유전자형만 보유)으로 구분 짓고 이를 지도학습과 비지도학습의 일종으로 간주하여 기계학습에 적용함으로써 육종가를 추정할 수 있다.
3. 결론
1. 기계학습 접근의 고려사항
새로운 기계학습에 직면했을 때 가장 먼저 고려해야 할 사항은 지도학습, 비지도학습, 준지도학습 중에 어떤 학습을 적용할 것인지 여부이다(Sathya, Ramadass, and Annamma Abraham., 2013). 몇몇의 경우 학습 방법의 선택은 제한되어있다. 사용 가능한 label이 없으면 스스로 학습하여 예측하는 비지도학습만 가능하다. 그러나 label을 사용할 수 있는 경우 지도학습이 최선의 선택은 아니다. 예를 들어, 생물 기능의 단위인 유전자에 해당하는 위치를 찾기 위해 유전체 서열분석에 지도학습을 적용할 수 있다. 하나의 label 된 데이터 세트를 가져와서 무작위로 training set과 test set으로 나누면 이 가정이 존중된다. 하지만 인간의 유전자 데이터를 사용하여 훈련된 ‘gene-finder’ 모델은 쥐 genome에서 유전자를 찾는데 잘 수행되지는 않을 것이다. 일반적으로 지도학습은 training set과 test set이 유사한 통계적 특성을 보일 것으로 예상되는 경우에만 사용해야 한다. 지도학습이 가능하고 label이 지정되지 않은 추가적인 데이터를 쉽게 얻을 수 있는 경우 지도학습 또는 준지도학습 접근방식을 고려할 수 있다. 준지도학습은 데이터에 대한 특정 가설을 설정해야 하며 실제로 이러한 가정을 평가하는 것은 종종 어려울 수 있다. 즉, 준지도학습은 적은 양의 label이 지정된 데이터와 많은 양의 label이 지정되지 않은 데이터가 있는 경우 사용하면 좋다. 전사체 분석을 통한 차등발현유전자(Differentially Expressed Genes, DEG)에 대한 발현양상을 heapmap으로 시각화하는 경우, 비지도학습이 적용된다. Heapmap에서는 주로 hierarchical clustering을 사용하는데, 이 방법이 비지도학습의 예이다. 발현값만을 기준으로 계층적 클러스터링을 진행하며, co-expression을 보이는 유전자 군집 확인 및 outlier 샘플을 확인할 수 있다.
심층학습인 딥러닝도 마찬가지로 데이터의 특성에 따라 CNN, RNN 접근방식 중 무엇을 선택할지 고려해야 한다(Javaid, Ahmad, et al., 2016). CNN은 인간의 시신경 구조를 모방한 기술로 이미지 프로세싱에서 시작해 글자, 이미지, 사진인식에 이르기까지 인식에 필요한 특징을 자동으로 학습하는데 특화된 알고리즘이다. 반면에 RNN은 시간에 따라 변화하면서 입력되는 데이터를 학습하는데 특화된 알고리즘이다. 신경망은 지금 들어온 데이터와 전에 들어온 데이터가 연관이 있는지 인지하고 이전에 입력받은 데이터를 잊어버리지 않도록 새로운 데이터를 입력받았을 때 이전 데이터를 다시 입력시킨다. RNN의 경우 먼 과거의 상태는 현재의 학습에 아무런 영향을 미치지 못하게 되는 문제점이 발생한다. 즉, 데이터의 특성뿐만 아니라 상태도 고려하여 학습에 적용시켜야 한다.
2. 앞으로의 연구방향
현재 인공지능은 기계학습과 딥러닝 기술의 발달로 인해 다양한 분야로의 접목이 가능해지게 되었다. 심층학습이라고 불리는 딥러닝은 인간의 신경망처럼 무수히 많은 인공신경망을 통해 기계가 다층구조를 통해 학습하도록 만든 기술이다. 딥러닝 기술은 음성인식, 신호처리, 이미지 분석 등 다양한 분야에 적용되어 최첨단의 결과들을 보여주고 있다. 여러 분야 중 가축 산업에서 표현형에 대한 기계학습의 적용은 동물의 행동학적 관찰을 통해 이루어지고 있다. 경제적, 산업적 측면에서 기계학습의 적용은 동물이 보여주는 이상행동, 정상행동을 구분하고 스트레스를 받는다면 조치를 통해 품질이 좋은 종을 생산할 수 있다. 행동 분석은 딥러닝 기술을 통한 이미지 분석의 한 예로 볼 수 있다. 이미지 분석은 수많은 데이터를 알고리즘을 통해 기계에 학습, 적용시킨 분석방법이다.
가축 산업 중 육류 소비량 관점에서 기계학습 기술의 발달은 돼지고기 선호 부위 중 가장 많은 비율을 차지하는 삼겹살 도체 등급판정 평가에서 효과를 보일 것으로 보인다. 선행연구를 통해 삼겹살의 근육 부분 중 몸통피부근과 넓은등근이 잘 발달되면 삼겹살 전 부위에서 지방비율이 높지 않고 탄력도가 높게 형성되어 품질이 우수하다는 것을 알 수 있다. 즉, 사전에 연구된 몸통피부근과 넓은등근의 발달 정도로 삼겹살의 품질평가가 가능하다. 이미지 분석처럼 수많은 삼겹살 단면 사진 데이터를 여러 픽셀로 나누어 각 픽셀에 가중치를 부여한 알고리즘을 통해 기계학습이 가능할 것으로 보인다. 이를 통해 사람이 육안으로 등급판정을 하는 것보다 더 빠르고 정확하게 등급판정이 가능해진다. 더 나아가 누구나 쉽게 사용할 수 있는 앱을 개발하여 시·공간의 제약을 받지 않고 삼겹살의 품질평가가 가능하다. 기계학습의 유전체 적용은 삼겹살 단면이라는 표현형뿐만 아니라 표현형으로 발현시키는 유전형의 적용을 가능하게 한다. 전장유전체연관분석법은 유전형과 표현형 2가지 요인에 의해 좌우되고, 해당 표현형을 나타나게 하는 유전 좌위가 어느 곳인지 통계적 방법을 통해 확인할 수 있다. 즉, 결정된 유전형에서 질병이나 특정 표현형에 영향을 미치는 확률을 계산하여 가장 유의성이 높은 유전자형-표현형의 관련성을 나타내는 SNP를 발굴하는 분석이다. 기계학습의 적용은 사전 연구에서 특정 질병에 대해 발굴된 유전자 및 유전 좌위 이외에 추가적인 마커를 발굴 할 수 있고 원하는 형질을 발현시키는 유전자에 대한 정보를 얻을 수 있다. 전반적으로, 유전체에 기계학습의 적용은 가축의 직접적인 도살 없이 품질 예측이 가능하고, 추가적인 마커를 발굴하여 원하는 형질을 가진 개체생산을 가능하게 할 것으로 보인다.
ACKNOWLEDGEMENT
This work was supported by the National Research Foundation of Korea (NRF) grant funded by the Korea government (MSIT) (NRF-2022R1A2C1005830).요약
현재 많은 정보와 데이터가 생산되는 빅데이터 시대를 맞이하였고 이를 지능형 서비스로 활용할 수 있는 기술로 인공지능(Artificial intelligence, AI) 기술이 각광받고 있다. 인공지능은 기계학습(Machine learning)과 딥러닝(Deep learning) 기술의 발달로 인해 신호처리, 음성인식에서 의료, 복지까지 다양한 분야로의 접목이 가능해졌다. 더 나아가 가축 산업에서 동물의 행동학적인 관찰에 대한 기계학습의 적용이 이루어지고 있다. 동물의 행동학적 관찰에 기계학습을 적용시키면 기계 스스로 학습을 통해 정상행동, 이상행동에 대한 예측 및 분석이 가능하다. 즉 표현형(Phenotype)에 대한 예측이 가능하게 되었다. 가축 산업에 기계학습을 동물의 행동학적인 관찰과 같은 표현형에 적용시키는 것 이외에도 유전체 분석을 통해 관련 형질 유전자 마커를 찾아 기계학습에 적용시킬 수 있다. 이러한 유전체 분석을 통한 기계학습의 적용은 유전능력 예측뿐만 아니라 형질별 우수 축종에 대한 원하는 품종 개량 연구도 가능할 것으로 보인다. 전장유전체연관분석법(Genome-Wide Association Study, GWAS)은 관련 형질에 대한 마커를 발굴할 수 있는 좋은 분석법 중 하나이다. 기계학습의 적용은 데이터를 단순히 기계에 학습을 시키는 것이 아닌 다양한 전략과 방법이 사용되고 있다. 어떤 전략과 방법이 최적의 결과물을 얻을 수 있을지 고려하여 적용시키는 것이 중요하다.