Introduction
1980년대부터 우리나라의 한우 개량사업은 한우 개량 사업소에서 씨수소를 선발하여 보급을 통해 본격적인 국가 단위의 개량 사업이 시작되었다. 개량 사업을 통해 우수한 육질을 보유한 육류로 국내의 소비자들에게 인식 되어 왔으며(J. Lee,2006), 한우의 개량 사업은 매년 능력검정을 통해 유전적으로 우수한 씨수소를 30두 가량 선발하고, 인공 수정에 필요한 씨수소의 정액들을 생산과 공급을 통해 축산업 전반에 한우 개량 가속화, 소득 증대에 기여하고 있다(Yeo et al.,2015). 하지만, 전국 단위의 한우의 사육두수는 증가하고 있으며, 한정된 한우 씨수소의 유전자원을 이용할 경우 유전적 다양성 및 유효 집단의 감소 및 근친도가 높아질 가능성이 있다, 따라서 지속적이고 체계적인 관리가 중요하며 유전적 다양성을 유지하는 것은 근교 퇴화로 인한 가축의 경제적 가치 변화를 고려할 때 집단 육종에 매우 중요한 요인으로 작용한다(Zenger et al.,2007). 특히 한우의 경우 한정된 씨수소를 이용한 개량 사업을 중심으로 산업 구조의 기반을 구축하고 있어 유전적 다양성 감소에 대한 문제점이 발생 할 수 있다. 한우 암소 집단의 혈통 구조 및 근교 계수를 분석한(Cho et al.,2014) 연구에서는 유효 집단의 크기가 지속적으로 감소하고 있음을 보고하였으며, 한우 보증씨수소 집단의 유전적 다양성 및 구조적 변화를 분석한(Shin et al.,2018) 연구에서는 최근 유전적 다양성의 감소가 심화 되고 있음을 보고하였다. 현재 국내 한우 산업의 가임 암소는 161만두로 추정되고 있으며, 약 100두가량의 보증 씨수소 정액을 이용하여 인공 수정을 통해 송아지를 생산하고 있다. 최근 한우 인공 수정용 정액의 판매가 유전적 능력이 우수한 정액과 능력이 비교적 낮은 정액과 선호도 차이로 인해 쏠림 현상이 심화되고 있으며, 그 결과 한우의 유전적 다양성이 급감되는 현상을 우려 할 수 있는 상황이 되고 있다. 이러한 유전적 다양성의 감소 현상은 혈통 관리의 오류가 발생하여 근친의 위험도가 중가하게 되므로 주의가 필요한 부분이다.
우리나라 한우 산업에서 개량을 위한 수단으로는 현재 혈통 관리와 씨수소의 유전능력에만 의존하고 있다. 하지만 최근 암소의 혈통을 기반으로 계획 교배하는 트랜드의 확산으로 인해 혈통의 검증과 오류의 수정은 개량을 위해서 반드시 필요한 기술이 되어 지고 있다. 하지만 실무 환경에서는 혈통의 오류는 다양한 이유로 빈번히 발생되어 지고 있다. 이를 보완하기 위한 수단으로 Microsatellite Marker를 이용한 친자 검정을 통해서 보완해 나갈 수 있다.
한우에서 Microsatellite Marker를 이용한 분석 기술은 2008년 축산물 이력제가 시행되면서 소고기의 이력 정보의 진위 여부를 판정하기 위해서 도입이 되었다. 이력 정보의 진위 여부를 판정하기 위해선 11개의 MS 유전자형을 분석하는 한우 genotyping kit을 이용한다. 도축된 소고기가 유통되는 소고기와 동일한 개체인지를 확인하는 동일성 검정을 위한 kit로 비교적 적은 수의 Marker를 이용하지만, 다양한 유전자형을 보유하고 있어 간단한 실험 방법을 통해 정확한 검정을 할 수 있는 장점을 가지고 있다. 한우 genotyping kit는 축산 산업 현장에서 검정사업, 친자확인을 위한 수단으로 널리 이용되어지고 있다. 하지만 이 방법으로 친자 검정을 할 경우, 혈통상의 아비와 일치 또는 불일치 여부를 확인 할 수 있으나, 불일치 판정이 나온 개체의 친부를 추정하는데에는 한계가 있다. 더욱이 누적된 개량 효과로 인해서 유전적 구조가 비슷해지고 다양성이 감소하면서, 한 개체의 정확한 아비를 추정하기 보다는 여러 개체의 아비 후보를 추정하는 경우가 많아지고 있는 실정이다.
혈통 오류의 수정을 보완하고 한우 산업 현장에서 지속적인 개량의 효율성을 확대하기 위해서는 보다 정확한 검증 기술의 도입이 필요한 시점이다.
최근 한우 보증씨수소의 선발 체계에 SNP chip 유전체 정보를 이용한 유전 능력 평가가 이용 되면서 활용도가 늘어나고 있다. SNP chip을 이용한 개량 방법은 높은 정확도의 유전 능력 평가가 가능하고 혈통 DATA의 오류에도 정확한 아비를 추정할 수 있어 개량의 효율을 높일 수 있다.
본 연구는 암소 사육 집단을 대상으로 SNP 정보를 이용한 혈통의 정확도 향상을 위한 친자 확인 분석 사례를 제시하고, 유전체 기반의 유전 능력 예측 기술과 함께 혈통 정확도를 향상 시킬 수 있는 대안을 제시하기 위해 연구하였다.
Materials & Methods
1. 공시 재료 및 DNA 추출
본 연구는 전국한우협회 정읍시지부에 소속된 한우농가를 대상으로 친자검정을 통해 친자여부가 확인된 615두의 MS 유전자형과 모근을 확보하여 공시재료로 이용하였다.
한우 615두의 유전자형 분석을 위한 시료는 확보된 모근에서 Genome Nucleic Acid Purification Kit (MagExtractor, Toyobo CO., LTD. Osaka, Japan)을 이용해 200 ng/ul 이상의 고농도, 고순도의 DNA를 준비하였다.
2. Microsatellite 유전자형을 이용한 아비추정 정확도 검증
개체의 혈통오류 또는 아비의 정보가 없을 경우 아비 추정 정확도를 확인하기 위해 한우 615두의 친자검정에 사용된 총 11종의 MS marker(TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA126, TGLA122, INRA23, ETH3, ETH225, BM1824) 유전자형을 확보하였으며, 아비의 유전자형에는 모든 개체들이 부부정이 나올 수 있는 임의의 유전자형을 편집하여 자료를 준비하였다. 이렇게 구성된 유전자형 정보를 이용해 아비를 추정하기 위해 농촌진흥청 축산과학원에서 배포하는 친자종합 프로그램(ver. 1.0)을 이용하였다.
3. SNP 유전체 생산 및 친자 확인
한우 615두 그리고 최근 생산된 850두의 보증씨수소(KPN)의 SNP 유전체 정보 생산을 위해 Illumina Bovine SNP50K Bead Chip version3(Illumina, SanDiego, CA, USA)을 이용하였다(42,257 SNPs). Minor allele frequency 0.05 이하 (4,584개), Missing Genotype rate 0.1 이상 (119개) 그리고 Hardy-Weinberg equilibrium test 1e-06이하 (167개)를 제외하고 37,464개의 SNP를 이용하여 친자 확인 검사를 수행하였다. 한우와 KPN간의 친자 확률 계산은 다음과 같다.
parentage score 1,
for i in 1:SNP numer, if sire SNPi genotype = 0 and cow SNPi genotype=2, parentage score 1 + = 1,
if sire SNPi genotype = 2 and cow SNPi genotype=0, parentage score 1 + = 1
parentage score 2,
= for i in 1:SNP numer, if sire SNPi genotype = 0, parentage score 2 + = 1,
if sire SNPi genotype = 2, parentage score 2 + = 1
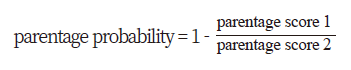
615두의 한우에 대해서 850두의 KPN 중 어떤 개체가 친아비 인지를 찾는 친자 확인 확률 추정을 수행하였다.
Results and discussion
본 연구에서는 기존 MS 유전자형을 이용하는 친자검정 방법과 유전체 정보를 이용하는 친자검정 방법을 이용하여 아비 정확도 비교를 통해 보다 효율적인 친자검정 방법을 제시하고 개량의 효율성 증진에 도움이 되고자 분석을 실시하였다.
본 연구에 이용된 한우의 유전자형 정보와 모근 샘플을 제공한 전국한우협회 정읍시지부는 태어나는 모든 송아지를 대상으로 이력제 등록을 위해 이표를 부착할 때 모근샘플을 채취하여 친자검정을 통해 혈통관리를 실시하고 있다. 이렇게 친자검정이 확인된 615두의 MS 유전자형과 모근샘플을 확보하여 본 연구에 활용하였다.
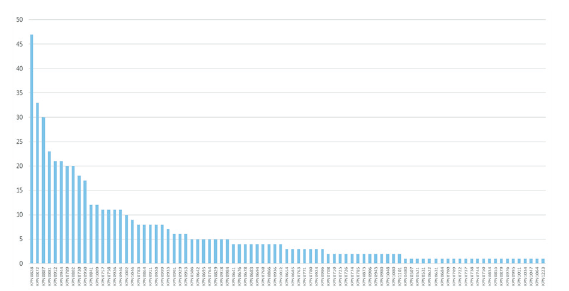
특정 지역에서 한우 인공수정에 이용된 씨수소의 정액 활용 실태를 Figure 1에서 제시하였다. 전체 615두 암소의 혈통을 조사하였으며, 이들 암소집단의 혈통상의 아비들은 총 108두의 씨수소로 확인되었다. 총 108 두의 씨수소 중 특정10두 씨수소가 본 연구에 활용된 암소집단에서 40% 이상을 차지하고 있음이 확인되어, 한우 산업 현장에서 정액의 쏠림현상이 심각한 상황임을 추론할 수 있는 결과가 확인되었다. 특정 씨수소의 혈통구조 점유율이 매우 극단적으로 높은 경향을 보임에 따라 기존의 11개 내외의 MS 표지인자를 이용한 친자확인은 실제 친자확인 분석 검증과정에서 유효한 유전적 다양성이 적어져 친자오류 개체의 정확한 친부를 추정하는데 어려움이 발생할 개연성이 높아진다. 이러한 약점을 보완하기 위해서는 현재 11개의 MS 표지인자의 수를 확대하거나 SNP 패널을 이용하는 방법의 도입이 필요할 것으로 판단된다.
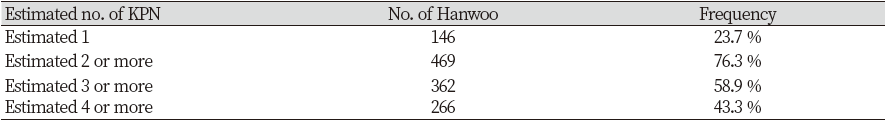
혈통의 오류 또는 아비의 정보가 없을 때 MS 유전자형을 이용한 아비의 추정을 통해 혈통 오류 수정의 가능성을 확인하기 위해서 한우 615 두의 아비를 추정하였다. 11개의 MS 유전자형을 이용해 한우 615 두의 아비를 추정한 결과를 Table 1에 제시하였다. 정확하게 하나의 아비를 추정한 개체는 146두로 전체의 23.7%를 차지하였다. 반면 두 마리 이상의 아비를 추정한 개체는 469 두로 전체의 76.3 %로 확인되었다. 추정된 아비 중 많게는 23마리의 아비가 추정된 개체도 있었다. 이는 서론에서 이야기한 바와 같이, 현장에서 혈통의 오류가 발생하였을 경우 혈통을 수정하기 위한 수단으로 활용하기에 매우 큰 약점을 지니고 있음을 확인하였다.
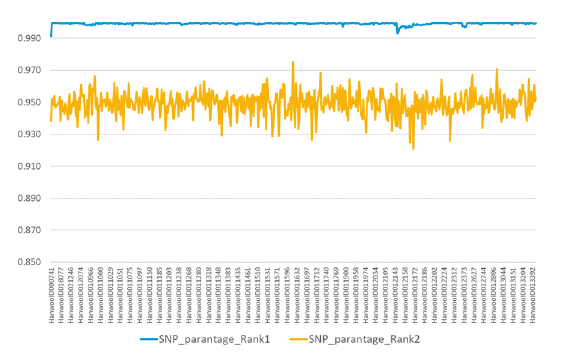
615두의 한우를 대상으로 Illumina 50K chip을 이용하여 42,257개의 SNP 데이터를 생산한 후에 850두 KPN의 Illumina 50K 칩 데이터를 이용하여 아비의 추정을 수행하였다. 각 개체에 대해서 850두의 KPN과의 친자 확률을 계산한 다음에, 아비일 확률이 높은 순으로 KPN을 정렬하여 1순위와 2순위의 확률 정보를 Figure 1에 나타내었다. Figure 2에서 볼 수 있듯이 615두 친자 검정 모두 99%를 넘는 친자 확인 확률을 갖는 아비는 한 마리만 확인되며, 나머지는 99% 이하로 나타나 친부일 가능성이 없음을 확인 할 수 있었다. 따라서 99%를 기준으로 SNP로 친자를 확인할 경우, 정확한 아비의 추정이 가능할 것으로 판단된다.
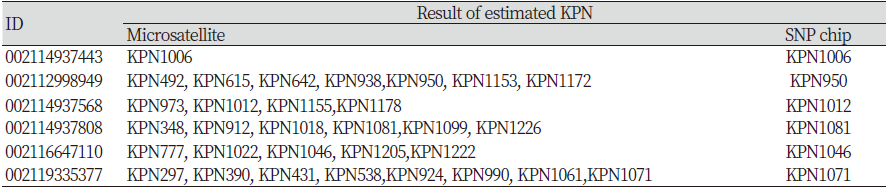
Table 2에서는 MS 유전자형을 이용해 아비를 추정한 결과와 SNP chip을 이용해 아비를 추정한 결과를 비교하여 제시하였다. MS 유전자형을 이용한 친자확인으로 아비를 찾을 경우, 이미 누적된 개량으로 인해 유전적 다양성이 감소하면서 한 개체에 여러 마리의 아비를 추정하는 경우가 빈번하게 발생하는 것이 확인 되었다. 본 연구에서는 615두의 한우에 대해서 MS 유전자형으로 아비를 검색할 경우 469 두의 한우가 여러 마리의 아비 추정이 확인이 되었고, 더 이상의 유전자형 정보가 없기 때문에 정확한 아비를 찾을 수 없음이 확인되었다. 반면, SNP chip을 이용한 아비 추정에서는 정확하게 한 마리의 아비를 특정하여 추정할 수 있음을 확인 하였다.
Conclusions
현재 MS 유전자형을 이용한 친자검정 기술은 상당히 정확한 확률로 혈통상의 아비가 참인지 거짓인지를 확인할 수 있다. 하지만, 혈통상의 아비가 거짓일 경우, 정확한 아비의 추정에는 한계가 있음을 본 연구를 통해 확인하였다. MS 유전자형을 이용한 친자검정 기술은 아비를 추정하였을 때, 한 마리의 아비를 특정하지 못하고 여러 마리의 아비를 추정하기 때문에 혈통의 오류 수정이 불가능한 경우가 76% 이상인 것으로 확인되었다. 하지만, SNP chip을 이용할 경우에는 정확하게 한 마리의 실제 아비를 추정할 수 있어 혈통 오류의 수정이 가능해진다. 이 방법은 혈통의 관리는 물론이고, 유전체 기반의 정확한 유전능력평가가 가능하기 때문에 한우 개량을 위한 매우 효율적인 수단으로 활용 될 것으로 사료된다.