서론
최근 우리나라는 국민소득의 증가와 더불어 맞벌이 부부의 증가, 간편식 추구, 삶의 질적 향상에 따른 여가 선호 등으로 식문화가 경량화 및 고급화가 되는 추세이며, 이에 따라 간단하게 먹을 수 있는 육가공품의 수요도 증가하고 있다(Maeng and Huh, 2002, Moon, 1990). 생산현황을 보면 2017년 6,464,658 톤에서 2020년 7,225,545톤으로 꾸준히 증가하여 육가공품 산업이 크게 성장하고 있음을 알 수 있다(aTFIS, 2021).
Yun (2003)이 보고한 내용에 따르면 우리나라 주부를 대상으로 한 설문조사에서 육가공품에 대한 신뢰도는 54.1%로 절반을 조금 넘는 편에 속했는데, 그 이유로는 첨가물 위해 여부 및 제품정보 전달의 부족에 대한 인식이 가장 큰 문제점이라고 보고하였다. 특히 햄과 소시지 같은 육가공품은 어린이 기호식품으로써 소비성이 높아 특정 동물 단백질 알레르기가 있는 소비자에게 큰 문제가 될 수 있으므로 제품의 표시사항이 매우 중요하다(Koh et al., 2013). 따라서 육가공품에 사용된 원료육에 대한 정확한 정보를 제공하는 것이 축산 및 육가공품 시장의 활성화와 소비자 신뢰도 증가에 큰 도움이 될 것으로 생각된다.
최근 식육감별법으로 극소량의 DNA를 통해 특정 종의 염기서열을 증폭함으로써 신속하고 정확한 종 구별이 가능한 PCR (Polymerase Chain Reaction) 분석법이 알려져 있다. 여러 가지 PCR 방법 중 single PCR은 종의 특정 primer를 이용하여 원료육을 분석하는 것으로 여러 연구가 진행되어왔고(Lahiff et al., 2001; Kesmen et al., 2007; Park et al., 2012a; Park et al., 2012b; Nagappa et al., 2014; Kim et al., 2014; Barakat et al., 2014; Heo et al., 2014), multiplex PCR은 두 가지 이상의 primer를 사용하여 다수의 target DNA를 동시에 증폭하는 방법으로 혼합된 시료에서 여러 종을 신속하고 간편하게 검출해 낼 수 있는 장점을 통해 여러 원료육 분석이 진행되어왔다(Markoulatos et al., 2002; Dalmasso et al., 2004; Ghovvati et al., 2009; Koh et al., 2011; Yoon, 2014; Kim et al., 2015). 그러나 두 개의 PCR 방법은 최종적으로 전기영동을 통해 성분확인이 가능하기 때문에 시간적 효율이 떨어지며, 이러한 단점을 보완하여 개발된 것이 Real-time PCR 방법이다.
Real-time PCR 방법은 증폭 산물의 실시간 모니터링과 정량화가 가능하다. 또한, internal control을 이용함으로써 신속하고 정확하게 정량적으로 핵산을 검출할 수 있다(Livark and Schmittgen, 2001). Real-time PCR의 측정방법으로 절대정량과 상대정량이 있다. 절대정량은 DNA 농도 또는 DNA copy number를 이용하여 Cycle quantification (Cq)를 구해 DNA양을 정량하는 방법이며, 상대정량은 양이 일정하다고 가정된 housekeeping gene과 target gene을 검출하여 동일한 시료 내에서 housekeeping gene으로 보정하여 상대적인 발현값을 도출해내는 방법이다. 이러한 특성으로 연구에서의 활용범위가 넓어져 지금은 육가공품에서의 종 식별과 정량에 많이 이용되고 있다(Ballin et al., 2009). 국내에서 실제 판매되고 있는 제품을 대상으로 사용된 원료육을 검출한 선행연구가 있으나, 표기된 축종을 모두 식별한 경우는 적었다(Park et al., 2012b; Yoon, 2014; Heo et al., 2014). 또한, 현장에서 판매하는 육가공품의 Real-time 분석으로 절대정량과 상대정량을 이용한 연구는 미비한 실정이다. 따라서, 본 연구는 Real-time PCR의 상대정량, 절대정량 두 가지 방법을 이용하여 유통시장에서 판매되고 있는 육가공품에 적용 가능성을 검증하였다.
재료 및 방법
1. 공시동물 및 DNA 추출
종 식별 및 효율성 평가로 사용된 원료육은 소(Bos taurus), 돼지(Sus scrofa), 닭(Gallus gallus)이며, 시중에서 판매되는 소고기 12개, 돼지고기 5개, 닭고기 7개 제품을 구매하여 분석에 사용하였다. 육가공품 표기검증을 위해 12개 브랜드의 햄류(8개), 소시지류(23개), 떡갈비류(7개), 패티류(7개), 캔 햄류(7개) 총 52개의 제품을 구매하여 분석에 사용하였다. 추출된 DNA는 NanoDrop ND-1000 spectrophotometer (Thermo, USA)로 흡광도를 측정하여 농도를 확인하였고, 적정 농도로 희석 후 Real-time PCR을 실시하였다.
2. 가열육 제조, DNA 추출 및 검출한계 평가
가열육에 대한 검출한계를 평가하기 위해, 원료육의 소, 돼지, 닭고기를 5 g씩을 잘라내어 식육 가공품의 가공조건을 고려한 70℃에서 30분간 열처리하였다. DNA 추출과 농도 확인 및 희석은 원료육과 같은 방법을 사용하였으며, 가열육에서 추출된 DNA의 검출한계를 시험하고자 적정 농도로 희석하여 Real-time PCR을 실시하였다.
3. Primer 설계
종 특이성 primer는 Clustal W version 2.1 program (Larkin et al., 2007)을 사용하여 NCBI GenBank database에서 제공된 각 종의 mtDNA (D-loop, Cytochrome b)의 염기서열을 Primer3 program (Untergasser et al., 2012)을 통해 정렬한 뒤 이용하였다. Real-time PCR annealing 온도 차이를 최소화시키면서 각각 다른 크기로 증폭되도록 소(Accession No. NC_006853.1), 돼지(Accession No. NC_000845.1)는 Cyt-b, 닭(Accession No. NC_001323.1)은 D-loop에서 설계하였다. 상대 정량 분석을 위한 housekeeping gene은 18S ribosomal RNA를 이용하였는데, 특히 가공식품의 복잡한 성분을 고려하면 보정을 통한 안정된 분석을 위해서는 필수적이다. 육가공품에서 추출한 DNA는 다양한 물질이 뒤섞어져 있는 상태로 품질에 영향을 주는 저해물질, 제품 가열 시 나타나는 DNA 파괴 등에 따라 목적유전자의 증폭 여부가 달라지는데, 이러한 문제점을 개선하는 것이 Housekeeping gene을 사용한 PCR 방법이며(Soares, et al., 2013), 환경적 및 유전적으로 발생 가능한 변화들을 제어하고 목적유전자를 발현시키는 방법으로 다양한 연구 분야에 사용되고 있다(Ballin et al., 2009). 따라서, 본 연구는 Koh 등(2013)의 연구를 참고하여 소, 돼지, 닭의 염기서열을 Clustal W version 2.1로 정렬한 뒤 공통서열을 찾아 설계하였고, Real-time PCR에 사용된 primer의 정보는 Table 1에 나타냈다.
|
Table 1. Information of species-specific PCR primers and 18S rRNA using in this study. 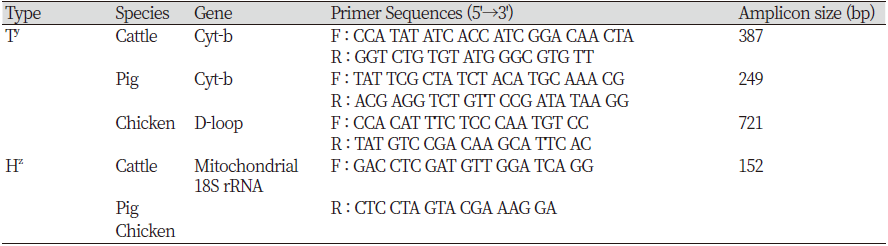
|
|
yTarget primer. zHousekeeping gene. |
4. Real-time PCR 반응액 및 반응조건
원료육과 육가공품의 Real-time PCR은 iQ SYBR Green Supermix (Bio-Rad Laboratories, Hercules, CA)를 이용하였다. Bio-rad에서 제공되는 설명서에 따라 반응액을 조성하였고, Final volume은 10 ㎕이다. PCR 반응조건은 95℃에서 3분간 denaturation을 실시한 후 증폭을 위해 95℃에서 10초간 denaturation, 61℃에서 75초간 annealing, 72℃에서 30초간 extension을 28회 반복하였으며, 60~95℃에서 0.5℃씩 0.05초 차이로 melting curve 분석을 수행하였다. 증폭은 CFX 98 instrument (Bio-Rad Laboratories, Hercules, CA)를 이용하였으며, negative control은 primer마다 수행되었다.
5. 절대정량
Bustin 등(2009)에 의하면 표준 곡선(standard curve)을 구축하기 위해 사용되는 시료는 분석에 사용된 시료와 동일한 유형으로 만들어야 하고 적합한 검량선을 만들기 위해서는 최소 3개 이상의 희석된 시료를 만들어 수행하라고 제안하였다. 본 연구는 각 축종에 따라 추출된 DNA 농도를 8단계로 구분하여 희석하였고, 10회 반복하여 기록하였다.
6. 상대정량
특이적인 PCR 증폭 산물을 확인한 뒤에 원료육과 가공육에 각각 housekeeping primer와 target primer가 적용된 Ct값을 비교하는 ΔΔC(t) 방법을 이용해 각 sample에 대한 유전자 발현율(2-ΔΔC(t))을 확인하였다(Livak and Schmittgen, 2001).
결과 및 고찰
1. 원료육 종 별 특이성
소, 돼지, 닭의 원료육 Real-time PCR 결과 Target primer의 특이성을 확인할 수 있었다(Table 2). 각 축종의 cycle quantification (Cq)값이 소는 16.09, 돼지는 15.41, 닭은 16.24로 나타났으며, Primer 특이성을 확인하기 위한 melting temperature (TM)는 소 80.5℃, 돼지 81℃, 닭 85.5℃로 나타났다. Target primer를 사용한 Real-time PCR에서는 다른 종과 교차반응이 일어나지 않았으며, 종에 따라 특이적으로 증폭된 것을 확인할 수 있었다.
|
Table 2. Species specificity results of raw meat using Real-time PCR. 
|
|
yCycle quantification. zMelting temperature. |
2. 가열육 검출한계
일반적으로 육가공품 제작 시 원료육은 열처리 과정을 거치므로 DNA 추출에 어려움이 따른다. 본 연구에서는 원료육을 열처리한 뒤 추출된 DNA를 이용하여 Real-time PCR에서 검출 반응 양상을 살펴보았다(Table 3). 그 결과, 육가공품에 자주 사용되는 소, 돼지, 닭의 DNA 추출은 전부 가능하였다. 소와 돼지는 최대 0.1 ng/㎕까지 검출되었으며, Cq 값은 각각 25.3, 26.82로 나타났다. 반면, 닭은 최대 1 ng/㎕까지 검출되었으며, Cq값은 24.78로 확인되어 소, 돼지가 닭보다 검출될 확률이 높다는 것을 확인할 수 있었다. DNA 농도에 따라 낮을수록 Cq 값이 높게 나타났는데, 닭의 경우 amplicon size가 다른 축종에 비해 크기 때문에 Cq값이 더 높게 나온 것으로 판단된다.
|
Table 3. Detection limit results of cooked meat using Real-time PCR. 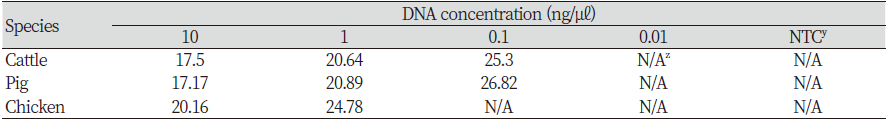
|
|
yNo Template Control. zNo Amplification. |
3. 절대정량과 상대정량
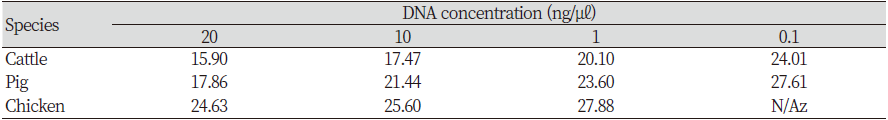
표준 곡선(standard curve)을 구축하기 위해 각 축종에 따라 추출된 DNA 농도를 200, 100, 75, 50, 25, 10, 1, 0.1 ng/㎕로 구분하여 희석하였고, 정량분석을 실시하였다. 축종별로 10회 반복하여 정량분석한 Cq 평균값과 표준편차를 확인하였다(Table 4). 소와 닭은 DNA의 농도가 감소할수록 Cq값이 점차 증가하였으나, 돼지에서 DNA 농도 50 ng/㎕에서 감소하였다가 10 ng/㎕에서 가장 크게 증가하는 것을 확인하였다. 축종에 따른 결과에서는 모든 농도에서 닭의 Cq값이 가장 높다는 것을 확인하였다. Table 5는 육가공품에서 추출한 DNA의 농도를 20, 10, 1, 0.1 ng/㎕로 희석 후 18S rRNA primer 추가하여 Real-time PCR을 통해 상대정량을 분석한 결과를 나타낸 것이다. Table 5를 살펴보면, 소, 돼지를 검출할 수 있는 한계는 0.1 ng/㎕이며, 닭은 최대 1 ng/㎕까지인 것을 확인하였다. 앞선 절대정량 분석과 마찬가지로 모든 축종에서 DNA 농도가 감소할수록 Cq값이 증가하였다. 또한, 모든 DNA 농도에서 소가 가장 낮은 Cq값을 가졌으며, 닭이 가장 높은 Cq값을 가진 것을 확인하였다. 이를 통해 임의적으로 농도를 낮추지 않는 이상 육가공품에서 DNA 추출과 종 식별이 가능한 것으로 판단된다.
|
Table 4. Cycle quantification mean of target primer 10 replication on the Real-time PCR. 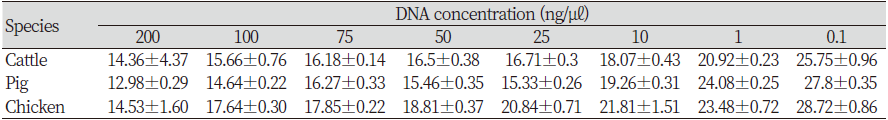
|
|
Mean and standard deviation of Cycle quantification values according to DNA concentration are shown. |
4. 육가공품 DNA 검출
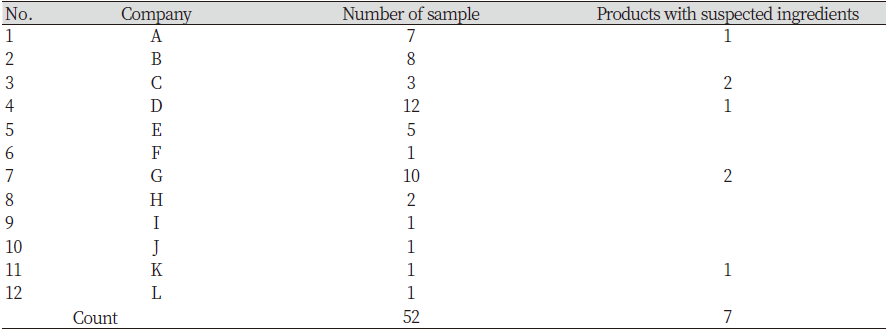
5종류로 분류한 52개의 육가공품을 Real-time PCR 분석을 실시하였고, 잘못된 표기를 사용한 육가공품을 확인할 수 있었다. 브랜드와 제품분류에 따른 잘못 표기된 제품의 수를 Table 6에 나타냈다. 모든 육가공품에서 소와 돼지는 표기된 제품에서 모두 검출되었다. 닭의 경우 제품 표기에는 없었지만, 햄에서 2번과 5번, 소시지에서 2번, 6번, 16번, 떡갈비에서 1번과 4번으로 총 7개 제품에서 검출이 확인되었으며, 표기에 있지만 검출되지 않은 것은 캔 햄 7번으로 확인되었다. 제조사별로 구분했을 때는 총 12개의 제조사로 구분되었고, A사에서 1개, C사에서 2개, D사에서 1개, G사에서 2개, K사에서 1개로 확인되었다.
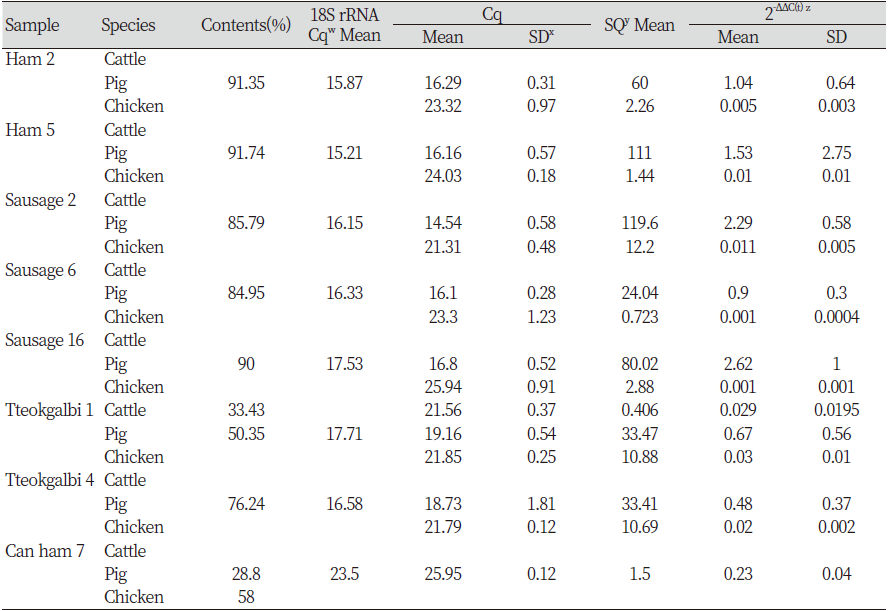
정량분석을 통해 생성된 표준 곡선에 샘플의 Cq값을 대입하여 starting quantification (SQ)값을 추정할 수 있으며, 이를 통해 축종별 DNA의 양을 유추할 수 있다. 표기에 대한 잘못된 제품인 햄 2번, 5번, 소시지 2번, 6번, 16번, 떡갈비 1번, 4번, 캔 햄 7번에 대한 자세한 성분 및 분석결과를 Table 7에 나타냈다.
햄 2번, 5번, 소시지 2번, 6번, 16번, 떡갈비 1번, 4번은 닭의 성분을 미표기한 제품이지만 분석한 결과를 보면 닭이 검출된 것을 확인할 수 있다. 각 육가공품에 따른 18S rRNA Cq 값은 평균 17.36으로 축종에 따른 적정 수준의 DNA가 있는 것을 알 수 있으며, SQ 값에서는 주 원료육의 DNA 함량에 상당히 높게 나타났다. 반면, 미표기된 닭의 Cq의 값은 평균 23.07로 상당히 낮은 수준의 DNA가 함량되어 있는 것을 확인하였다. 그리고 2-ΔΔC(t)값을 살펴보면, 주 원료육의 목적유전자 발현율보다 미표기된 원료육의 발현율이 상당히 낮은 것을 확인할 수 있다. 이를 통해 원래 표기에 닭고기가 있는 제품과 비슷한 값을 가진 제품은 의도적인 혼입이 의심되지만, 더 낮은 값이 검출된 제품은 비의도적으로 함유될 가능성도 있다는 것을 확인하였다. Koh 등(2013)의 보고에 따르면 비의도적 혼입이란 제조과정 중 기계의 세척이 제대로 되지 않아 잔여물이 남았거나 칼이나 분쇄기를 공동 사용하여 발생할 수 있는 혼입이라고 규정하였고, 이에 따른 식육원료의 의도적, 비의도적 혼입을 원료혼합방법과 유전자추출 뒤 혼합방법을 통해 비교하였을 때 유의적 차이가 없음을 검증한 다음 돼지 DNA에 닭 DNA를 100 ~ 0.1%까지 혼합한 후 Cq 값을 비교하였다. 비의도적 수치를 1%라고 가정하였으며, 100% 시료와의 cycle 수가 4 cycle 이내라면 의도적 혼입이라고 보았고 6 cycle 이상이라면 비의도적 혼입이라고 예상하였다. 본 연구에서는 떡갈비류를 제외한 육가공품에서는 평균 7.6 cycle 이상 차이가 났으며, 떡갈비류는 약 3 cycle 정도가 차이가 난 것을 확인하였다. 이를 통해 떡갈비류는 의도적 혼입으로 의심할 수 있으나, 반복적인 실험을 추가하여 정확한 결과를 확인할 필요가 있다고 사료된다.
캔 햄 7번은 돼지, 닭이 표기되었지만, 돼지만 검출되었다. 가열육에서 DNA 추출이 원활하지 않았거나 캔 가공제품의 특성상 보존 및 신선도를 오래 하기 위해 살균을 목적으로 방사선 조사를 할 가능성도 있는데, 이는 방사성 물질이 붕괴할 때 나오는 γ선의 이온화 에너지를 식품에 쐬면 식품의 주형 DNA 구조가 파괴되고 이와 동시에 포름알데히드, 벤젠 등 PCR 저해물질들이 생성되어 식품 중에 남게 되어 나타난 결과로 판단된다(Yoon, 2014). 또한, 육가공품을 제조할 때 각각 어느 부위를 이용하는지는 표기하지 않으며 아무리 균일하게 혼합되었다고 하여도 DNA 추출 시 세포, 조직에 따라 mtDNA 양이 다르므로 농도 차이가 있을 것으로 판단된다. Ballin 등(2009)은 정량분석에서는 핵에 있는 single-copy genomic DNA를 이용하여 유전체 대 유전체로 표현하라고 권장하였다. 하지만 그것은 근육 외 다른 조직에서 추출되는 DNA 양 또한 차이가 나기 때문에 다른 부위가 육가공품에 혼합된다면 정확한 혼합비율을 알 수 없다고 보고되었고(Laube et al., 2007), 실제 육가공품에 쓰이는 부위를 정확히 알고 그 원료육과 동일한 조건에서 가공한 고기를 이용하여 표준 곡선으로 분석한다면 가장 정확하겠지만 실제적으로는 어려운 상황이다(Prado et al., 2009). 따라서 본 연구에서 검출된 DNA 양은 모든 원재료가 근육이라는 가정하에 신뢰성을 얻을 수 있을 것으로 사료된다.
결론
이번 연구를 통하여 육가공품에서의 종 식별 마커의 적용 가능성을 확인하였으며, 총 5종류 52개의 육가공품을 대상으로 Real-time PCR을 수행한 결과로 캔 햄 7번을 제외하고 표기된 성분이 모두 검출이 되었으며, 제품 표기와 다른 성분이 있는 제품 7가지를 확인하였다. 이를 통해 미표기된 원료육이 혼합될 가능성을 확인할 수 있었다. 현재 식품 공정에 관한 법령의 경우 육가공품의 제조 기준과 포장재 표기 사항 등의 내용은 기재되어 있지만, 원료 성분을 확인할 분석방법에 관한 규정은 없는 실정이다. 따라서, 이러한 검사법을 통해 최종 육가공품의 원료 종류 및 함량을 분석할 수 있는 기술이 필요한 것으로 보이며, 소비자 관점에서 동일한 유형의 육가공품에 대하여 사용한 원료육의 품질구분이 어렵다는 점을 고려했을 때 고기 함량 표기에 세부사항까지 표기할 수 있도록 하는 규제가 필요한 것으로 사료된다.