Introduction
칡소는 우리나라의 토종 한우의 아종 으로 질병에 강하고 힘이 세다는 특징이 있다. 온몸을 둘러싼 검은색 줄무늬가 호랑이와 비슷해 ‘호반우(虎班牛)’라고도 불려 왔다. 우리나라의 역사적 기록에 따르면 고구려 전기 시대(서기357년)의 고분벽화 ‘안악 3호분’에는 검정소, 누렁소 및 얼룩소가 채색 벽화로 남아있으며, 이 벽화에 그려진 소는 견봉이 없고, 가는 목과 뿔, 경사진 엉덩이 등의 특징으로 현재 한우와 같은 형태를 지니고 있다(Suh et al., 2015). 일제강점기를 거치며 모색 호칭의 통일과 모색 단일화 조치로 다양한 한우의 모색이 갈색으로 통일되어갔으며(Kim et al.,2011), 1970년 한우 심사표준에서는 한우의 모색을 황갈색으로 규정하여, 모색 규정에 따라 종축을 선발하였고, 유전적 다양성의 중요성은 배제된 상태에서 황갈색의 한우만 등록함으로 이모색의 종축들은 대부분 도태되어 황갈색 이외의 재래 소의 개체 수는 100두 이하로 급감하였다(Shon et al., 2000). 1980년대 중반 이후 일부 소수 농가에 의해 재래 가축의 수집, 보존 및 증식을 통한 복원사업이 일부 연구소를 중심으로 시작되어, 그 결과 칡소의 개체 수는 증가하게 되었고, 2020년 기준 칡소는 전국적으로 대략 4000두 정도 사육되어지고 있다. 이러한 역사를 갖고 있는 칡소 품종은 2004년 국제식량농업기구(FAO)에 등재되었다(Choi et al.,2021, Suh et al.,2014). 전 세계적으로 경제 가축 품종에 대한 개량은 각 국가 별 소비 트랜드에 맞게 진행이 되면서 유전적 다양성이 급감하는 추세이다. 소비트렌드의 변화, 유전적 다양성의 급감, 기후변화, 질병의 창궐 등으로 축산업은 다양한 환경적 위기 요소들과 직면하게 되었다(Oh et al.,2008). 우리나라 한우 산업 또한 국가단위의 체계적인 개량 시스템을 통해 안정적인 개량이 이루어지고 있지만, 산업 전반의 절대적인 비중이 한우(황갈색 한우)에 집중되어 있어, 유전적 다양성 확보에 대한 문제는 아직도 과제로 남아 있는 것이 현실이다. 최근 국내 육류 소비 트렌드가 고급화, 다양화 중심으로 빠르게 변화하고 있어, 우리나라 육류 산업의 경쟁력을 높이기 위해서는 세대별 육류의 소비 격차를 완화하고, 육류 생산의 다양성을 확보해야 한다는 주장이 제기되고 있다. 이와 같은 추세에 대응하기 위해서는 경쟁력을 갖춘 다양한 유전자원의 확보가 매우 중요한 요소들 중 하나이며, 국내 재래 품종의 효율적인 생산과 관리 시스템의 구축을 통해 다양한 유전자원의 확보가 가능할 것으로 사료된다. 하지만 한우와 토종 닭을 제외한 다른 재래 품종의 경우 산업적 기반이 매우 미흡한 수준이며 장기적인 안목에서의 연구와 투자가 필요한 시점이라 할 수 있겠다. 칡소의 경우 분만과 포유 능력 및 육질이 뛰어나며, 사육 희망 농가도 증가하고 있는 추세이다(Park et al.,2010). 하지만 산업적 활용을 위해서는 아직 해결해야할 문제가 많은 것이 현실이다. 한우에 비해 경제적인 능력이 상당히 미흡한 수준이며, 개량을 위한 기초 데이터도 아직은 미흡한 수준에 있는 것이 현실이다. 이러한 문제의 해결을 위해 칡소의 유전적 특성을 명확히 파악하고 개량의 방향과 체계를 구축해야 한다. 하지만 관련된 연구가 아직까지도 미흡한 수준에 머물러 있는 것이 현실이다. 현재까지 칡소의 유전 특성과 관련된 연구는 모색 유전자의 발현, 칡소 집단(지역)간의 유전적 구조의 차이 그리고 유전적 다양성 등에 대한 연구가 일부 보고된 바가 있으나(Park et al.,2012, Lee et al.,2013, Suh et al., 2014, Suh et al.,2021), 한우에 비해 매우 미흡한 수준이다. 2017년 시작된 칡소 복원 사업을 통해 품종의 복원이 완료되었으며, 현재는 축군의 증식 및 개량을 통해 개체 수를 확대시키는 과정에 있다. 한우산업의 유전적 다양성을 확대하고 변화하는 소비 트랜드를 반영하기 위해서는 칡소의 개량과 증식 과정에서부터 구체적인 전략을 수립하고 이에 따른 체계적인 시스템의 마련이 필수적이라 할 수 있다. 이러한 시스템의 구축을 위해서는 칡소 집단의 복원과 개량 과정에 발생한 유전적 특성의 변화와 현황을 정확히 파악할 필요성이 있으며, 이를 바탕으로 개량의 방향을 설정하고 구체적인 전략의 수립이 가능하다. 따라서 본 연구는 칡소 집단의 세대별 분류와 분석을 통해, 복원과 증식 과정 전반의 유전적 특성 및 다양성의 변화를 확인하여, 칡소 유전자원의 산업적 활용 전략 수립의 기초 자료로 활용하고자 실시하였다.
Materials & Methods
1. 공시 재료
본 연구에서는 충청북도, 전라북도의 축산위생연구소와 강원도, 충청남도 그리고 경상북도의 축산기술연구소, 축산과학원 가축개량과, 한우개량사업소에서 1990년도부터 2020년까지의 국내에서 사육· 도축된 칡소 중, Microsatellite Marker 유전자형(11좌위) 분석을 실시한, 총 9,669두의 유전자형 정보를 제공받아 분석에 이용하였다.
2. 출생 년도별 집단 분류
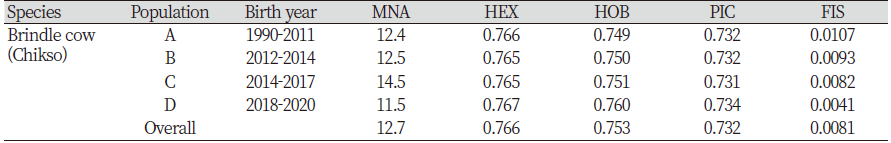
본 연구는 칡소 품종 유전자원의 복원 이후 현재까지의 증식 과정 중 유전적 특성과 다양성의 변화를 분석하기 위해 출생 년도를 기준으로 4개의 그룹으로 나누어 분류하였다. A그룹(1990-2011년) 1,644두, B그룹(2012-2014년) 2,262두, C그룹(2015-2017년) 3,318두, D그룹(2018-2020년) 2,439두으로 설정하였고, A그룹은 명종위기 상태의 칡소를 증식하는 과정에서 개체의 수가 많지 않아 1990-2011년도에 출생한 개체들을 하나의 그룹으로 설정하였으며 자세한 내용은 Table 1에 제시하였다.
3. Microsatellite Marker
개체의 유전적 다형성을 분석한 Microsatellite Marker는 “농림수산식품부 고시 제 2009-26호”에 의해 시행되어지고 있는 축산물 이력제에 사용되는 11종 (BM1824, BM2113, ETH10, ETH225, ETH3, INRA23, SPS115, TGLA122, TGLA126, TGLA227, TGLA53)의 DNA Marker이다.
4. 통계분석
Microsatellite Toolkit software (Park,2000)와 GeneAlEx ver6.5 (Peak and Smouse,2006)를 이용하여 분석 개체 별, 집단 별 분류한 후 Cervus ver2.0 program을 이용하여 관측 이형접합율(Observed heterozygosity ;HOB), 기대 이형접합율(Expected heterozygosity ; HEX)), 유전적 다형성 정보지수(Polymorphism information content ;PIC) 결과 값을 산출하였다. 그리고 칡소와 한우 집단의 유전적 병목현상(Bottleneck)을 추정하기 위해 Bottleneck ver1.2.02 (Piry,1999)을 이용하여 Infinite Allele Model (IAM), Two Phase Model (TPM), Stepwise Mutation Model (SMM)의 방식으로 결과 값을 산출하였다.
Results and discussion
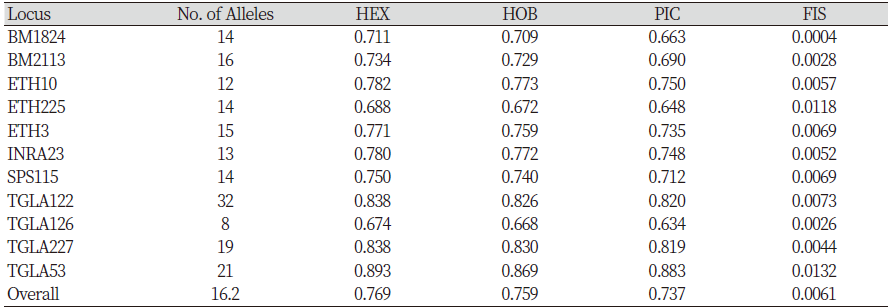
본 연구는 칡소의 복원과 증식 과정 전반의 유전적 특성 및 다양성의 변화를 확인하기 위해 수행하였다. 국내에서 사육된 칡소 9,663두의 11개 Microsatellite marker의 유전자형 정보를 제공받아 유전자 다형성 분석에 활용하였다. 칡소 집단의 각 마커별 대립유전자의 수(No. of Allele), 유전적 다양성 지수인 기대이형접합율(HEX), 관측이형접합율(HOB), 다형정보지수(PIC) 및 FIS값을 계산하여 Table 2에 제시하였다. 대립유전자의 수를 분석한 결과 평균 16.2개로 확인되었으며, 가장 많은 대립유전자수를 보유한 마커는 TGLA122로 32개의 대립유전자를 보유하였고, 가장 작은 것은 TGLA126으로 8개의 대립유전자를 보유한 것으로 확인되었다. 서 등(2014)은 울릉도 지역의 칡소 52두를 대상으로 분석한 결과 가장 많은 대립유전자수를 보유한 마커는 TGLA122, TGLA53으로 14개 보유하고 있고, 가장 작은 것은 BM1824로 5개를 보유하고 평균 8.09개로 보고되었다. 최 등(2015)에서는 전국에 사육되어진 칡소 개체 2,086두를 대상으로 분석한 결과 가장 많은 대립유전자수를 보유한 마커는 TGLA122에서 24개, 가장 작은 것은 BM1824로 8개를 보유하고 평균 13.18개로 보고되었다. 이전에 보고된 칡소 집단의 대립유전자 수에 비해 본 연구의 대립유전자의 수가 많은 것으로 확인되었다. 서 등(2014)의 보고에서는 52두를 대상으로 평균 8.09개, 최 등(2015)의 보고에서는 2,086두를 대상으로 평균 13.18개, 본 연구에서는 9.663두를 대상으로 평균 16.2개의 대립유전자가 확인되었다. 이는 집단의 크기가 대립유전자의 수에 영향을 미친다는 보고 (Schmid, 1999)가 있었으며, 또한 본 연구의 경우 2015년도에 보고 된 최 등(2015)의 연구 이후 2020년까지의 누적된 개체들의 유전자형 정보가 포함되어 기인한 것으로 추정할 수 있다.
관측이형접합율(HOB)의 평균은 0.759, 기대이형접합율(HEX)의 평균은 0.769로 확인되었다. 관측이형접합율(Hob)의 평균 값이 기대이형접합율(HEX)의 평균 값보다 낮은 것을 확인하였다. 이전에 보고 된 칡소의 관측이협접합율(HOB)과 기대이형접합율(HEX)의 결과를 확인해보니, 서 등(2021)은 관측이형접합율(HOB)의 평균은 0.753과 기대이형접합율(HEX)의 평균은 0.765, 최 등(2015)은 관측이형접합율(HOB)의 평균은 0.753과 기대이형접합율(HEX)의 평균은 0.766으로 보고 되었다. 선행연구와 본 연구 모두에서 칡소 집단은 관측이형접합율(HOB)이 기대이형접합율(HEX)에 비해 낮은 것으로 확인되었다. Hardy-Weinberg Equilibrium에 따르면 기대치(HEX)보다 관측치(HOB)가 낮으면 이형접합체 결손 현상이 발생했다고 할 수 있다. 기대이형접합율(HEX)은 유전적 평형 상태에서 기대되는 이형접합율을 계산한 값이며, 관측이형접합율(HOB)은 실제 집단에서 관측되어진 이형접합율을 계산한 값이다. 경제 가축의 경우 우수한 종축을 선발하고, 선발되어진 한정된 종축을 이용하여 계획교배가 이루어지기 때문에 관측이형접합율(HOB)이 기대이형접합율(HEX)보다 작아지는 현상이 발생할 수 있다. 이러한 현상은 유전적 다양성 감소와 근친도 증가의 지표로 활용되어 진다. 이 집단의 유전적 다양성이 존재하는지 판단 할 수 있는 다형정보지수(PIC)는 평균은 0.737로 가장 낮은 BM1824의 값은 0.663, 가장 높은 TGLA53은 0.883의 값이 확인되었다.
칡소 집단의 유전적 고정 및 근친의 정도를 확인하기 위하여 F-statistics 분석을 통해 FIS 값을 확인하였다. FIS 값이 1에 가까운 양의 수이면 개체들간의 상관정도가 높고, 근친도의 위험이 존재하고 있음을 의미한다. Table 2에 제시한 칡소 집단 FIS 값의 평균은 0.0061으로 확인되었다. 2008년도에 이 등(2008)이 보고한 한우 지역 브랜드 개체들의 FIS 값의 평균이 0.05인 것과 비교한다면, 최근에 복원 사업이 마무리되어 증식과 개량 과정에 있는 칡소 집단의 FIS 평균이 0.0061로 확인되었다는 것은 유전적 다양성과 근친의 위험성에 대한 관리가 비교적 잘 관리되고 있는 것으로 추정되어 진다.
오랜 시간 개량되어진 한우를 칡소와 비교해 보았을 때 어떤 분석 결과 차이가 존재하는지 확인해보았다. 신 등(2018)은 1995년부터 2015년까지의 보증씨수소 844두를 분석한 결과 대립 유전자의 수는 평균 10.54로 본 연구의 대립유전자 수의 평균보다 높게 나왔고, 기대이협접합율(HEX)과 관측이형접합율(HOB)은 0.764, 0.773로 본 연구 평균 값의 양상과는 반대로 이형접합체 초과 현상이 발견되었다. 다형정보지수(PIC)는 0.727로 본 연구의 결과 값보다 작았다. 그리고 Fis의 값은 본 연구의 결과는 양수이지만 선행 연구에서는 -0.014로 음수의 값이 보고되었다.
칡소의 유전적 특성 및 다양성의 추세 변화를 확인하기 위해 4개의 세대별 그룹으로 나누어 분석한 결과를 Table 3에 제시하였다. 칡소의 세대집단별 보유 대립유전자수의 평균 값의 변화를 살펴보면, A그룹(1990-2011년)과 B그룹(2012-2014년)은 유사한 반면, C그룹(2015-2017년)에서 갑자기 증가한 것을 확인하였으며, D그룹(2018-2020년)에서는 현격히 감소한 것으로 확인되었다. 기대이형접합율(HEX)의 평균은 0.766으로 확인 되었으며, 모든 그룹에서 큰 변화 없이 평균과 비슷한 추세를 확인할 수 있었다. 관측이형접합율(HOB)의 평균은 0.753으로 확인되었으며, A그룹(0.749)에서 C그룹(0.751)으로 서서히 증가되었고, D그룹(0.760)에서 가파르게 상승한 것이 확인되었다. 다형성정보지수(PIC)의 평균은 0.732로 확인되었으며, 가장 최근에 출생한 집단인 D그룹(2018-2020년)이 0.734로 가장 높은 값이 확인되었다. 마지막으로 FIS 값은 A그룹(0.0107)에서 D그룹(0.0041)으로 오면서 감소하는 추세를 확인할 수 있었으며, 가장 최근에 출생한 집단(D그룹)의 감소 폭이 가장 큰 것으로 확인되었다.
A그룹(1990-2011년)은 칡소의 복원 사업의 시작 시점이 포함되어 있고, B그룹(2012-2014년)은 지역별 일정 수준의 증식이 이루어진 시점이라 할 수 있으며, C그룹(2015-2017년)과 D그룹(2018-2020년)은 개량을 위한 혈통 정립 및 기반 구축을 위한 사업이 시작된 시점이라 할 수 있다. 이러한 세대의 흐름과 맞물려 분석된 결과를 정리해보면, C그룹(2015-2017년)에서 급격하게 대립유전자의 수가 증가했던 것은 칡소 복원 사업을 진행한 각 지역 기관들이 연계 사업을 추진하여 유전자원이 서로 공유되는 과정에서 기인한 것으로 추정되며, 대립유전자의 수는 급격히 증가한 반면, 유전적 다양성에 관련된 지표들(HEX, HOB, PIC, and FIS)은 B그룹(2012-2014년)에 비해 큰 변화가 없었던 것으로 확인되었다. 이는 여러 지역의 유전자원들이 공유됨에 따라 단지 대립유전자의 수가 증가했을 뿐 유전적 다양성의 증감에는 영향을 미치지 못한 것으로 추정이 된다. 반면, D그룹(2018-2020년)에서는 C그룹(2015-2017년)에 비해 대립유전자의 수는 급격히 감소한 것이 확인되었고, 유전적 다양성에 관련된 값들의 변화가 확인되었는데, 기대이형접합율(HEX), 관측이형접합율(HOB,), 다형성정보지수(PIC) 값은 증가하였고, FIS 값은 감소한 것으로 확인되었다. 대립유전자의 수는 감소한 반면, 유전적 다양성의 지표의 값이 증가하였다는 것은 개체들의 선발과 도태를 통해 유전적으로 열등한 개체들이 제거되고, 체계적인 계획 교배가 이루어져 유전적 다양성이 관리되고 있음을 나타내는 자료로 활용 가능할 것으로 판단된다. 또한, 기대이형접합율(HEX)에서 관측이형접합율(HOB)을 뺀 수치가 A그룹에서는 17, B그룹에서는 15, C그룹에서는 14 그리고 D그룹에서는 7으로 확인되어, 이형접합체 결손 현상이 최근으로 오면서 점점 줄어들고 있음을 확인 할 수 있었으며, FIS 또한 최근 세대로 오면서 점점 낮아지는 것이 확인되었다. 이러한 결과들은, 품종 복원에 맞추어져 있던 칡소 집단에서 품종 개량을 위한 집단으로 서서히 변화해 가고 있음을 간접적으로 시사하는 것으로 추정된다.
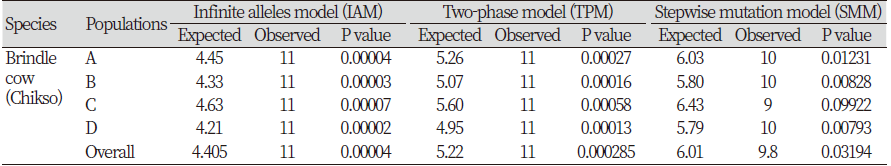
마지막으로, 칡소의 복원사업이 진행되는 과정에서 한정된 칡소 개체를 이용한 증식 등으로 인해 유전적 병목 현상과 관련된 문제는 없는지 Bottleneck test를 실시하였다. Bottleneck test 역시, 출생 년도를 기준으로 한 4개의 집단을 대상으로 실시하였으며, IAM (Infinite alleles model), TPM (Two-phase model), SMM (Stepwise mutation model)을 이용해 분석된 결과를 Table 4에 제시하였다. SMM의 C그룹(2015-2017년)을 제외한 모든 결과들은 0.05 이하의 p 값이 확인되어 유의적 수준에서 유전적 병목 현상에 대한 문제가 없는 것이 확인되었다. 하지만 SMM의 C그룹(2015-2017년)에서는 p 값이 0.09922로 확인되어 다소 유전적 다양성에 대한 문제가 발생했던 것으로 확인되었다. 이는 앞서 분석된 결과에서 제시된 바와 같이, 지역간 칡소 유전자원의 교류가 시작되면서 일시적으로 대립유전자의 수가 증가했던 것에서 기인한 것으로 해석되어진다. 그리고 바로 다음 세대인 D그룹(2018-2020년)에서는 다시 유의적인 수준의 유전적 병목 현상에 대한 문제가 나타나지 않은 것으로 확인되었다. 칡소 집단의 Bottleneck test 분석 결과는 현재 유전적 병목현상에는 큰 문제는 없어 보이지만, 품종의 유전적 다양성 관리가 체계적이고 지속적으로 이루어지지 못할 경우에는 유전적 병목 현상이 바로 발생 할 수도 있다는 것을 시사하는 중요한 자료인 것으로 사료된다.
Conclusions
우리 나라 한우 산업은 국가 단위의 체계적인 개량 시스템을 통해 안정적인 개량이 이루어지고 있지만, 산업 전반의 절대적인 비중이 한우(황갈색 한우)에 집중되어 있어, 유전적 다양성 확보에 대한 문제는 아직도 과제로 남아 있는 것이 현실이다. 따라서 칡소 유전자원의 복원과 개량 사업은 한우 산업의 유전적 다양성 확보를 위해서 매우 중요한 역할을 할 것으로 기대되고 있다. 본 연구는 칡소의 복원과 증식 과정 전반의 유전적 특성 및 다양성의 변화를 확인하기 위해 수행하였다. 칡소 집단의 세대별 관측이형접합율(HOB)의 평균은 0.753으로 확인되었으며, A그룹(0.749)에서 C그룹(0.751)으로 서서히 증가되었고, D그룹(0.760)에서 가파르게 상승한 것이 확인되었다. 다형성정보지수(PIC)의 평균은 0.732로 확인되었으며, 가장 최근에 출생한 집단인 D그룹(2018-2020년)이 0.734로 가장 높은 값이 확인되었다. FIS 결과 값 또한 최근 세대로 오면서 점점 낮아지는 것이 확인되었다. 이러한 결과들은 품종 복원에 맞추어져 있던 칡소 집단에서 품종 개량을 위한 집단으로 서서히 변화해 가고 있음을 간접적으로 시사하는 것으로 추정된다.