Introduction
가축의 개량에 있어서 가장 중요시 여겨지는 것은 경제적으로 유용한 형질을 선발하고 생산성을 떨어뜨리는 불량형질을 도태시키는 데 있다. 이런 불량형질의 대표적인 예로는 유전 질병이 있으며 유전 질병의 제거는 가축의 건강뿐만 아니라 농가의 잠재적인 재정손실을 막는 데 큰 도움을 줄 수 있다. 한국 고유의 유전자원이며 대표 육우인 한우의 경우, 선발 및 교배 시 유전 질병을 일부 조사한 연구내용은 있지만 이를 지속적으로 확인 하거나 도태시키는 연구가 진행되지는 않고 있는 실정이다(Jung et al., 2005). 따라서, 현재의 한우 개량 시스템에서 유전 질병의 확인 및 제거는 불가능한 상태이고, 이를 확립하는 일은 한우의 지속적인 개량 및 생산성 향상에 꼭 필요한 연구임을 의미한다. 한우와 유전적 거리가 가까운 일본 화우의 경우 상당수의 유전병에 관한 연구가 보고되고 있으며 이를 선발 및 도태에 적극적으로 활용하고 있음이 보고되고 있다(Kunieda. 2005). 이러한 결과는 한우에서도 유전 질병과 관련된 원인 변이가 확인될 수 있을 가능성을 뒷받침하고 있으며 지속적인 유전 질병 원인 유전자를 탐색하고 모니터링할 필요성이 있을 것으로 판단된다. 따라서 본 연구는 일본화우에서 밝혀진 유전 질병을 모니터링하기 위하여 Haemophilia A, Chediak-Higashi syndrome (CHS), Spherocytosis, Xanthinuria Type II 질병과 유전변이의 존재 유무를 한우에서 탐색하기 위하여 실시하였다.
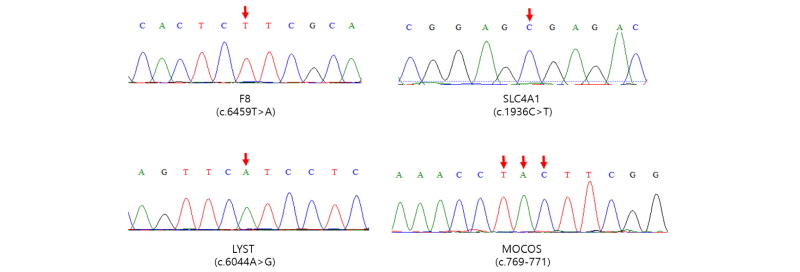
Haemophilia A은 X 성염색체에 위치한 Blood coagulation factor VIII (F8) 유전자 변이에 의해 발병하게 되는 출혈성 성염색체 유전 질병이다. 소의 Haemophilia A 경우 F8 유전자의 단일염기변이의(c.6459T > A) 결과로 발생한 아미노산의 변화(p.Leu2153His)가 질병의 원인이 된다(Khalaj et al., 2009). Haemophilia A 변이가 확인되는 개체는 F8의 활성도가 정상개체에 비해 유의적으로 낮은 수치를 보이며, 관절 및 근육, 내부장기에서 자연적인 출혈이 발생하고, 상해가 발생하면 출혈 시간이 연장되는 증세를 보인다.
Chediak-Higashi syndrome (CHS)은 lysosomal trafficking regulator (LYST) 유전자 변이에 의해 발병하게 되는 출혈성 상염색체 유전질병이다. CHS는 LYST 단백질의 기능 이상에 의한 탈과립 장애로 면역력 감소 및 부분 백색증, 출혈증상 등을 나타낸다고 알려져 있다. 소의 CHS의 경우 28번 상염색체에 위치한 LYST 유전자의 단일염기변이에(c.6044A > G) 의한 아미노산의 변화(p.His2015Arg)가 질병의 발병원인이 된다(Kunieda et al., 1999).
Spherocytosis는 적혈구의 구조를 유지하는 Solute carrier family 4 member 1(SLC4A1) 유전자 변이에 의해 나타나는 상염색체 유전질병이다. Spherocytosis 변이가 확인되는 개체는 적혈구 특유의 오목한 타원형의 형태를 유지하지 못하고 구형의 적혈구의 모양을 나타내게 되고 이는 혈중 가스교환에 영향을 주어 비교적 경미한 산독증 증세와 빈혈증세를 보인다. 소의 경우 19번 상염색체에 위치한 SLC4A1유전자의 단일염기변이(c.1936C > T)에 의한 것으로 이 변이는 유전자 발현과정에서 조기에 종결코돈을 형성해 질병 발병의 원인이 된다(Inaba et al., 2015).
Xanthinuria Type II는 퓨린 대사에 관여하는 Molybdenum cofactor sulfurase (MOCOS) 유전자의 결실 변이에 의해 나타나는 상염색체 유전질병이다. Xanthinuria Type II변이가 확인되는 개체는 크산틴뇨증을 보이며, 요로결석 등의 증상이 나타난다. 소의 경우 24번 상염색체에 위치한 MOCOS 유전자의 3bp (c.769-771) 크기의 아미노산 결실 변이가 원인이 된다(Watanabe et al., 2000).
앞서 언급한 4개의 유전질병인 Haemophilia A, Chediak-Higashi syndrome (CHS), Spherocytosis, Xanthinuria Type II 유전자의 변이를 확인하기 위해 영암군 10개의 한우농장에서 한우 보증종모우 (KPN)와 농가 자체 내 종빈우 사이에서 생산한 F1 개체 376마리를 이용하였다. Genomic DNA (gDNA) 추출을 위해 준비 된 혈액 샘플을 300g의 속도로 15분간 원심분리를 실시하여 중간부에 위치한 백혈구 층을 분리하고 PrimePrepTM Genomic DNA isolation kit (Genetbio, Korea)를 이용하여 gDNA를 추출하였다. 추출된 gDNA는 Nanodrop 2000c 분광흡도계(Thermo Scientfic, USA)를 이용하여 농도 및 품질을 측정하였다. 각 유전 질병에 따라 비교적 분석이 용이한 F8과 LYST 유전자는 PCR-RFLP 기법을 이용하고 SLC4A 1 및 MOCOS 유전자는 RFLP를 위한 restriction enzyme 확보가 어려워 KASP assay genotyping 기법을 각각 이용하였다. QC 과정을 거친 gDNA 샘플은 conventional PCR증폭과 KASP genotyping을 위해 25 ng/µL 및 5 ng/µL로 각각 희석을 실시하였다. F8과 LYST유전자의 경우 PCR 증폭을 위해 총 볼륨이 20 µL 인 Mixture를 제작하였다. Mixture는 genomic DNA (25 ng/µL) 2 µL, Prime Taq 10X buffer 2 µL, 2.5mM dNTP 2 µL, forward 및 reverse 프라이머(10pmol/µL)를 각각 0.8 µL, 2.5 unit의 Prime Taq polymorase를 이용하였다. PCR 증폭 조건은 F8 및 LYST 모두 95°C로 5분간 예비변성 후 95°C 30초, 60°C 30초, 72°C 30초씩 총 35 cycle 실시후 72°C에서 5분간 최종 증폭을 시행하였다. SLC4A 1 및 MOCOS 유전자의 경우 변이 발생 타겟좌위를 증폭할 수 있는 assay를 제작하였으며 Bio-Rad real-time PCR (Bio-Rad, USA)을 활용해 타겟좌위의 증폭 유무를 판단해 Allele discrimination analysis로 서로 다른 유전자형 구분을 수행하였다.
한우의 Haemophilia A, Chediak-Higashi syndrome (CHS), Spherocytosis, Xanthinuria Type II 의 유전 질병의 유무를 확인하기위해 농가에서 보유하고 있는 한우 376마리의 유전자형 분석을 실시해본 결과 다행히 탐색한 모든 유전 질병에 대하여 이형접합 또는 열성동형접합 상태의 개체가 확인되지 않았다. 이전의 연구결과를 확인해보면 78두의 한우 보증종모우(KPN)와 22두의 일반농가 한우를 대상으로 Spherocytosis 및 CHS 유전변이를 확인한 결과 두 연구집단 모두에서 유전질병과 관련된 변이가 확인되지 않았음을 알 수 있었다(Jung et al., 2005; Lim et al., 2017). 반면, 일본 화우의 연구에서는 2800두 이상의 연구집단에서 CHS, Spherocytosis, Xanthinuria Type II의 유전 질병 관련 변이를 조사한 결과 CHS는 4.7%, Spherocytosis는 2.7%, Xanthinuria Type II는 0.1%의 개체가 유전 질병과 관련된 원인 변이를 포함하고 있는 것으로 보고되었다(Sasaki et al., 2004). 이러한 결과들은 일본 화우에서 유전 질병의 비율이 상대적으로 낮은 비율로 존재함을 의미하고, 한우에서 는 아직 직접적으로 확인되지 않았지만 낮은 비율로 존재할 수 있음을 시사한다. 따라서 본 연구에서 조사한 유전 질병의 경우 추가샘플을 확보해 질병의 유무를 조사할 필요가 있으며 본 연구에서 조사한 질병 이외에도 한우가 보유 할 가능성이 있는 유전 질병 유전자를 탐색하고 지속적인 관측이 필요 할 것이라고 판단된다. 더불어 한우의 질병관련 유전자를 조사하여 한우의 개량과정에 적극적으로 활용한다면 한우의 생산성 향상과 농가의 잠재적 재정손실을 예방할 수 있을 것으로 사료된다.