Introduction
충남 논산시 연산 화악리 지역의 오계는 1980년 천연기념물 제 265호로 지정된 한국의 가금류 중 유일한 천연기념물이다. 이러한 연산 오계는 현재 미국 FAO (Food and Agriculture Organization of the United Nations)의 가축다양성시스템(Domestic Animal Diversity Information System;DAD-IS)에 등재되어 있으며 깃털, 피부, 벼슬 및 내장 기관과 뼈까지 모두 검은색을 띄는 독특한 가금 유전자원이다.
하지만, 최근 연산 오계는 산란율 저하와 함께 성비의 불균형으로 집단보존에 어려움을 겪고 있다. 이론적으로 집단의 성비는 암, 수에 관계없이 동일한 성비를 갖는다고 알려져 있다(Fisher, 1930). 그러나 이러한 성비의 불균형은 성체 도달 전까지의 성별 에 따른 사망률의 차이가 클 때 발생하며, 이는 품종 보존이나 집단의 유지 과정에 bottle neck 효과로 악영향을 줄 수 있다(Ewen et al., 2001). 현재 오계의 경우 육성 과정중의 암수의 폐사율의 차이가 적고 육성중인 성계는 암컷보다 수컷의 비율이 상대적으로 높아 발생중지란의 성별 확인을 통해 발생 과정중의 배아 사망률의 확인이 필요할 것으로 사료되었다
병아리와 같이 암수의 외형이 유사하고 생식기의 특징이 확인하기 어려운 동물의 경우 외형만으로 성별의 판단이 불명확하고 어렵다고 알려져 있다(Elliott and Norris-Caneda, 1998). 일반적으로 초생추의 자웅감별을 위하여 감별사에 의한 생식돌기의 차이를 감별하는 방법이 있으나 숙달된 전문가의 도움이 필요하고 발생중지란의 경우 성별판단이 불가능에 가깝기 때문에 실험실에서 신속하고 정확한 발생중지란의 성별을 판단하기 위해서는 유전자 마커를 활용한 성별의 확인이 요구된다. 이러한 닭의 성별 확인에 용이한 방법으로 Chromo-helicase DNA-binding protein (CHD)유전자를 이용하는 것이다. 조류의 CHD 유전자는 성염색체 Z와 W에 각각 존재하며, 성별에 따른 발현의 차이가 없는 반면 염기서열에서는 부분적으로 차이를 나타낸다고 알려져 있다(Garcia-Moreno and Mindell, 2000; Boutette et al., 2002). 따라서 본 연구의 목적은 Chromo-helicase DNA-binding protein (CHD) 유전자의 Polymerase chain reaction (PCR) 증폭을 통한 연산 오계 발생중지란의 성비를 확인하기 위해 실시하였다.
Materials and Methods
공시 재료는 충남 논산시 연산 화악리 오계 농가에서 한 세대의 발생과정에서 획득한 총 153개의 발생중지란을 실험에 이용하였다. 획득된 발생중지란의 조직 25 mg 과 PBS (phosphate buffer saline) buffer 180 ㎕를 혼합하여 혈액을 묽게 희석한 뒤 PrimePrepTM Genomic DNA isolation kit (Genetbio, Korea)를 이용하여 Genomic DNA (gDNA)를 추출하였다. 추출된 gDNA는 Nanodrop 2000c 분광흡도계(Thermo Fisher Scientific, USA)를 이용하여 DNA의 농도 및 품질을 측정하였다. 측정된 농도를 바탕으로 각각의 gDNA를 3차 증류수를 첨가하여 25 ng/㎕의 농도로 균일하게 희석하였다. 준비된 gDNA는 실험 전까지 -20℃로 보관하였다.
Ensembl (https://asia.ensembl.org) 데이터베이스에 공개된 염기서열 정보를 획득하여 오픈소스 프로그램인 Clustal Omega (https://www.ebi.ac.uk/Tools/msa/clustalo/)를 이용하여 닭의 성별 구분이 가능한 염기서열 구역을 탐색하였다. 성염색체 Z 및 W 염색체의 데이터인 NC_006127.5 및 NC_006126.5에서 chromo-helicase DNA-binding protein (CHD) 유전자를 포함한 구간의 Alignment를 확인하여 Z 및 W 염색체에 차이를 나타내는 특이적인 구간을 선택하였고, 이 영역을 Primer3Plus (http://www.bioinformatics.nl/cgi-bin/primer3plus)로 타겟 유전자를 증폭하기 위한 primer를 디자인하였다. 이어서 NCBI (https://www.ncbi.nlm.nih.gov/)의 Primer-BLAST 프로그램을 이용하여 디자인된 primer의 특이성을 평가하여 목표한 염기서열의 증폭이 정확히 유도되었는지 확인하였다.
PCR 증폭을 위해 총 볼륨이 20 ㎕ 인 mixture를 제작하였다. Mixture는 각각 50 ng의 gDNA, multi-HS premix (Genetbio, Korea) 10 ㎕, forward 1, forward 2 및 reverse primer (10 pmol/㎕) 0.8 ㎕씩 혼합하였으며 잔여 볼륨은 3차 증류수를 이용하여 충당하였다. PCR 증폭 조건은 95°C로 10분간 pre-denaturation 후 95℃ 30초, 62℃ 30초, 72℃ 45초씩 denaturation, annealing, extension을 총 30 cycle 반복 후 72°C에서 10분간 final-extension을 수행하였다. PCR 증폭이 완료된 산물은 TAE buffer로 제작된 2.0% agarose gel을 이용하여 전기영동의 방법으로 유전자형을 확인하였다.
통계 분석은 R 프로그램의 내장 함수를 이용하여 chi-square test를 실시하였다. 기대값의 설정은 실험에 이용된 153마리의 개체를 1:1 비율로 나눈 값을 이용하였고, 관측값은 실제 관측된 결과값을 이용하였다.
Results & Discussion
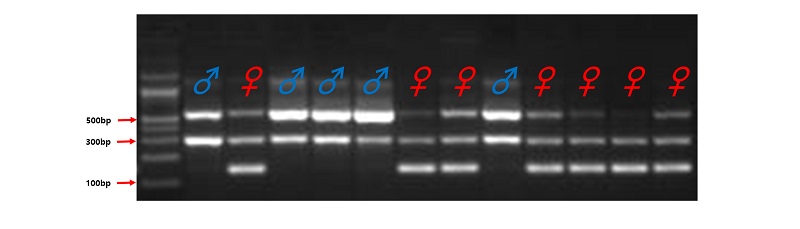
Alignment 결과 Z 염색체의 chromo-helicase DNA-binding protein (CHD) 유전자와 W 염색체의 CHD 유전자는 85.79%의 일치율을 보였다. PCR증폭 결과 밴드 양상에 따라 성별의 구분이 명확하게 이뤄지는 것을 확인 할 수 있었다(Fig. 1). 전체 153마리의 오계 발생중지란 개체의 성별을 확인한 결과 총 91마리가 암컷으로, 62마리가 수컷으로 각각 확인되었다. 수컷의 경우 ZZ 염색체를 갖기 때문에 568bp, 300bp의 두 가지의 밴드를 갖게 된다. 반면, ZW 염색체를 갖는 암컷의 경우 W 염색체를 포함하고 있기 때문에 568bp, 300bp, 143bp의 세 가지의 밴드 결과를 보여주었다. 이는 CHD 유전자의 12번 intron의 차이에 기인한 것으로 Z 염색체의 경우 intron의 크기가 511bp인데 반해 W 염색체는 86 bp로 염기서열의 크기 및 구성에서 차이를 나타낸다. 이때 Forward_2_primer의 경우 Z 염색체의 intron 내에 상보적인 primer로써, W 염색체의 서열과 상이하기 때문에 Z 염색체 내에서는 증폭을 유도하는 반면, W 염색체에서는 증폭을 유도할 수 없다. 따라서 Forward_2_primer에 의해 증폭된 300bp 크기의 밴드는 Z염색체와 W염색체와의 구분점을 나타내게 된다(Table 1).
PCR증폭을 통한 성별확인 결과의 유의성을 확인하고자 관측된 153마리의 오계 발생중지란 개체의 chi-square test를 실시하였다(Table 2). 일반적인 기대 성비인 76.5 : 76.5 (1:1) 암수 비율을 기대값으로 설정하고 측정된 성비인 91 : 62 (1:1.468) 암수 비율을 관측값으로 분석을 수행하였다. 그 결과 본 실험에서의 분석 결과 관측치의 성비가 기대 성비와 유의적인 차이가 존재하는 것을 확인하였다(p-value < 0.05). 따라서, 본 연구에서는 연산오계의 부화과정에서 얻은 발생중지란은 ZW의 암컷이 유의적으로 더 많은 것으로 확인되었으며, 이는 암컷 개체가 수컷 개체에 비해 상대적으로 높은 배아 사망률을 나타내고 있음을 의미한다. 이러한 발생중지란에서 나타난 편향된 성비의 원인은 크게 두 가지로 예상해 볼 수 있다. 첫 번째는 외부 환경적인 요인에 의한 성비의 차이이다. 산란계 집단에서의 성비 관련 연구에서도 본 실험의 결과와 유사하게 암컷 배아 사망률이 더 높게 보고되었다(Feng et al., 2006). 이것은 배아의 인큐베이션 환경에 따른 암수의 생리학적 저항성의 차이인 것으로 사료된다(Niu et al. 2001; Cook and Monaghan 2004). 또한 Ewen의 조류의 성비 관련 연구에 따르면 최적화 되지 않은 야생의 환경에서 일반적으로 암컷의 배아 사망률이 수컷보다 유의적으로 더 높게 나타난다고 보고되었다(Ewen et al., 2001). 또한 두 번째 원인으로는 반성치사유전에 의한 성비의 차이이다. 만약 Z 염색체에 치사 인자가 연관되었다고 가정할 경우 ZZ 염색체를 갖는 수컷에 비해 ZW 염색체를 갖는 암컷은 상대적으로 치사유전자의 발현빈도가 지극히 높게 나타날 수 있다. 하지만 아직까지 닭에서 발생하는 반성치사유전자는 보고된 바는 없지만 그 가능성을 열어두고 지속적인 모니터링을 통한 질병 발생의 초기 대응이 필요할 것으로 판단된다.
본 연구에서는 천연기념물 연산 오계에서 발견된 153마리의 발생중지란을 이용한 성염색체의 성별확인 결과 암수의 성비의 불균형을 확인하였고(p-value < 0.05), 이는 암컷 개체의 배아 사망률이 더 높게 나타난 결과를 확인할 수 있었다. 한국 고유의 유전자원이자 천연기념물인 연산 오계의 원활한 보존을 위해 추가적인 연구를 통하여 성비 불균형의 주요 원인을 다각도로 파악하고, 다양한 연구와 해결방법을 통해 이 문제를 개선해야할 필요성이 있을 것으로 사료된다