서 론
한우의 기원은 Bos primigenius에서 유래된 Bos tsurus가 중국 북부지역을 통하여 만주로 유입된 것으로 추정하고 일부는 인도를 거쳐 중국남부로 이동된 Bos Indicus와 혼혈되어 한반도 안에서 사육된 것으로 알려져 있다. 한우는 농경, 물자운반, 퇴비생산, 제사 및 고기생산을 위해 사육되다, 광복 이후 1954년 가축보호법 제정으로 소를 보호 증식하기 시작하였고 1963년 축산법 제정으로 한우 사육 목적이 고기생산으로 바뀌면서 개량과 증식에 많은 관심을 가지게 되었다
번식기술의 발달로 인공 수정이 보편화되면서, 1980년대 이전에는 축산진흥대회와 같은 품평회를 통한 외모가 우수한 씨수소를 선발하여 활용하였고, 1980년대 이후에는 당·후대검정결과를 동물개체모형(Animal Model)을 이용하여 추정된 육종가를 활용하여 씨수소 선발에 활용하고 있다(당창권, 2011).
현재 한우의 정액은 매년 66두의 후보씨수소를 선발하여 후대검정 공시 후 30두의 보증씨수소를 선발하여 농가에 보급하고 있으나, 적은 수의 씨수소 사용은 혈연관계가 있는 개체들이 교배 될 가능성 또한 증가되어 세대 당 근교 된 개체의 확률이 높아 지고(Weigel, 2001), 근교는 보통 근교퇴화로 알려져 있는 개체의 성장, 비유, 및 생존능력 감소를 가져오며, 유효집단크기가 작 아지면 개체의 유전적 다양성이 감소(FAO, 2008)시킨다고 알려져 있다.
본 연구에서는 한우육종농가에서 생성된 혈통 자료를 이용하여 한우 집단의 근교계수 및 혈통 분석을 통해 육종농가 집단의 유전적 다양성 정도를 알아보고 한우개량에 대한 기초자료로 활용하기 위하여 실시하였다
재료 및 방법
1.공시 재료
본 연구는 혈통의 정확도가 근교계수에 미치는 영향을 알아보기 위하여, 전체집단과 혈통이 정확한 친자감별을 마친 집단으 로 나누어 진행하였다. 한우개량사업소(농협중앙회)에서 제공한 1990년~2017년 3월까지의 98개 육종농가에서 사육되었던 전 체 개체자료 90,800두의 친자감별 유무 및 혈통자료, 후대검정우 36~62차 5,544두의 자료를 이용하였고, 중복개체 658두를 제외 한 95,686두였으며, 관련된 개체의 혈통정보를 (사)종축개량협회의 121,336건을 제공받아 진행되었다
친자감별을 마친 개체는 39,729두, 침자감별을 미실시한 개체는 51,071두로 친자감별을 마친 개체 중 육종농가 사업이전 3,032 두와 육종농가 사업이후 36,697두였다. 육종농가 개체와 후대검정우 개체를 합하여 45,273두였으나, 중복개체 428두 제거 후 총 44,845두를 본 연구의 대상 개체로 선정 분석하였으며, 아비의 혈통정보는 종축개량협회에서 제공한 자료를 사용하였으며, 총 혈통자료로 사용한 두수는 64,175두였다.
육종농가의 지역별 농가 두수는 강원도, 경기도, 경상남도, 경상북도, 전라남도, 전라북도, 제주도, 충청남도, 충청북도 각각 13 농가 12.346두, 11농가 8,211두, 9농가 9,550두, 16농가 13,706두, 16농가 13,414두, 16농가 20,126두, 2농가 1,053두, 16농가 4,590 두, 9농가 7,804두이였고, 육종농가 사업이전 사육개체는 43,048두, 육종농가 사업이후 47,752두이였다
2. 분석방법
1) 혈통지수(pi; pedigree index) 계산 및 표기방법
본 연구의 혈통 분석을 위하여 개체식별번호와 종축개량협회 혈통등록번호, KPN 명호를 통합하여 개체정보를 정리하였으며, 친자감별을 마친 집단의 개체정보는 육종농가 자료를 우선 사용하였다.
1) 혈통완성도
집단의 크기, 혈통완성도 및 육종계획 등은 개체의 근교계수에 영향을 미치며(Gutierez et al., 2003), 이 중에서도 혈통완성도 는 추정된 근교계수의 정확도에 영향을 끼친다. 본 연구에서 혈통완성도는 개체, 부모세대, 조부모세대, 그리고 증조부새대까지 총 4대에 걸쳐 조상을 알고 있는 비율로 나타내었으며, 평균혈통완성정도는 각 개체 별로 부, 모, 조부, 조모, 외조부, 외조모, 조 부의 부, 조부의 모, 조모의 부, 조모의 모, 외조부의 부, 외조부의 모, 외조모의 부 및 외조모의 모를 알고 있는 비율을 아래와 같 은 공식으로 계산하였다.
혈통완성도(%) = (알고 있는 선조의 개수 ÷ 14) × 100
2) 근교계수
근교계수의 정의는 공통조상이 하나 이상인 경우도 각 공통조상을 통하여 전수동일이 될 확률을 합해줌으로써 모든 공통조상 으로 연결되는 모든 독립적인 경로를 통한 전수동일의 확률로 이를 수식으로 나타내면 다음과 같다(김 et al0. 025).
여기서, FX는 개체의 근교계수이고, n은 부친으로부터 공통조상에 이르는 세대이며, n'는 모친으로부터 공통조상에 이르는 세 대 FA는 공통조상의 근교계수이다. 개체별 근교계수 계산을 위하여 김 et al. (2012)에 의해 이용된 포트란 코드를 사용하였으며, 프로그램은 GNU Fortran Compiler로 컴파일하였다. 평균 근교계수는 Meuwissen & Luo (1992)의 알고리듬을 이용하여 계산 하였으며, 이를 수식으로 나타내면 다음과 같다
여기서, △F는 근교계수의 비율이며, n은 세대수, △Ft는 (Ft-Ft-1)/(1-Ft-1)이고, t는 세대 수에 태어난 개체들의 평균 근교계수 이다.
3) 세대간격
세대간격은 아비에서 수송아지, 아비에서 암송아지, 어미에서 수송아지 그리고 어미에서 암송아지 등 4가지 경로에 의해 계산 되어진다. 그 수식은 다음과 같다.
여기서, L은 세대간격이고, Lff는 아비의 생년월일에서 수송아지의 생년월일을 뺀 값들의 평균, Lfm은 아비의 생년월일에서 암 송아지의 생년월일을 뺀 값들의 평균, Lmf은 어미의 생년월일에서 수송아지의 생년월일을 뺀 값들의 평균이며, Lmm은 어비의 생 년월일에서 암송아지의 생년월일을 뺀 값들의 평균이다.
4) 유효집단 크기
유효집단의 크기는 아래와 같은 수식에 의해 계산하였다(Falconer and Mackay, 1996).
여기서, Ne는 유효집단 크기이고, L은 세대간격, △F는 평균 세대간 근교계수 병화량이며, 세대 간 이형접합체의 상대적 감소 크기로 아래와 같이 계산된다.
여기서, Ft는 t세대의 근교비율, 그리고 Ft-1은 t-1세대의 근교비율이다
결과 및 고찰
1. 혈통완성도 및 근교계수
1) 혈통완성도
근교계수에 영향을 주는 요인은 혈통의 정확도로 근교계수가 높은 개체임에도 혈통정보가 없으면 근교계수는 0으로 추정된 다. 집단의 혈통완성도는 개체 간 혈연계수에 연관되어 있어 근교계수에 영향을 미친다(Gutierez et al., 2003). 따라서 본 분석에 앞서 대상 두수에 대한 4대 혈통까지 추적하여 혈통완성도를 조사하였다
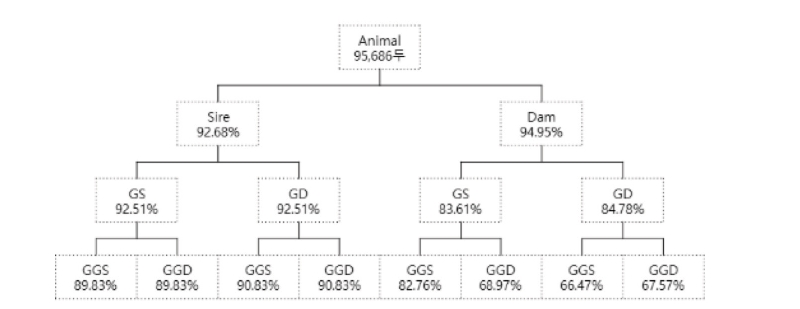
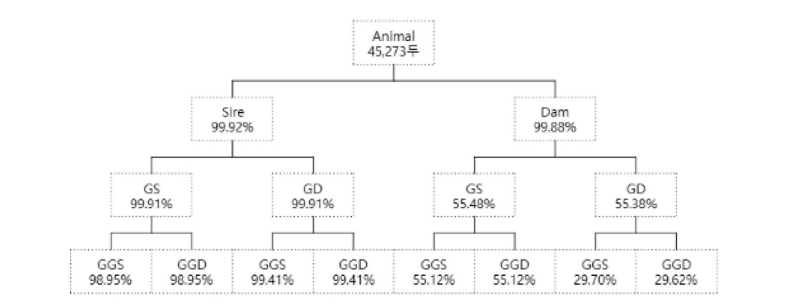
육종농가와 후대검정우의 전체혈통완성도의 결과는 Fig 2와 같으며, 친자감별을 마친 개체와 후대검정우의 혈통완성도의 결과는 Fig 3으로 나타내었다.
전체집단의 경우 95,686두 부를 아는 비율은 92.68%, 모를 아는 비율은 94.95%였으며, 조부모를 아는 비율은 92.51%, 외조부모를 아는 비율은 84.20%, 증조부모세대를 아는 경우는 66.47~90.83% 나타났으며, 총 84.87%로나 타났다.
친자감별을 마친 집단의 경우 45,273두 부를 아는 비율은 99.92%, 모를 아는 비율은 99.88%였으며, 조부모를 아는 비율은 99.91%, 외조부모를 아는 비율은 55.43%, 증조부모세대를 아는 경우는 29.62~99.41% 나타났으며 총, 76.72%로 나타났다
두 집단간 부모 세대에서는 친자감별을 마친 집단이 6.06% 높게 추정되었으며, 조부세대와 증조부세대에서는 각각 10.68%, 10.10%로 전체집단이 높은 것으로 나타났다. 세대를 모두 알고 있는 경우는 전체집단에서 63.856%, 친자감별을 마친 집단은 28.422%으로 나타났다.
원정일. (2015)은 우리나라 Holstein 검정우 집단의 혈통완성도는 부모세대를 아는 경우는 69%, 조부모세대를 모두 아는 경 우는 42%, 증조부모세대를 아는 경우는 22~66%로 보고하였으며, 14개의 선조를 모두 아는 비율(100%)은 19.06%로 본 연구의 한우의 집단과는 차이가 있었다
조 et al. (2014)은 한우암소검정사업 참여여개체 중 혈통완성도가 가장 높은 지역은 강원도로 부모를 모두 아는 암소의 비율 을 90%, 조부모를 모두 아는 경우 90%, 외조부를 모두 아는 경우 67% 증조부모를 아는 경우는 39~90%로 보고하였으며, 강원 도가 평균 80.0%로 가장 높게 평가 된 것으로 보고 하여, 본 연구의 전체집단보다는 낮은 혈통완성도를 친자감별을 마친 집단보 다는 높게 나타났다.
선조를 알고 개수의 비율은 Table 2와 같으며, 친자감별을 마친 집단의 혈통완성도에서 8개의 선조를 알고 있는 비율이 가장 높았으며 전체집단은 14개 선조를 모두 아는 비율이 가장 높게 확인되는데, 이는 친자감별을 마친 집단에서의 선조가 친자감별 이 되지 않아 제외 됐을 것으로 사료된다.
|
Table 2. Number and percentage of animals having complete pedigree by generation in whole and subset group 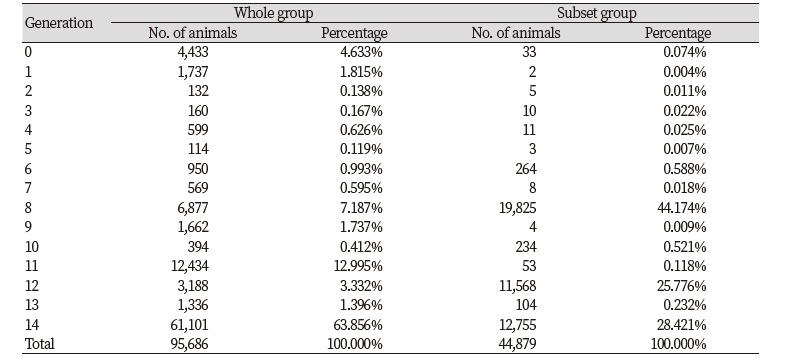
|
2) 근교계수
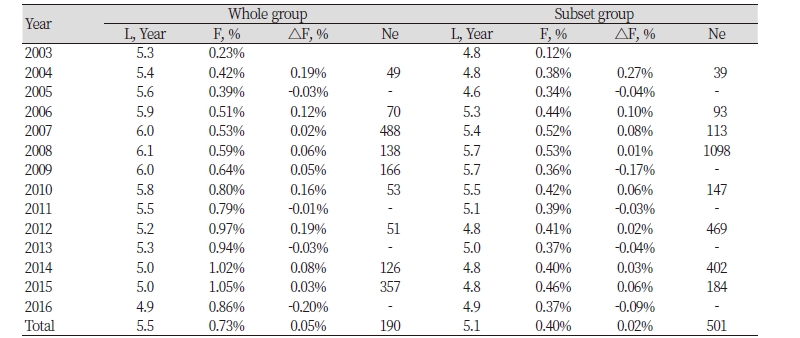
본 연구에서 공시된 육종농가, 후대검정우 개체의 근친 비율은 Table 3에 표시하였다. 전체집단과 친자감별을 마친 집단 모두 근친된 개체의 비율은 년도가 지날수록 증가하고 있으며 2016년 근친인 개체의 비율은 전체집단, 친자감별을 마친집 단에서 각 각 95.50%와 57.84%것으로 나타났으며, 평균 근교계수는 각각 0.73%와 0.40%이였고, 전체집단에서는 2015년 근교계수는 1.05%로 가장 높았으며, 친자감별을 마친 집단에서는 2008년 근교계수는 0.53%로 가장 높게 나타났는데, 이는 친자감별이 2008 년부터 시행되면서 농가의 인식이 많이 바뀐 결과로 보이며, 2009년 이후 0.36~0.46%로 평균과 유사한 근교계수를 보였다.
|
Table 3. Number of animals, number and percentage of inbred animals and average inbreeding coefficient by birth year in whole and subset group 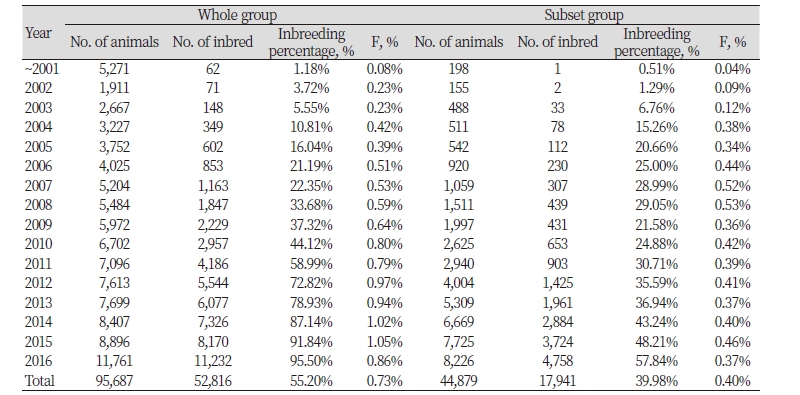
|
Melka et al. (2013)은 2003년~2007년까지 분석한 캐나다 젖소 5품종Ayrshire, Brown Swiss, Canadienne, Guernsey 및 Milking Shorthorn에 대해 근교계수를 각각 0.93%, 1.07%, 1.26% 1.09% 및 0.80% 보고하여 본연구보다 다소 높게 나왔으며,황 et al. (2013)은 1995~2007년까지 수집하여온 한우개량육성농가사업의 암소 혈통자료 분석에서 2007년 근교계수를 0.30%로, 근친의 개체 수의 비율은 1998년 0.2%에서 2007년 11.4%로 증가하고 있다고 보고하여, 본 연구가 높게 나타났다.
당 et al. (2011)은 1983~2008년까지 1,128두에 대한 보증 및 후보씨수소의 분석결과 평균 근교계수는 2007년 0.71%, 2008년 0.97%로 보고하였으며, 근친비율은 2007년 12.77%, 2008년 42.86%로 본 연구의 2007년, 2008년 근교계수는 0.524%, 0.532% 보다 높게 보고하였으며, 근친비율은 동일년 28.99%, 29.05%로 2007년보다는 낮게, 2008년 보다는높 게 보고하였다.
조 et al. (2014)은 2000~2012년의 한우암소검정서업 참여여개체 지역별 근교계수의 평균을 강원도 0.63%, 경기도 0.46%, 경 상남도 0.49%, 경상북도 0.48%, 전라남도 0.71%, 전라북도 0.51%, 충청남도 0.40%, 충청북도 0.61% 제주도 0.55%로 보고하였 다.
2. 세대간격 및 유효집단 크기
Table 4는 연도별 전체집단과 친자감별을 마친 집단의 세대간격으로, 전체집단에서의 세대간격은 아비와 수송아지에서 5.9~8.7년, 어미와 수송아지는 1.8~4.3년,아비와 암송아지에서 5.9~8.3년, 어미와 암송아지는 3.5~4.3년으로 나타났으며 평균의 경우 각각 7.1년, 3.7년, 7.2년, 3.9년이며, 아비의 평균은 7.2년, 어미의 평균은 3.8년, 수송아지의 평균은 5.4년, 암송아지의 평균 은 5.6년이였으며, 세대간격은 5.5년으로 나타났다.
|
Table 4. Generation intervals across 4 selection pathways and the weighted average for Hanwoo population 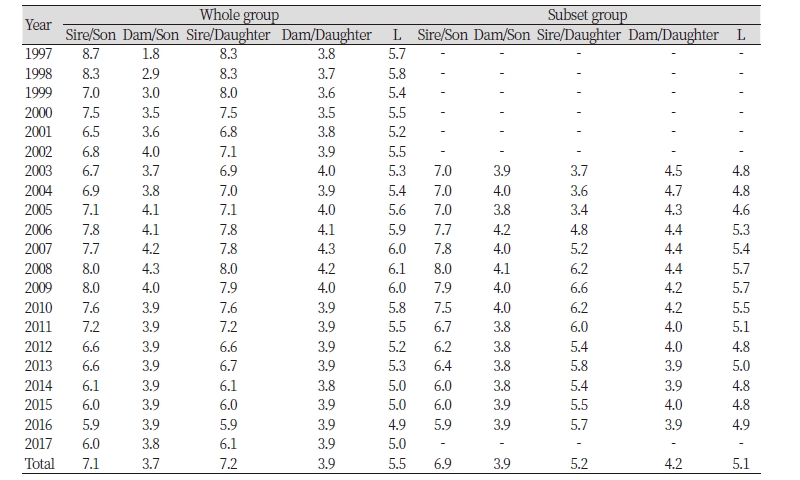
|
친자감별을 마친 집단에서는 각각 5.9~8.0년, 3.8~4.2년, 3.4~6.2년, 3.7~4.7년으로 평균 각각 6.9년, 3.9년, 5.2년, 5.1년으로 나 타났으며, 아비의 평균은 6.1년, 어미의 평균은 4.1년, 수송아지인 경우 평균 5.4년, 암송아인 경우 평균 4.7년이였으며, 세대간격 은 5.1년으로 나타났다
한우는 종모우를 약 5년간 당대, 후대검정을 통해 선발되며 평균 2년간 정액이 사용되는 것으로 보아 아비의 세대간 격은 약 7 년으로 추정되며, 7.2년과 6.1년으로 이 친자감별을 마친 집단은 약1년 짧은 것으로 나타났으며, 어미의 경우 약 24개월에 초산 을 하고 번식간격이 약 12개월인 것으로 보아 두집단 모두 약 3산차로 추정된다. 또한 친자감별을 마친 집단의 경우 수송아지의 아비와 암송아지의 아비에서 약 1,7개월의 차이가 있는 것으로 후대검정우 자료의 영향으로 판단되며, 전체집단에서는 집단의 크기가 큰 관계로 상쇠된 것으로 사료된다.
당 et al. (2011)은 아비에서 수송아지의 세대간격 범위를 2.97년~8.54년 어미에서 수송아지의 세대간격 범위를 1.79~6.96년으 로 평균 각각 6.95년, 5.17년 세대간격의 범위를 2.38~7.23년으로 평균 6.06년으로 추정하였고, 조 et al. (2014)은 아비에서 암송 아지, 어미에서 암송아지의 각각 세대간격을 7.63년, 4.11년으로 평균 5.87년으로 보고하여, 본 연구와 다소 차이를 보였다.
유효집단의 크기는 Table 5와 같으며, 근교계수의 증감에 의해 유효집단의 크기도 증감의 폭이 크게 나타났으며, 전체집단에 서는 평균 190두, 친자감평을 마친 집단에서는 평균 502두로 당 et al. (2011)의 연구에서의 유효집단 크기는 46~220두, Martinez et al. (2008) 콜롬비아 3개 품종에 대해 27~143두로, Maiwashe et al. (2006) 아프리카 4개 품종에 대해 108~168두, Nomura et al. (2001) 일본 흑모화우의 14~52두 보고하였는데, 본 연구 결과와 다소 차이가 있는 것으로 나타났다.