포유동물 후각체계의 구성과 후각 수용체
후각이란 공기중의 다양한 냄새 분자를 인식하는 과정으로 먹이를 식별하거나 포식자를 회피하는 등, 생존과 밀접한 표현형이다. 그러므로 비교적 후각 의존도가 높은 육상동물의 경우 양성선택(positive selection)을 통한 후각 수용체 유전자군의 확장이 발생한 반면, 조류 및 수생동물의 경우 음성선택(negative selection)에 의한 유전자군의 축소가 발생하였음을 볼 수 있다(Niimura and Nei, 2007). 또한 포유류, 파충류, 초파리 등의 진화적 상관관계가 비교적 먼 종들을 비교한 결과, 비강, 혀, 더듬이 등의 후각인지 기관의 해부학적 차이에도 불구하고 OSN에서 시작되는 냄새분자(odorants)의 인식 기전에 대한 유사성을 볼 수 있다(Firestein, 2001).
포유류의 OSN은 해부학적으로 비강 상부의 비갑개(turbinates)로 불리는 연골성 조직에 위치하며, 비갑개 외곽의 점액층에서 후각 수용체(olfactory receptor:OR) 와 odorants간의 구조 특이적 결합현상이 일어난다(Mombaerts, 1999). OSN은 신경세포 중에서 유일하게 외부로 노출 되어있는 세포로써 동물의 후각 인지력은 OSN이 발현하는 후각 수용체의 다양성에 의해 크게 영향을 받는다(Touhara, 2002; Dryer, 2000). 다양한 odorants를 식별하기위해 후각수용체의 다양성은 필수적이고 따라서 후각수용체 유전자 시스템은 게놈 내에서 동일한 기능의 수행을 위하여 가장 많은 수의 유전자를 가지는 시스템으로 진화하였다(Firestein, 2001).
후각수용체와 결합한 odorants는 OSN에 활동기 전위(action potential) 를 발생시키며 OSN의 축삭돌기(axon)을 따라 중추신경계로 전달된다. 여러 OSN을 통해 전달된 신호는 대뇌 기저부의 후각망울(olfactory bulb)의 사구체층(layer of glomeruli)에서 통합되고 통합된 후각 신호는 OSN과 시냅스를 형성하고 있는 주출력 세포(main output neuron)인 승모세포(mitral cell)에 의해 대뇌로 전달되어 최종적으로 ‘냄새’로 인식된다(Firestein, 2001). 1991년 냄새분자와 결합하는 동물의 OR유전자가 처음 발견되었으며 (Buck and Axel, 1991), 이러한 결과를 바탕으로OR의 특징 및 다양한 동물 종의 OR유전자 시스템의 분석이 진행되었다(Zhang and Firestein, 2002; Malnic et al., 2004; Lee et al., 2012; Lee et al., 2013).
현재까지 밝혀진 OR의 유전자 발현상의 특징으로는 하나의 OSN은 한 종류의 OR 유전자만을 발현하며, OR은 G protein coupled receptor(GPCR) 패밀리에 속하는 인트론이 없는 단일엑손 유전자이며 게놈구성의2%를 차지할 만큼 거대한 유전자군이라는 것이다 (Firestein, 2001). OR의 정확한 3D 구조는 아직 명확하지 않으나 광신호 전달에 중요한 GPCR인 로돕신과 비슷한 구조를 가지는 것으로 예측된다(Palczewski et al., 2000).
후각수용체의 유전자 레퍼토리(repertoire)
후각수용체는 물고기의 후각수용체와 유사한 class I타입과 육상동물에 제한적으로 존재하는 class II타입의 2가지 하위분류군(subfamily)로 구분된다. Class I OR은 수용성 냄새분자를 감지하고 class II OR은 공기중의 휘발성 냄새분자를 감지하는데 특화되어 있다(Freitag et al., 1995). 이러한 특성에 따라 금붕어(Carassius auratus)는 class I 후각 수용체 유전자만 가지고 있으며 고래의 일종인 줄무늬 돌고래(Stenella coeruleoalba) 에서는 class II 후각수용체 유전자가 전부 기능을 상실한 비기능성 위유전자화(pseudogenization) 되어 있다(Freitag et al., 1998). 이러한 결과는 후각수용체 유전자는 환경의 변화에 따라 빠르게 변화되고 있다는 것을 나타낸다.
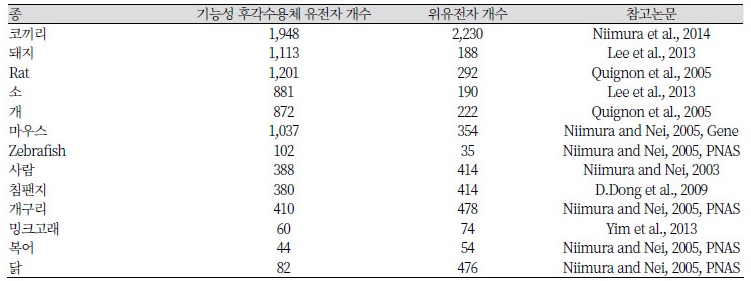
초기의 OR 유전자 분석은 DNA hybridization 방법을 이용하여 생쥐, 쥐, 사람 및 물고기에 대한 유전자 분석을 시도하였다(Buck, 1996; Mombaerts, 1999). 그러나 차세대 염기서열분석 기술의 발전에 따라 다양한 종의 게놈분석 자료를 알 수 있게 되었고, 이를 바탕으로 OR 유전자의 annotation이 진행되었다. 그 결과 쥐(Zhang and Firestein, 2002), 사람(Malnic et al., 2004), 개(Olender et al., 2003), 돼지(Lee et al., 2012), 소(Lee et al., 2013) 밍크고래(Yim et al., 2013) 등 다양한 척추동물의 후각수용체 유전자가 동정되었다(표1). 이러한 연구를 통해 후각수용체 유전자의 개수는 종에 따라 100개 이하에서 1,000개 이상으로 큰 차이를 보인다는 것과(Firestein, 2001), 기능을 가지는 기능유전자(functional gene)와 기능을 상실한 위유전자(pseudogene)의 비율 또한 종 사이에 현저한 차이가 존재한다는 것을 알 수 있었다(표 1).
기능성 후각수용체 유전자와 위유전자의 비율은 동물의 후각 감지 능력을 평가하는 지표가 될 수 있다. 예를 들어 후각이 매우 발달한 동물인 소, 개, 마우스, rat, 돼지 코끼리의 경우 기능성 후각 수용체 유전자의 개수가 800개 이상으로 이러한 동물의 특성과 일치 한다. 최근 밝혀진 코끼리의 후각 수용체 유전자수는 4000개 이상으로, 코끼리의 후각 의존성이 다른 종들보다 월등히 높다는 것을 알 수 있다. 실제로 코끼리의 사회적 관계와 후각 간의 밀접한 관계 (Langbauer, 2000), 또는 이들의 해부학적으로 발달된 후각 기관의 존재(Shoshani et al., 2006) 등은 코끼리의 OR 유전자 확장현상을 설명하고 있다. 반면 밍크고래를 비롯한 수생동물의 경우 확인된 OR 유전자 수가 134개로 상대적으로 적었으며(Yim et al., 2013) 특히 고래목 동물의 위유전자의 비율은 50% 이상으로 다른 수생 동물보다 상대적으로 높게 나타났다(Kishida et al., 2007).
후각수용체 단백질의 구조 및 서열 다형성
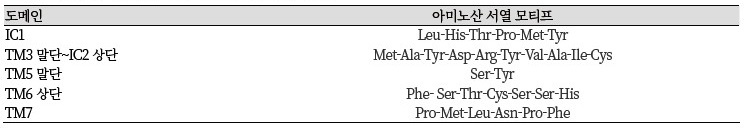
후각수용체는 7개의 알파-나선구조의 막관통(transmembrane:TM) 영역과 이들을 연결하는 intracellular & extracellular loop으로 구성되어 있다(Mombaerts, 1999). 척추동물의 후각 수용체는 다른 GPCR과는 다른 아미노산 모티프를 가지며(표2.)(Mombaerts, 1999; Lee et al., 2012; Lee et al., 2013; Yim et al., 2013), 이를 통하여 게놈 수준의 후각수용체 발굴 전략으로 이용될 수 있다.
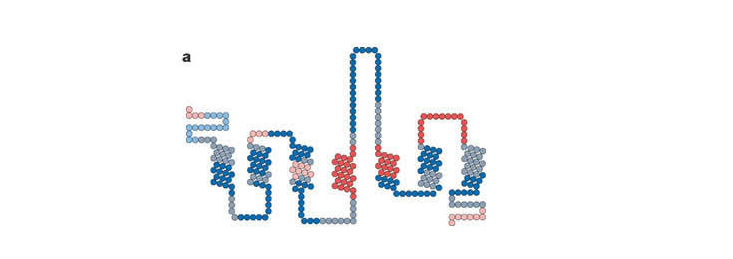
OR단백질의 아미노산 서열을 분석한 결과, OR의 7 TM 영역 중 TM 3, 4 및5에서 아미노산의 변이가 비교적 높게 나타났으며(그림 1.), 이 지역은 다양한 냄새 분자의 구조적 특징을 인식하고 식별하는 ‘결합 포켓’을 형성한다(Katada et al., 2005). 또한 아미노산 잔기 등의 구조적 특성을 공유하는 OR의 경우 상이한 OR이 동일한 odorants를 인식하여 후각 신호를 전달할 수도 있다(Kajiya et al., 2001).
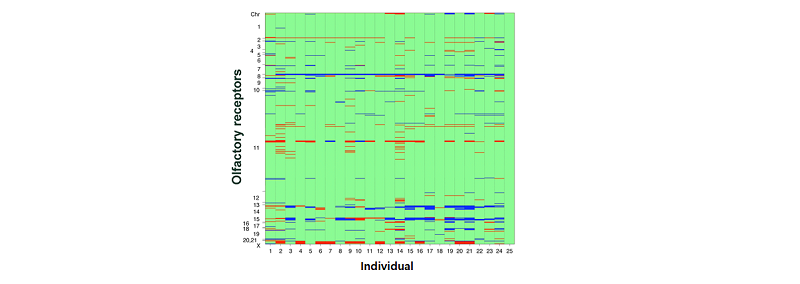
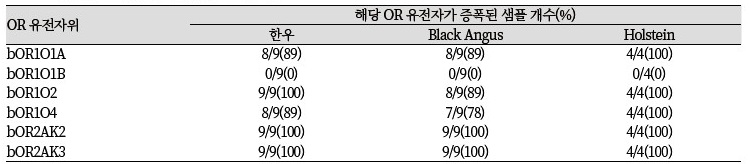
후각수용체의 기능을 파악하기 위해서는 OR다형성에 대한 정보를 파악하는 것이 매우 중요하다. 동물의 후각수용체 다형성에 대한 연구결과에 따른 공통적인 특징은 동일 종의 동일 후각수용체 유전자임에도 불구하고 copy number variation(CNV)나 단일 염기 다형성(single nucleotide polymorphism, SNP) 등에 다양한 차이를 보인다는 것이다. 예를 들어 사람 25명으로부터 851개의 후각수용체에 대한 CNV를 관찰한 결과 25명 모두에서 다양한 CNV 패턴이 관찰되었다(그림 2.)(Hasin et al., 2008). 또한 사람의 후각수용체 유전자좌의 기능에 영향을 주는 CNV나 SNP가 전체 후각수용체 유전자 레퍼토리의 15~20%를 차지할 정도로 높은 빈도를 나타냈다(Waszak et al., 2010). 한우, Black Angus, Holstein의 3품종에 대해 10가지 후각수용체 유전자의 카피 수를 PCR 증폭을 통해 조사한 연구에서는 3개의 후각 수용체 유전자 (bOR1O2, bOR1O4 and bOR9M7) 가 품종에 따라 CNV를 나타내었다(표3.)(Lee et al., 2013). OR 다형성과 관련된 또 다른 연구(Robin et al., 2009)에서는 6 품종의 48마리 개를 대상으로 다형성을 조사하였는데, 그 결과 사람과 마찬가지로 매우 많은 SNP가 관찰되었다. 주목할 만한 점은 이들의 SNP는 사람처럼 품종과 개체별로 다양하였으나 다형성이 일어나는 유전자위는 품종에 관계없이 일정한 패턴으로 나타났다는 것과 후각수용체 유전자에 따라 SNP의 개수가 0~22개로 다양성이 매우 높았다는 것이다. 예를 들어German Shepherd에서는 총 926개의SNP가 존재했고, 페키니즈에서는 총 SNP개수가 628개로 비교적 적었다. 셰퍼드가 훌륭한 후각을 바탕으로 마약탐지견 등으로 사육되는 반면 페키니즈는 그렇지 못한 것으로 볼 때, 집단내의 후각수용체 다형성과 후각능력의 연관성에 대한 분석은 매우 흥미로울 것으로 판단된다.
후각수용체의 진화 및 종 특이성
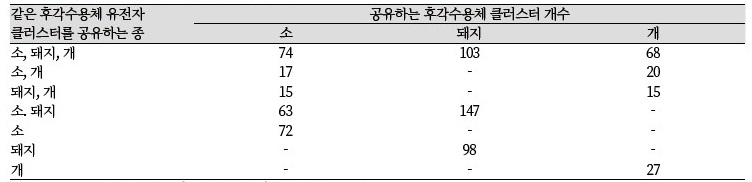
수용성 냄새분자를 감지하는 class I 후각수용체는 해양생물의 주 OR 이며 반대로 공기중의 휘발성 냄새분자를 감지하는 class II 수용체는 육상동물의 주 OR 이다. 이처럼 후각수용체의 진화는 생물이 살아가는 환경과 밀접한 연관을 가지고 진화하였다(Hayden et al., 2010). 생물의 진화는 DNA 염기서열의 변화 및 이에 따른 아미노산 서열 변화에 따른 단백질의 기능변화에 의해 발생한다. 5 종의 포유류(소, 돼지, 사람, 쥐, 개)의 전체 OR 유전자를 비교한 결과 우제류인 소와 돼지가 공통적으로 공유하는 OR 유전자의 수가 소와 개, 또는 돼지와 개가 공유하는 후각수용체 유전자 숫자 보다 훨씬 많게 나타났다(표 4.)(Lee et al., 2013). 즉, 소와 돼지에서 공통적으로 인식될 수 있는 냄새 분자가 개와 소, 또는 개와 돼지가 공통적으로 인식할 수 있는 냄새 분자보다 훨씬 많다는 이야기로 해석이 가능하다. 이러한 결과는 소와 돼지의 진화적 근접성에 의한 것일 수도 있지만 풀을 뜯거나 땅을 헤집어 먹이를 찾는 소나 돼지에 필요한 후각 감각이 개들에게 필요한 후각 감각보다 더욱 비슷하게 연관되어 있기 때문에 나타나는 현상일 수도 있다. 또한 소에서만 특이적으로 발현되는 후각수용체는 72개이고 돼지의 경우는 98개였다. 이는 개에서 특이적으로 발현되는 후각수용체 개수 27개보다 훨씬 많다(Lee et al., 2013). 따라서 후각수용체 유전자는 생태학적 필요성에 따라 다양한 개수와 양상으로 진화하였다는 것을 알 수 있다.
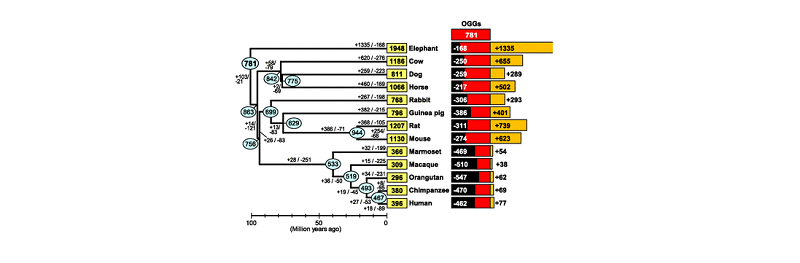
후각수용체 유전자의 개수 변화는 진화 과정에서 빈번하게 일어나는 현상이다(그림 3.)(Niimura et al., 2014). 그림 3의 결과처럼 각 계통에서 수백 개의 OR유전자가 손실되거나 새로이 생겨났다. 따라서 비슷한 수의 후각수용체 개수를 가졌다고 하더라도 실제 이들의 후각수용체 유전자 양상은 매우 다를 수 있다. 예를 들어 개와 기니피그의 경우 비슷한 개수의 후각수용체 개수를 가졌지만 이들의 51% 이하만이 공통적으로 공유되는 후각수용체였다. 이와 같이 후각수용체의 진화는 끊임없이 이어지는 자연선택의 결과물이다.
후성 유전적 기작에 의한 후각수용체 발현의 조절
동물들이 다양한 odorant를 인식하여 특정 냄새를 인지하는 것은 후각신경세포(OSN) 가 발현하는 후각 수용체의 다양성에 기인한다. 그러나 후각 수용체의 발현과 관련한 정확한 기전은 아직 명확하지 않다. 많은 수의 OR 유전자는 유전자 발현과 관련한 조절 기전을 필요로 한다. 특히 OR의 경우 멘델 양식을 따른 유전자발현 패턴과는 달리 하나의 후각신경세포는 오직 한 개의 OR 유전좌위 만을 발현하는 조절된 발현 양상을 나타낸다(Firestein, 2001). 따라서 발현되는 하나의 후각 수용체 유전자 외에 다른 후각 수용체 유전자 전체에 대한 발현을 억제하는 조절 기전이 반드시 필요하다. 이처럼 후각 수용체의 발현 조절은 후각 뿐만 아니라 유전자의 조절기전에 대한 이해를 위해서도 반드시 연구되어야 할 영역이다.
최근 연구결과에 의하면 후각 수용체 유전자의 발현 억제(silencing) 현상은 히스톤 H3의 9번 리신의 trimethylation(H3K9me3)과 히스톤 H4의 20번 리신의 trimethylation(H4K20me3)이 관여하는 것으로 분석되었다. 그리고 히스톤 탈메틸화에 의해 특정 OR 대립유전자의 H3K9me3와 H4K20me3가 탈메틸화 되면 해당 OR 유전자에 대해서만 전사가 활성화되어 후각 수용체 단백질을 발현하게 된다. 발현된 OR은 다른 후각 수용체 대립유전자의 추가적인 발현을 억제하고, 발현이 활성화된 OR 유전자의 지속적인 발현을 위한 피드백 신호를 유도한다 (Magkalra et al., 2011). 즉 후각 수용체의 발현은 특정 후각 수용체에 대한 발현 신호에 의해 이루어지는 것이 아니라 발현되지 않을 수많은 OR 유전자의 발현을 억제하는 방식으로 이루어지는 것이다.
그러나 매우 드물지만 후각 신경이 발현하는 후각 수용체의 종류가 후각신경세포의 일생 동안 일정한 것이 아니라 변화되는 경우도 관찰되었다(Shykind et al., 2004). 후각수용체 유전자의 탈메틸화 관련된 연구에 의하면 히스톤 탈메틸화 효소인 LSD1과 Adcy3(Adenylyl cyclase 3)의 발현 또한 OR 유전자의 발현에 중요한 것으로 밝혀졌다(Lyons et al., 2013). LSD1은 OR 유전자의 탈메틸화에 의한 OR 발현을 유도하며 Adcy3은 LSD1의 발현 억제를 통한 OSN의 분화를 촉진하는 것으로 보고되었다.
가축의 후각수용체 연구
가축의 OR 연구는 비강 내에 존재하는 OSN의 해부학적 분포 (Firestein, 2001) 및 OR 총유전자수(repertoire)에 대하여 돼지와 소, 개에서 각각 보고되었다 (표1). 그러나 개체 간의 OR 유전자 다형성의 비교, 정소를 포함한 비강이외의 조직에서의 OR 유전자 발현 (Parmentier et al., 1992; Vanderhaeghen et al., 1993; Vanderhaeghen et al., 1997), OR 수용체 다형성과 odorant 특이성 등에 대한 연구는 미미한 수준이다. 가축의 행동학적 특징과 CNV를 포함한 유전적 다형성과의 상관관계 분석은 후각과 관련한 가축의 특성 이해를 위한 흥미로운 연구가 될 것으로 판단된다. 그러나 가축의 후각 신경세포와 관련한 연구는 OSN이 비강내의 뼈 조직 내에 존재하는 해부학적 구조 및 유전자 발현의 후성 유전학적 특이성, 많은 OR 유전자수 및 유전자 서열의 유사성, genome assembly의 개선 필요성 등의 어려움에 대한 극복이 필요하다.
고등동물은 왜 후각에 이토록 많은 투자를 하도록 진화하였는가? 시각이 발달함에 따라 기능성 후각수용체의 개수가 감소한 인간의 경우도 무려 388개의 기능성 후각수용체 유전자가 존재한다(Niimura and Nei, 2003). 하나의 후각 수용체가 냄새분자의 농도에 상관없이 냄새 분자와 결합 또는 결합하지 않음의 2가지 단계로만 반응한다고 하더라도 구분 가능한 냄새의 수는 2388가지에 이른다. 이러한 후각체계의 엄청난 다형성과 복잡성 때문에 후각 체계에 대한 연구는 매우 흥미롭지만, 동시에 다양한 수용체의 상호작용을 연구할 수 있는 통합적인 연구 방법이 필요한 시점이다.