Introduction
돼지 육종은 지난 30년동안 animal model을 이용하여 이전보다 획기적이고 체계적인 분석을 이루었고, 이를 통해 돼지의 개량에 크나큰 성과를 이루었다. 이제 새로운 기술, 유전체 선발(genomic selection) 의 도입이 현실화되면서, 돼지뿐 아니라 모든 농업에 이용되는 종(種)들에 대한 선발이 진일보하고 있다. 이러한 유전체 선발은 지난 30년 동안 발생한 통계 유전학과 분자 유전학의 성과를 성공적으로 통합하는데서 비롯한다. 현재 세계적인 돼지 육종 회사들은 유전체 선발의 가능성을 확인하고 현실에 맞게 적용하는 노력을 기울이고 있다. 더불어 시대의 흐름과 고객의 요구에 맞게 육종 목표를 설정하고, 선발 지수에 반영하고 있다. 이 글에서는 육종 회사들의 육종 목표를 살펴보는 동시에 새로운 기술이 어디까지 발전되었는지, 현실 적용에 문제점은 무엇인지 파악하며, 돼지 육종 연구에서 일어나는 새로운 연구들은 무엇인지에 대해서도 언급해 보고자 한다.
Breeding Object
세계의 돼지 육종 회사는 2000년 이후 큰 회사들의 인수와 합병이 심심치 않게 이어져왔다. 2005년에는 Genus가 PIC를 인수하였고, 2008년에는 Gepork와 Hermitage Genetics가 협업 협약을 맺은바 있으며, 2014년 6월 Topigs International과 Norsvin International AS 가 한 회사Topigs Norsvin으로 합병된 바 있다. Topigs Norsvin는 이 합병으로 Hypor를 제치고, PIC에 이어 세계에서 두번째로 큰 돼지 육종 회사가 되었다. 각 회사의 육종 목표는 시대의 흐름과 고객의 요구에 발맞춰 조금씩 달라지고 있는데, 전체적인 흐름은 큰 테두리 안에서 사료 효율에 초점이 맞춰지고 있는 양상이다. 또한, 대부분의 육종회사들이 선발의 정확도를 극대화하기 위해, 전통적인 선발 방법으로부터 유전체 선발 방법을 적용하고 있거나, 적용을 심각하게 고려하고 있는 모습이다.
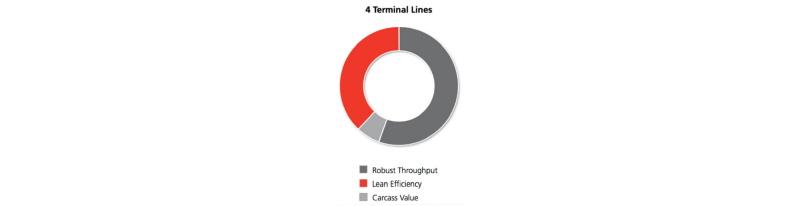
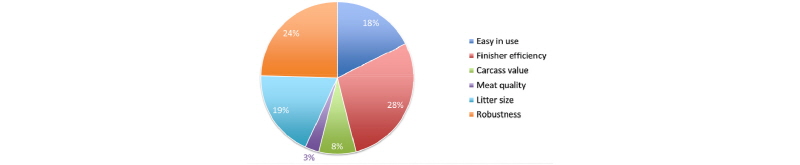
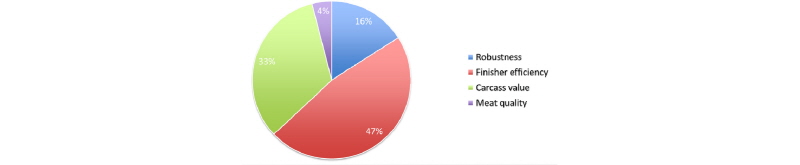
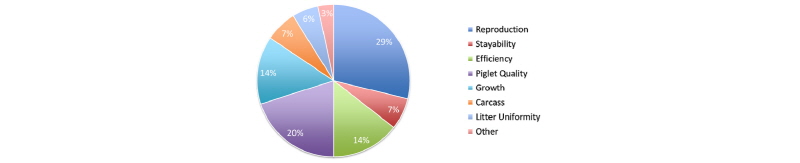
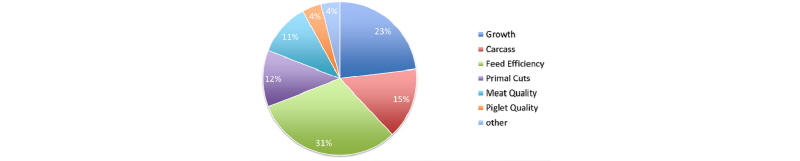
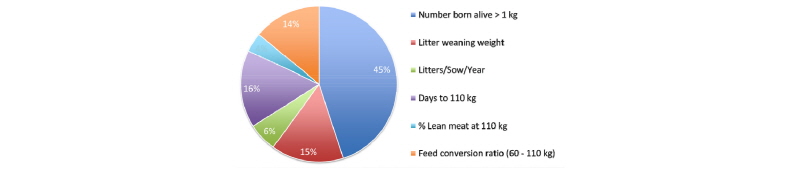
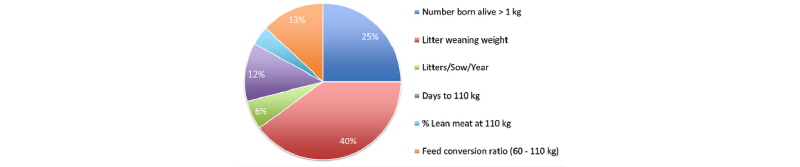
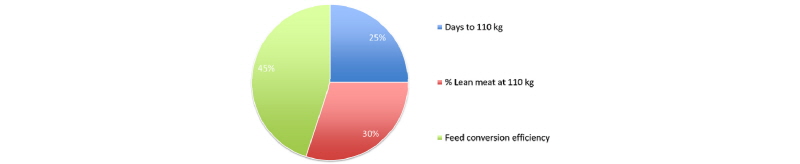
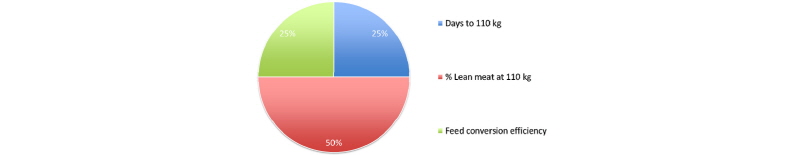
선발 방법은 최신의 기술을 도입하지만 육종 목표는 여전히 고민의 대상이다. PIC는 강건성, 사료 효율, 도체 가치 등의 세가지 부분에 초점을 맞추어 선발을 하고 있다(Fig. 1). Topigs Norsvin은 비육돈 효율과 도체가치, 강건성 등에 더 가중을 하고 있는데, 모계 라인의 경우 비육돈 효율(28%)과 강건성(24%), 산자수(19%) 등의 순으로 선발 지수에 가중을 두고 있으며(Fig. 2), 부계 라인 역시 비육돈 효율(47%)과 도체 가치(33%), 강건성(16%) 등을 중요한 형질로 여기고 있다(Fig. 3). Hypor의 경우 모계 라인 중 하나를 예로 보면, 번식(29%), 자돈 품질(20%), 효율(14%), 성장(14%) 등에 상대적으로 고른 가중을 두고 있으며(Fig. 4), 부계 라인도 사료 효율(31%), 성장(23%), 도체(15%) 등으로 가중을 두어(Fig. 5) 타사와 다소 다른 육종 전략이 보인다. Hermitage Genetics의 모계 라인은 산자수와 복체중(Fig. 6, 7) 에, 부계 라인은 사료 효율과 정육율(Fig. 8, 9) 에 상당한 가중을 두고 있다. 그 밖의 여러 회사들의 육종 목표들은 해당사의 웹사이트에서 확인할 수 있으나, 편의를 위해 글 말미에 그림으로 정리해 놓았다. 각 회사들이 보여주고 있는 라인들을 동시에 게재하여, 각 회사가 추구하는 육종 목표를 한 눈에 비교할 수 있을것이다.
그렇다면 육종 목표에 포함시킬 형질들은 어떤 기준으로 결정하는 것이 맞을까? Knap (2014)은 목표 형질들을 언제, 어떤 이유로 선택되어야 하는지에 따라 6 개 그룹으로 분류하고 있다.
그룹 1 : 복잡하지 않은 유전적 개량을 추구하면서, 일반적으로 오래 전부터 수익 모델의 명백한 부분을 형성하는 형질들. 즉, 중도 내지 고도의 유전력을 가지는 고전적인 생산 형질들과 기록이 쉬운 형질들이 여기에 속한다. 이들은 전형적인 저(低)기술 육종 프로그램에서 진지하게 선발되는 형질들이라고 기술하고 있다. 돼지의 경우 성장율과 등지방두께가 이 그룹에 해당된다.
그룹 2 : 수익 모델에서 그룹 1에 속하는 형질들과 똑같이 중요한 부분을 차지하지만, 개량을 위해서는 전문적인 기술이 필요한 형질들이다. 돼지에서는 BLUP이 필요한, 산자수와 같은 유전력이 낮은 생산 형질 또는 사료 섭취와 같은 복잡한 데이터 기록을 가지는(장비가 필요한) 형질들이 이 그룹에 속한다고 할 수 있다. 이러한 형질들은 1970 년대 후반부터 일부 유럽 육종 시스템에서 심각하게 고려되었던 목표 형질들인데, 능력검정을 위해 개개의 축사도 필요하고, 전체적으로 정교한 계획이 필요하여 상당한 노력을 필요로 한다고 기술하고 있다. 이 형질들에 대한 실질적인 유전적 개량 역시 적절한 기술이 도입된 10 년에서 20 년 후에야 달성되었다고 한다. 초창기에는 몇몇 회사들만 시도하였는데, 주목할만한 유전적 개량을 달성하자 시장에서의 반응이 좋아졌고, 나머지 회사들도 국제 경쟁을 고려하여 따라해야만 했다고 한다.
그룹 3 : 그룹 2와 같은 형질들이지만, 적대적 형질이면서 수익성에 영향을 미치는 형질들이고, 여기에는 치사율 같은 형질들이 포함된다고 볼 수 있다. 치사율은 수익 모델에 포함되는 즉시 수익성에 큰 영향을 미치게 된다. 이 그룹에 속하는 형질은 1990 년대 초부터 고려되기 시작했지만, 아주 많은 양의 혈통 기록과 다양한 관리 조건에서 필요로 하기 때문에 실제 생산 환경에서는 적용되기 쉽지 않았다. 한편, 앞서 봤듯이 일부 회사는 강건성을 중요하게 여기고 있는데, 생산성과 강건성은 부(否)의 상관을 보인다. 따라서, 앞으로는 이러한 생산성과 강건성 사이의 관계같이 형질들간의 경제적 가치를 최적화할 수 있는 방법을 찾는 것이 육종의 핵심 요소가 될 것이라고 기술하고 있다.
그룹 4 : 새로운 정책으로 인해, 시장에서 갑작스러운 경제적 가치를 창출하는, 수익 모델의 일부를 형성하는 형질(예: 웅취)이 여기에 속한다. 유럽 연합은 2018 년까지 외과적 거세에 대한 금지 조치를 준비하고 있다. 이러한 형질들은 생산자 수익에 실제 어떤 영향을 미치는지 측정하기 어렵기 때문에, 초기에는 경제적 가치를 산정하기 어렵다고 한다.
그룹 5 : 그룹 4와 같은 형질들이지만, 수익성에 미치는 영향이 발생하기 전에, 미래의 기대에 의해, 그리고 분명히 원하는 측면에서 채택되는 형질들을 이 그룹으로 분류하고 있다. pH나 근내 지방함량, 지방산 조성 등과 같은 육질 형질들은 실제 육가공 업체로부터 수익이 분배되지 않지만 육종업체들은 그 형질들을 고려하고 있다.
그룹 6 : 시장에서 가치를 지니기 때문에 유전적 개량이 필요한, 오래된 형질들이 여기에 속한다. 생산자 수준에서 수익성에 대한 기여를 정량화하기 어렵기 때문에 적절한 수익 모델을 적용하기 힘든 형질들로, 예를 들면, 지제 및 유두수, 근육의 형태 등이 여기에 속한다고 한다.
위에 나열한 6개의 그룹을 살펴보면, 현재 자신이 어떤 육종을 하는지 가늠할 수 있는 좋은 잣대가 될 수 있을 것 같다. 수직 계열화된 기업의 한 부분으로써 작동하는 육종회사는 사실상 크게 영향을 받지 않겠지만, 그렇지 않은 경우라면 어떤 형질을 왜, 어떻게 개량할 것인가를 고민하는 계기를 갖는 것도 필요한 시점이 아닌가 생각해본다.
Development of Technologies
양적 유전학(quantitative genetics)을 기반으로 한 육종 전략은 지난 40-50 년 동안 돼지의 유전적 개량를 주도했다. 1980 년대 후반부터 동물 모델(Animal Model)의 도입으로 본격적인 유전적 개량이 시작되었다. 대부분의 종에서 동물 모델이 공통적으로 도입되었지만, 돼지 육종 프로그램에서는 1990년대 초에 분자 유전학의 도입으로 또 다른 혜택을 입게 된다. 돼지 스트레스 증후군(PSS)을 일으키는 PSE 돈육의 보인자을 확인하기 위한 DNA 검사의 개발로 대부분의 돼지 집단에서 이 돌연변이를 관리하거나 제거할 수 있었다. 이를 시작으로 후보 유전자와 양적 형질 유전자좌(QTL)를 확인한 많은 다른 연구들이 생산 형질과 관련된, 잠재적으로 흥미로운 다른 DNA 마커들을 찾아내었으나, 몇 가지 예를 제외하고 경제적으로 중요한 형질에서 유전적 다양성을 그다지 많이 설명하지 못하여 실제 현장에서 완전히 활용되지 못했다(Samore and Fontanesi, 2016).
이러한 문제는 마커 또는 유전자 보조 선발의 향상된 버전으로 간주될 수 있는 유전체 선발 개념의 도입으로 새로운 전환기를 맞는다. Meuwissen et al.(2001)은 유전체 선발을 처음으로 제안하였는데, 유전체를 모두 커버하는 수천 개의 단일 염기 다형성(single nucleotide polymorphism; SNP)에서의 유전자형에 기초하여 개체의 유전가(genetic value)를 예측하였다. 이들의 방법은 각 마커의 개별적인 중요성에 대해 걱정하지 않고, GEBV (genomic estimated breeding value) 를 계산한다. 6만여개의 마커 효과 추정치가 포함되므로, 대부분의 중요하지 않은 효과는 서로 상쇄될 것이라고 가정한다. 이것은 유전체 차원의 연관성 연구(GWAS)에서 일반적인 관행처럼 유의성 검정을 매우 엄격하게 수행하였던 초기 방법과는 현저한 대조를 이룬다. Nielsen et al.(2010)은 BLUP 평가에 SNP 정보를 더하면, genotyping (譯註: 유전자형이 무엇인지 파악하는 과정) 된 개체 및 그렇지 않은 개체 모두에서 육종가 추정치의 신뢰도를 높일 수 있다는 사실을 보여주었다. 따라서, 유전체 선발은 유전체에 대한 모든 SNP 대립 유전자의 효과를 합산하여 각 개체의 육종가(유전체 육종가 또는 GEBV)를 예측한다. 이 SNP 마커의 효과는 훈련 자료(training data set), 즉, 표현형 자료와 유전체 정보가 모두 있는 집단에서 유전자형에 대한 표현형의 회귀로써 추정된다. 여기서 구한 추정치들을 가지고, 표현형 정보가 없지만 유전체 자료만 있는 개체에 대해 GEBV를 예측한다. 이는 어린 연령에서 축산 동물을 선발하는 것을 가능케하고, 더불어 세대 간격을 줄이게 되어 유전적 개량량을 증가시킨다. 또한, 유전체 정보로 인한 선발 정확도의 상승으로 개량량을 더욱 높이게 된다.
유전체 선발의 개념이 처음 정의되었을 때는 필요한 기술과 정보가 충분치 않았지만, 이후 수백만개의 다형성(SNP) 이 파악되고, 동시에 비용은 낮아지면서, 새로운 통계적 방법이 개발되어 유전체 선발의 수행 가능성을 한층 끌어올렸다. 이러한 진보는 2009 년 미국에서 젖소 평가를 통해 유전체 선발의 첫번째 실용적이고 광범위한 구현을 촉발시켰다(VanRaden et al., 2009). 그 이후로 세계 대부분의 젖소 개량 기관들은 유전체 선발 프로그램에 동참하고 있다. 유전체 선발은 기존의 방법으로는 개량하기 어려운 형질에 대해 많은 정보를 제공한다. 개량하기 어려운 형질이라 함은 대개 측정하기 어렵고, 생애 주기에서 후기에 나타나며, 때로는 한쪽 성별에서만 측정이 가능하고, 심지어 유전력이 낮은 경우를 말한다(Knol et al., 2016). 따라서, 유우의 개량에서는 유전력이 낮지만 경제적으로 유의미한 번식 능력 개량(예: 딸소의 임신율) 에서 소기의 성과를 거둔 것으로 평가받고 있다(Adriana et al., 2016).
돼지 유전체 선발에서 고려되는 형질
돼지에서도 유우에서와 같은 시도가 이루어지고 있다. 돼지 유전체 선발을 위해 고려되는 형질은 능력 또는 번식 형질과 같은 전통적으로 선발된 형질에서부터 웅취, 장수성, 건강 및 질병에 관련된 형질에까지 다양하다. 돼지 육종에서 모계 라인의 번식 및 기능 형질은 모계 라인의 상업적 경쟁력을 위해 중요하며, 효율적이고, 지속 가능한 생산을 보장해야 한다. 그러나, 일반적으로 낮은 유전력으로 인해, 이러한 형질들에 대한 유전적 개량은 매우 느리고, 이전에 사용해왔던 BLUP 예측은 친척 기록에 큰 비중을 두고 있으며, 친척들간의 유사한 육종가 추정치를 양산하여, 근친도 상승에 대한 문제를 야기한다. 반면, 유전체 선발은 후보 개체들의 보다 정확한 선발과, 근친도의 감소를 어느 정도 방지할 수 있으며, 표현형 기록이 없는 후보 개체들의 정확한 육종가 추정을 가능하게 한다. 이에 따라 등지방 두께 및 도체중, 일당증체량 등과 같은 생산 형질(Akanno et al., 2014; Jiao et al., 2014), 지제 및 건강 형질 등과 같은 장수성에 관한 형질(Boddicker et al., 2014), 육질에 관한 형질(Miar et al., 2014), 총산자수 및 사산, 이유전 치사율, 자돈 생존율 등과 같은 모체 번식 형질(Lillehammer et al., 2011; Tusell et al., 2013) 등에서 유전체 선발에 대한 연구가 발표된 바 있다. 이유후 치사율은 양돈에서 경제적으로 아주 중요한 형질 중 하나이지만, 측정하기 쉽지 않은 형질이다. 낮은 발생률(약 5%) 과 낮은 유전력(약 0.05) 을 보이면서, 이진으로 표현(0 또는 1) 되고, 환경효과에 많이 좌우된다(Knap, 2014). 따라서, 이러한 형질이 유전체 선발에 잘 활용되려면 표현형을 정확하게 수집해야 하고, 그와 더불어 합리적인 통계력(statistical power) 을 얻기 위해 매우 많은 자료의 양을 필요로 한다(Knol et al., 2016).
유전적 개량량은 유전적 변이, 선발 강도, 세대 간격, 육종가 추정치의 정확도 등에 달려있다. 보통 가장 정확한 육종가 추정은 후대 검정에서 비롯된다. 유전체 선발이 도입되기 전에 유우에서는 후대 검정을 통해 높은 정확도를 유지하여 유전적 개량량을 실현한 반면, 돼지에서는 세대 간격이 짧다는 점을 이용하여 유전적 개량의 실현을 추구해 왔다. 그럴 수 밖에 없는 것이 소의 경우는 세대 간격이 상대적으로 길고, 인공 수정의 파급력이 돼지보다 훨씬 강하기 때문에, 그와 같은 방법을 통해 수컷의 선발 강도를 높일 수 있었던 것이다. 따라서, 유전체 선발의 장점 중 하나인 어린 연령에서 육종가 추정의 정확도를 향상시키는 것은, 유우에서 세대 간격을 획기적으로 단축시키는 한 방편이 되지만, 돼지에서는 그것이 그다지 큰 장점이 될 수 없다. 대신 돼지의 경우는 이전과 동일한 선발 연령대에서 정확도가 상당히 높아지는 이점으로 대체된다(Knol et al., 2016).
참조 집단(reference population)
유전체 선발을 위해서는 표현형 기록 또는 후대 검정을 통해 높은 정확도의 육종가를 가진 개체들이 genotyping되어 참조 또는 훈련 집단으로 구성되어야 한다. 이를 통해 마커 효과 추정치를 구하고, genotyping된 선발 후보 개체에 적용하여 GEBV를 계산한다. 이 후보 개체들은 중복 계산을 피하기 위해, 참조 집단의 일부로 하지 않으며, 표현형 기록을 가질 필요가 없다. 참조 집단 구성의 핵심은 선발 후보군과의 유전적 관계이고, 이것은 GEBV의 신뢰도와 직접적으로 연결된다(Clark et al., 2012). GEBV의 정확도는 세대에 걸쳐, 급속히 감소할 수 있기 때문에, 특히 선발 후보 개체들과 관련해서, 세대에 따라 표현형을 지속적으로 수집하여 유전체 선발 예측 모델을 업데이트해야 한다. 따라서, 참조 집단은 정기적으로 업데이트되어야 마커 효과 추정치를 다음 세대의 육종 집단과 일치시킬 수 있다. 이러한 이유로 참조 집단과 주요 육종 집단은 동일하지 않다. 이는 높은 생물학적 보안이 유지되는 핵돈군보다 질병이나 영양, 환경 등 “현장”에 더 가까운 출하용 양돈장에서 참조 집단 그룹을 유지함으로써 그 효과를 극대화할 수 있다(Knol et al., 2016). 또한, 마커 효과 추정치를 충분히 신뢰할 수 있는 참조 집단에서 충분한 genotyping과, 적절한 선발 강도를 얻을 수 있도록 선발 후보 개체에서도 충분한 genotyping을 진행해야 하는 한편, genotyping하는데는 비용이 적지않게 들기 때문에, 비용 문제를 최적화하기 위한 전략적 시스템도 필요하다.
단일 단계(single-step) 모델과 다단계(multistep) 모델
유전체 평가를 계산하는 방법은 단일 단계 방법과 다단계 방법으로 분류된다. 단일 단계 방법에서는 실제 기록을 분석하고, 마커 효과와, 마커 효과에 포함되지 않는 상가적 유전 변이가 추정된다. 축군-연도-계절 또는 성별과 같은 효과도 모델에 포함된다. 단일 단계 방법에는 몇가지 단점이 있다.
1) 분석 모델에 포함된 방대한 수의 방정식 때문에 광범위한 컴퓨팅이 필요하다.
2) 대다수의 기록이 있는 동물이 유전자형을 가지고 있지 않다는 문제를 해결해야 한다.
3) 방대한 양의 방정식 때문에, 솔루션은 반복(iteration) 을 통해서만 유도될 수 있으며, 여기에서 수렴에 대해 문제가 제기될 수 있다.
다단계 방법에서는 먼저 표현형 자료와 혈통에 기초하여 계산된다. 그 다음 유전자형을 가진 개체의 유전 평가가 유전자 마커의 함수로 분석된다. 다단계 방법의 장점은 정기적인 평가에 변화를 가져오지않고, genotyping된 어린 개체에 대한 유전체가(genomic value)를 예측하는 것이 간단하는 것이다. 또한, 전체 컴퓨팅 시간은 단일 단계 방법보다 훨씬 적게 든다. 단점에는 분산 성분(Guillaume et al., 2008) 또는 선발 지수(VanRaden et al., 2009)에서 가중치에 대한 문제, 정보의 손실, 편의(biased)된 평가(Misztal et al., 2009; Aguilar et al., 2010)등이 언급되고 있다. 또한, 다형질 평가나 test-day model과 같은 분석 모델로의 확장이 분명치 않다. 따라서, 전반적인 추세는 단일 단계 모델을 사용하는 방향으로 진행되고 있다. 단일 단계 방법이 이론적인 고려 사항에서 우위에있는 것처럼 보이지만, 실제 데이터에 대한 두 가지 방법 간의 예측 정확도 차이는 미미한 수준이라고 한다(Weller, 2016).
경제적 측면
육종가 추정의 정확도를 높이기 위해서 유전체 선발이라는 도구가 도입되고 있지만, 비용에 대한 문제는 여전히 의문점으로 남아있다. 유전체 선발을 하더라도 여전히 표현형 수집에는 비용이 들고, 무엇보다도 genotyping하는 데 비용이 들기때문에, 유전체 선발을 구현하는데 드는 비용 정도는 회수할 수 있을만큼 정확도가 향상되어야 하는 것은 기업의 입장에서는 당연하다. Genotyping의 비용은 1990년부터 10,000 배 이상 감소했고, 여전히 감소하고 있지만, 유전체 선발에서 요구되는 자료의 수는 그만큼 증가하고 있다. 따라서, 이러한 접근 방식은 여전히 상당한 비용이 발생할 수 밖에 없는 구조이다. 이러한 문제는 Habier et al.(2009)에 의해 제안된 imputation 방법, 즉, 저밀도 칩에서 얻어진 유전자형을 고밀도 칩에서의 유전자형으로 확대하는 방법을 통해 해결될 수 있었다. 초기 집단은 집단의 일배체형(haplotype)을 결정하기 위해 필요한데, imputation을 통해 저밀도 칩으로부터 유전자형을 추론할 수 있기 때문이다. Imputation이 사용되면, 선발 후보 개체들은 저밀도 칩을 사용하여 genotyping되고, 그 이후 선발된 개체들은 고밀도 칩으로 다시 genotyping된다. 비용 절감을 위해서 육종회사는 수컷만 genotyping하는 것으로 방향을 정할 수도 있다. 이 방법으로 비용을 절감할 수 있지만, 한쪽 성(性)에만 제한되어 있는 형질이나, 표현형 자료가 정기적으로 수집되지 않는 형질의 경우, GEBV의 정확도가 감소될 수 있다고 한다.
Abell et al. (2014) 은 시뮬레이션을 통해 유전체 선발에 필요한 비용을 살펴보았다. 이들은 저밀도 SNP 칩을 사용하는 것이 genotyping 비용을 줄이는 효과적인 방법이 될 수 있다고 말한다. 이들의 결과에 의하면, 시뮬레이션에서 가정한 조건이 모두에 적용될 수는 없고, 유전체 선발에 투자한 육종회사가 수익을 어떤식으로 달성하는지에 따라 계산이 달라지겠지만, 출하 단계의 이유 자돈을 기준으로 로열티를 받는 것으로 가정했을 때, 연간 비용은 모계 라인 및 터미널 라인의 정액 팩당 3.68불 및 0.18불의 비용이 발생한다고 한다. 이유 자돈 당으로 계산했을때는 모계 라인에서 0.083불, 터미널 라인에서 0.047불의 비용을 산정하였다. 각 회사마다 다양한 전략이 있겠지만, 계열화된 생산자는 출하 부분에서 개량된 생산 효율로부터 이익을 얻거나, 소비자에 대한 소매 가격을 올림으로써 유전체 선발 비용을 만회할 수 있을 것으로 기술하고 있다. 반면, 육종회사는 시장 점유율을 높이거나, 자신들의 유전 자원에 프리미엄을 더 붙여서 유전체 선발의 비용을 회수해야 할 것이다. 한가지 더 생각해야할 사항은 위에서 계산된 비용은 모두 예상된 유전적 개량량을 바탕으로 산정된 것이라는 문제이다. 출하 단계에서의 환경 요인은 유전체 선발시의 생산 환경과 다르기 때문에 실제로 실현되는 유전적 개량량은 예상보다 작아질 수 있다. 따라서 이러한 경우 선발시의 생산 환경, 즉 핵돈군 수준에서 더 많은 유전적 개량이 요구될 수 있다. 반면, 이들은 유전체 선발로 인한 마케팅 가치에 대한 요인도 언급하면서, 투자금을 회수하기 위한 최소한의 유전적 개량이 꼭 그만큼 되지 않을 수도 있다고 기술하고 있다.
전체적으로 볼 때, 표현형에 따라 자료를 수집하는데 필요한 인프라와 관련된 비용이 상당히 높게 발생할 수 있기 때문에, 유전체 선발이 육종가의 정확도를 높이는 것은 사실이지만, 드는 비용을 상쇄할 수 있을만큼의 유전적 개량량의 증가도 필수적이므로 회사 운영진의 현명한 판단이 필요할 것으로 보인다.
고려해야할 측면
유우에서의 성공사례는 홀스타인 종인데, 모두 순종으로 쓰이며, 혈통적인 면에서 밀접하게 관련된 소들이 전세계에 엄청난 수로 존재한다. 이들 모두가 잠재적으로 표현형을 참조집단에 기여할 수 있고, 또한 여러 국가와 기관이 협력하여, 16,000두 이상의 숫소를 참조 집단으로 확보해 놓아 유전체 선발의 적용을 최적화시키고 있다. 하지만, 돼지 육종의 경우는 각 육종회사와 순종 협회들이 별도로 조직 운영하기 때문에, 표현형 가용성은 매우 단편적이다. 전형적인 대형 순종 돼지 집단은 약 2,000두의 모돈을 가지고, 일년에 약 50두의 웅돈을 선발하며, 대략 평균 40두의 딸을 생산한다. 홀스타인 종에서 유전체 선발 초기에 설정했던 1,500두의 보증 종모우에서 각각 100두가 넘는 딸의 표현형이 있었던 상태와 비교하면, 30년의 누적 자료가 필요한 것이다.
또, 한가지 문제점으로 제기되는 것 중 하나는 소와 다르게 출하시 사용되는 품종은 대부분 교잡종이어서, 순종이 대부분 포함되어있는 핵돈군에서 일어나는 유전체 선발이 실제 생산 환경에서는 그 결과를 장담할 수 없다는 것이다. 더욱이 질병 저항성과 같은 중요한 형질은 핵돈군에서 측정할 수 없다. 유전체 모델을 사용하면, 표현형 자료와 유전자형 자료를 모두 가진 교잡종 개체에 대해 마커 효과를 추정하여, 유전체 정보가 있는 순종 라인을 평가할 수 있기는 하다. 하지만, 잡종강세는 교잡종 돼지 생산에서 많은 형질에 중요한 역할을 하기 때문에, GEBV가 교잡종 능력 자료에 기초하게 된다면, 통계 모델 내에 잡종 강세를 설명할 수 있는 비상가적 유전 효과를 포함해야, GEBV의 신뢰도를 높이고, 편의(bias)를 줄일 수 있다. GEBV 예측에서 비상가적 효과를 설명하는 것은 이론적으로나 계산 상으로 복잡할 수 있으며, 추가 연구를 통해 이러한 유전적 구성 요소를 분석하여 이로부터 이점을 취할 필요가 있다(Su et al., 2012; Zeng et al., 2013; Boysen et al., 2013). 유전체 선발의 도입에 대한 전망은 일반적으로 유전력이 낮은 형질 또는 집단의 일부에서만 기록되는 형질, 즉 암컷에서만 기록되는 산자수나 건강 형질에 대해서 이점이 클 것으로 보인다. 대조적으로 중도에서 고도의 유전력을 지닌 능력 형질이나 돼지 검정 프로그램의 일부를 사용하여 기록된 형질에 대해서는 이점이 크지 않을 것으로 생각된다(Samore and Fontanesi. 2016).
Other Trends
육종가의 역할 중의 중요한 하나는 새로운 형질의 발굴이다. 경제적으로 중요한 새로운 형질들을 육종에 사용하기 위한 시도들이 꾸준히 이어지고 있다. 이미 수차례 보고된 바 있지만, 공격성에 대한 육종 시도가 꾸준히 진행되고 있다. 지난 2016년 12월에 열린 NSIF에서도 돼지 행동 연구에 대한 보고가 많이 있어, 새로운 연구 방향의 하나로 여겨지고 있다. 그룹 사육(group housing)은 여러 단계에서 이뤄질 수 있는데, 이유후나 육성/비육 단계에서도 이뤄질 수 있다. 문제는 그룹화 된후 첫 3일동안은 싸움이 심하게 발생하여, 신체에 상해를 입거나, 그로 인한 질병 또는 사료 섭취의 저하로 이어지게 된다(Siegford, 2016).
다시 말해 이러한 싸움이 면역계를 억압하고, 질병 발생의 가능성을 증가시키며, 사료 섭취량을 낮추기 때문에 공격성에 주의해야 한다고 한다. 육성돈과 비육돈을 따로 사육했을때, 같이 사육했을 때보다 육성돈은 일일 사료 섭취량이 15 % 이상 증가했으며, 비육돈은 적어도 10 % 이상 증가했다고 한다. 분만전 스트레스도 공격성의 전초가 된다. 임신돈이 같이 있으면 싸움을 통해 종속과 지배 관계의 서열화를 진행한다. 스트레스를 겪은 어미로부터 생산된 자돈은 더 공격적인 행동을 보이고, 통증에 대한 반응도 과민한 것으로 관측되었다고 한다. 이유후 성장율도 모돈을 같이 키운 그룹의 자돈에서 더 낮았다고 한다. 이렇게 생산된 자돈이 미경산돈으로 교체되면 또다시 어미로써의 역할을 제대로 하지 못하여, 자신의 자돈에게 더 공격적이 되기 때문에, 생존율도 낮아진다고 한다(Turner, 2016). 미시건 대학 연구팀은 돼지 신체에 발생한 상처를 바탕으로 주관적인 점수를 계산해 GBLUP 모델에 적용한 바 있다. 대상은 1,079두의 요크셔 순종이었고, 절반은 미경산돈, 절반은 거세돈이었다. 혈액을 채취해 70K SNP 칩을 이용하여 분석한 결과, 혼합 후 첫 24시간동안의 유전력은 0.17에서 0.42였고, 3주동안의 유전력은 0.05에서 0.27이었다고 한다(Siegford, 2016).
때로 공격성은 육종을 할 수 없거나 하기 힘든 형질일 것이라고 생각하기 쉬우나, 그와 정반대 성격의 형질인 온순함 또는 순종성, 길들이기 쉬움의 정도 등으로 육종에 성공한 사례가 있다. 러시아의 한 연구팀은 여우를 50여년간 위와 같은 형질을 기준으로 선발실험을 진행한 바, 여우에서 개와 같이 꼬리를 치며, 사람에게 순종적으로 변하는 모습을 볼 수 있었다고 한다(Trut, 1999). 연구팀은 공격성이 어떤 면에서 지적으로 뛰어난, 즉 영리함을 반영하는 것이라고 연결짓기도 했다. 이것은 인류가 반려동물로 손꼽는 개가 어떻게 회색 늑대종(種)로부터 유래되었는지 설명하는 한 단서가 되었다. 따라서, 돼지의 공격성도 거시적인 측면에서 보았을 때, 분명히 육종이 가능할 것이지만 그것이 생산형질과 또 어떻게 어떠한 형태로 상관을 보일지는 또 다른 연구의 대상일 것이다.
돼지에서 또 다른 육종 시도는 장내 미생물과 성장의 관계를 알아보려는 것이다. 미네소타 대학 연구팀은 가축의 여러 품중에서 장내 미생물을 측정하여, 다른 품종간에 미생물 조성이 다르다는 것을 발견하였다. 또 최근의 한 연구에서는 사료효율이 좋은 암소와 그렇지 않은 암소의 미생물 조성이 다르다는 것을 보고한 바 있다. 흔히 언급되고 있는 장내 좋은 박테리아와 나쁜 박테리아의 비율이 성장에 어떤 영향을 미치는지 알아낸다면, 이는 돼지의 건강 증진과 질병 예방을 동시에 추구할 수 있을 뿐 아니라 돼지 육종의 주된 목적인 성장율 극대화와 사료 효율 증진에 직결되는 해결책이 될 수 있을 것이다(Maltecca, 2016).