1Sobaeksan National Park Office, Korea National Park Service, Yeongju 36044, Republic of Korea
2National Park Service Business Registration, Wonju 26466, Republic of Korea
3Deogyusan National Park Office, Korea National Park Service, Muju 55557, Republic of Korea
4Woraksan National Park Office, Korea National Park Service, Jecheon 27226, Republic of Korea
5Gayasan National Park Office, Korea National Park service, Hapcheon 50201, Republic of Korea
6National Park Institute for Wildlife Conservation, Korea National Park Service, Yeongju 36015, Republic of Korea
Correspondence to Sang-Hyun Han, E-mail: hansh04@knps.or.kr
Volume 9, Number 3, Pages 157-170, September 2025.
Journal of Animal Breeding and Genomics 2025, 9(3), 157-170. https://doi.org/10.12972/jabng.2025.9.3.6
Received on August 28, 2025, Revised on September 23, 2025, Accepted on September 29, 2025, Published on September 30, 2025.
Copyright © 2025 Korean Society of Animal Breeding and Genetics.
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0).
This study examined the geographic distribution of maternal lineages and genetic diversity in the endangered Eurasian flying squirrel population in Sobaeksan National Park (NP), South Korea, based on the sequence polymorphisms of mitochondrial DNA (mtDNA) D-loop region using noninvasive fecal samples. A total of 201 D-loop sequences were further identified as 52 individuals based on microsatellite (MS) genotypes. The entire population had eighteen haplotypes with high levels of haplotype diversity (Hd) (0.919±0.020) and nucleotide diversity (π) (0.0267±0.0015). The three populations within the Sobaeksan NP were related to each other by matrilineal lineage regardless of isolation by geographic distance. Genetic differentiation (Fst) was higher among populations within Sobaeksan NP and Gangwon-do (0.196-0.227) than those among three populations within Sobaeksan NP (0.044-0.117), and migration rate (Nm) was lower (0.85-1.03) than those among three populations (1.89-5.43). Phylogenetic analysis results showed that the genetic distances between the three populations of Sobaeksan NP were close (0.0273-0.0321), so those are estimated to be closely related to each other, but those appeared to be genetically distant from the haplotypes found in Gangwon-do (0.0422-0.0495) and Jeollanam-do (0.0513-0.0551). These findings suggest that the results of this study will provide crucial information for developing future management strategies to manage the habitat and enhance genetic diversity of the endangered Eurasian flying squirrel in the Sobaeksan NP, South Korea.
endangered, Eurasian flying squirrel, genetic diversity, haplotype, maternal
주로 나무 위에서 생활하는 야행성 포유동물인 하늘다람쥐(Pteromys volans)는 북유럽의 핀란드에서부터 극동 아시아인 러시아 Sakhalin, 일본 Hokkaido, 한반도, 중국 동북부 지역에 걸쳐 분포한다(Won, 1967, 1968; Nowak, 1999). 우리나라에서는 도서 지역들을 제외한 전국 대부분의 높은 산지에서 발견되지만, 지역별 개체수는 많지 않다. 20세기 후반 경제발전과 산업화를 위한 무분별한 산림개발로 자연 서식지가 사라지고 일부 지역에서는 한약재로도 이용되면서 개체군이 급감하였다(Won, 1967; Won and Smith, 1999; Yoon et al., 2004). 현재 우리나라에서 하늘다람쥐는 환경부 멸종위기야생생물 II급, 문화유산청 천연기념물로 지정되어 보호되고 있다(Ministry of Environment, 2005; Cultural Heritage Administration, 2009). 전 세계적으로 개체군이 감소 중이며, 국제자연보전연맹은 관심이 필요한 단계-least concern-로 선정하였다(Shar et al., 2016). 멸종위기종을 포함한 야생동물의 종, 집단의 보호와 보전을 위해서 유전적 분화와 집단 구조, 유전적 다양성에 대한 정보는 매우 중요하다(Pritchard et al., 2000; DeSalle and Amato, 2004; Ghildiyal et al., 2023). 집단 내에서 고유한 유전 형질의 소실, 지리적 격리에 의한 근친과 고립, 집단 분화에 대한 자료들은 실질적인 집단-집단 또는 아집단-아집단 사이의 생식적 연결성, 지역 간 이동, 유전적 안정성을 판단하는 분자적 근거가 된다(Kobayashi et al., 2012; Saremi et al., 2019; Mellya et al., 2025)
최근 분자생물학 분석기술의 발달로 DNA 정보 수집이 용이해지면서, 동물의 계통발생과 진화, 유전적 다양성 연구에서 중대한 성과들을 보고하고 있다(Harris et al., 1998; Iverson et al., 2007; Xu et al., 2023). 미토콘드리아는 세포 내에 수백에서 많게는 수만 개까지 존재하여 적은 시료에서도 효과적으로 DNA 정보를 습득할 수 있다. 또한 mtDNA가 모계유전을 하고, 핵 DNA에 비해 높은 돌연변이율을 나타내어 빠르게 진화하기 때문에(Brown et al., 1979; Giles et al., 1980; Clayton, 1982), 종의 동정, 사람과 가축, 야생동물의 진화와 유전적 다양성 연구에 활발히 이용되고 있다(Caragiulo et al., 2014; Gonzalez-Oliver et al., 2018; Laxmivandana et al., 2022). 야생동물의 분변을 이용한 비침습적 연구들이 시도되고 있으나, 체외에서 산소, 물 등과 접촉하면서 빠르게 산화되어 DNA가 붕괴되기 때문에, 회수할 수 있는 DNA의 양이 적다. 또한 장내에서 유래된 미생물, 소화되지 않은 잔여물, 외부 생명체 등에 의한 오염은 유전자 정보 수집과 해독을 더욱 어렵게 하고 있다(Reed et al., 1997; Hajkova et al. 2006; Lampa et al., 2008). 그렇지만 야생동물을 직접 포획하거나 상해를 주지 않고 유전자 정보를 취득할 수 있어, 분변 DNA 연구는 종의 서식 확인, 개체 추적, 행동권 파악, 유전적 다양성과 연결성 평가, 생태계 내 개체군의 크기 산출 등 다양한 분야에서 활발히 연구되고 있다(Tende et al., 2014; Mumma et al., 2015; Rodriguez-Castro et al., 2018; Ibouroi et al., 2021). 1968년 하늘다람쥐의 번식에 대한 연구보고(Won, 1968) 이후, 현재까지 국내에서 하늘다람쥐에 대한 연구는 서식지의 분포, 서식지 이용 특성과 행동권 예측, 이용하는 나무와 분변 장소의 계절적 특성 등에 관한 연구들이 주를 이루고 있다(Cho et al., 2013; Han et al., 2018; Kim, 2018; Lim et al., 2021). MtDNA cytochrome B (CYTB) 유전자의 다형성에 대한 연구에서 우리나라 집단은 극동 아시아 계통군에 속하지만, 러시아, 중국, 일본(Hokkaido) 집단과는 확연히 구분되어 유전적으로 분화되어 있으며(Oshida et al., 2005; Lee et al., 2008b), 국내에서도 지역적 분화의 가능성을 제안하고 있다(Kim, 2018). 경상북도 북부와 충청북도 북동부의 백두대간에 위치한 소백산국립공원은 우리나라 하늘다람쥐의 주요 서식지 중 하나로, 고치령을 비롯한 여러 지역에서 하늘다람쥐가 지속적으로 관찰되고 있다(Kim, 2010; Lim et al., 2021; Shin, 2023). 하지만 소백산국립공원 내 여러 지역 집단들의 유전적 연관성을 설명할 수 있는 자료는 아직 없다.
이번 연구에서는 소백산국립공원 내에서 하늘다람쥐가 지속적으로 관찰되는 지역에서 수집한 분변에 대한 비침습적 DNA 분석을 통해 집단의 유전적 다양성과 지리적 분포의 상관성을 설명할 수 있는 자료를 마련하고자 하였다.
연구에 이용한 하늘다람쥐 분변은 2018년 2월부터 2025년 5월까지 소백산국립공원에 위치한 하늘다람쥐 모니터링 지역(삼가, 단산)과 하늘다람쥐 분변이 지속적으로 관찰되고 있는 고치령에서 수집하였다(Fig 1; Table 1). 동일한 위치에서 같은 날짜에 수집된 분변 시료 중 두 마리 이상에서 유래된 것으로 추정되는 분변들은 크기와 모양, 색깔, 부식 정도 등을 고려하여 별개의 시료로 구분하여 DNA 추출에 이용하였다. 분변 DNA는 QIAamp Fast Stool Kit (Qiagen, Hilden, Germany)를 이용하여 공급자의 안내에 따라 추출하고, 중합효소연쇄반응(polymerase chain reaction, PCR)의 주형으로 이용하였다.
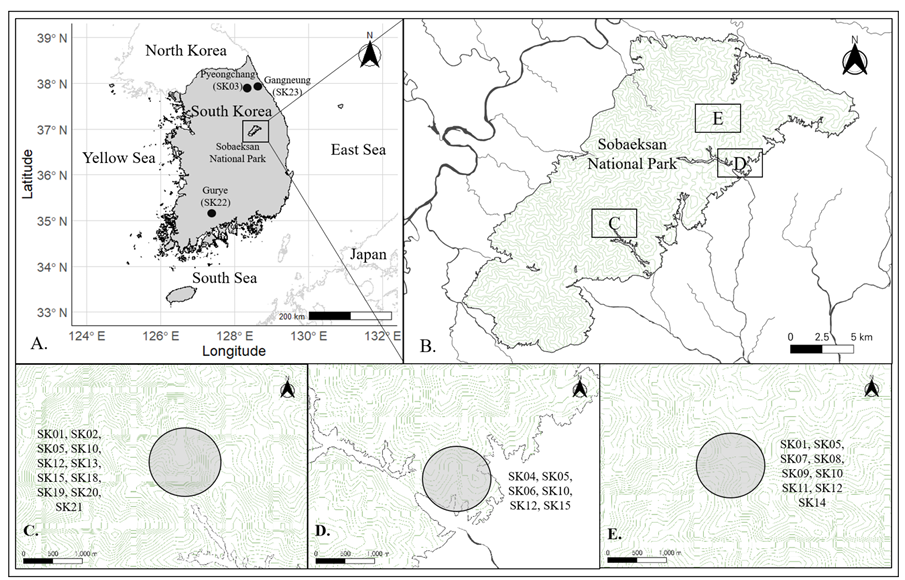
Figure 1. Sample collection sites and haplotypes found in local populations of Eurasian flying squirrels in the Sobaeksan National Park and other locations previously reported. A is a map of a part of Korean Peninsula, including the Sobaeksan National Park (square) and locations (dark circles) where sequences were reported in previous studies, and the codes in parentheses represent the haplotypes of each location. B represents the three sample collection and study sites (C-E) in the Sobaeksan National Park. C-E represent Samga, Dansan, and Gochiryeong research locations, respectively, and gray-colored circles indicate sample collection sites. Codes within the map are the D-loop haplotypes found in each population.
Table 1. Information on the number of samples collected, the number of samples used for analysis, and the number of animals determined through MS genotyping
테이블
1some samples were further subdivided according to size, shape, color and status, and separately used for DNA extraction.
2was determined by comparing the MS genotypes in each population.
추출한 DNA를 주형으로 mtDNA D-loop 영역은 Ryu et al. (2013)이 보고한 미토콘드리아 DNA 전체 서열(NC_019612)의 1545715945 nt.의 489-bp 절편을 포함하는 영역을 대상으로 하였다. PCR 프라이머는 NC_019612를 기준으로 Primer3 web program (Untergasser et al., 2012)을 이용하여 제작하였다: PvolDLF 5’-TGC TAT TAC TCG ACC AGT TAC CAA A-3’, PvolDLR 5’-CCA GAT GCC AGA TAA AGT TTC AG-3’. PCR은 Multiplex Plus PCR Kit (Qiagen, Hilden, Germany), DNA 용액과 프라이머, 증류수를 혼합하여 20 ul로 반응하였고, Mastercycler Nexus Gradient (Eppendorf, Wesseling-Berzdorf, Germany)로 증폭하였다. PCR 반응은 94℃에서 5분간 초기 변성 후, 94℃ 30초, 58℃ 45초, 72℃ 45초로 이어지는 연쇄반응을 35회 수행한 후, 72℃에서 5분간 최종 신장하였다. 증폭이 끝난 PCR 산물은 1.5% agarose 겔 상에서 전기영동하여 확인한 후, DNA 염기서열 분석에 이용하였다. 정제한 PCR 산물은 dyetermination 방법으로 반응한 후, ABI Automated DNA Sequencer 3730XL (Applied Biosystems, Foster City, CA)을 이용하여 서열을 결정하였다. 결정된 서열은 미국 국립생명공학정보센터의 nucleotide database에서 basic local alignment search tool (BLAST, https://blast. ncbi.nlm.nih.gov/Blast.cgi) 검색을 통해 기존에 보고된 서열 중 가장 유사한 서열을 탐색하여 종을 재확인하고, 계통 유연관계 분석을 위해 하늘다람쥐와 근연종에서 보고된 서열의 정보를 수집하였다(Table 2).
Table 2. Genetic diversity for mtDNA D-loop sequences and haplotypes in the Eurasian flying squirrel populations in the Sobaeksan NP, South Korea
테이블
Sobaeksan NP indicates the Sobaeksan National Park, South Korea.
1was determined by comparing the MS genotypes in each population.
S, number of variable sites; π, nucleotide diversity; nH, number of haplotypes; nP, number of private haplotypes; Hd, haplotype diversity; SD, standard deviation.
**significant Fu’s Fs values (P<0.01).
집단별 개체수 확인을 위해 Zittlau et al. (2000)의 MS 마커 1종(GS-10), Painter et al. (2004)의 마커 4종(PvolE1, PvolE5, PvolE6, PvolE10)의 대립유전자형을 결정하였다. 사용한 MS 마커 5종은 사전 연구결과에서 다형정보량이 0.6 이상이며(Zittlau et al., 2000; Painter et al. 2004; Tian et al., 2025), 사전 시험에서 PCR 증폭 효율이 50% 이상인 마커들을 선정하였다(자료 미기재). MS 마커의 정방향 프라이머는 형광염료 FAM을 5’-말단에 표지하였다. PCR은 Multiplex Plus PCR Kit (Qiagen, Hilden, Germany), 프라이머, DNA 용액과 증류수를 혼합하여 10 ul로 반응하였다. PCR 증폭은 Mastercycler Nexus Gradient (Eppendorf, Wesseling-Berzdorf, Germany)를 이용하였고, PCR 반응은 94℃에서 5분간 초기 변성 후, 94℃ 30초, 65℃-55℃까지 순차적으로 온도를 감소시키는 결합반응 45초, 72℃ 45초의 touchdown 방식을 10회 반복하고, 94℃ 30초, 58℃ 45초, 72℃ 45초로 이어지는 연쇄반응을 25회 추가한 후, 72℃에서 15분간 최종 신장하였다. 각각의 MS 마커들은 Taberlet et al. (1996)의 제안에 따라 다중-튜브 접근 방식으로 3회 반복 수행하였다. 증폭된 PCR 산물은 1.5% agarose 겔 상에서 전기영동하여 확인하고, 2배의 증류수, GeneScan 500 LIZ Dye Size Standard (Life Technologies Ltd., Woolston, WA, UK)와 혼합하여 자동염기서열 분석장치 ABI Automated DNA Sequencer 3730XL (Applied Biosystems, Foster City, CA)로 전개하였다. 대립유전자형은 GeneMapper ver. 5 (Applied Biosystems, Foster City, CA)를 이용하여 PCR 산물의 길이에 따라 결정하였다. 전체 5종의 MS 마커 중 3개 이상의 마커에서 정보를 습득한 시료들만을 대상으로 집단별 개체수를 판독하였다. 시료별 유전자형 정보는 CERVUS ver. 3.0.3 (Kalinowski et al., 2007)을 이용하여 동일성검사를 수행하고, 대립유전자형 조합을 비교하여 개체수를 산출하였다.
이 연구에서 결정된 D-loop 서열들과 기존 연구 정보들 수집하여(Table 1), CLUSTAL W (Thompson et al., 1994)로 다중 정렬하였다. 정렬된 서열들에 대한 단상체형 결정, 유전적 다양성 지수와 중립성 지수 산출, 유전적 분화 시험은 DnaSP ver. 6.10.04 (Rozas et al., 2017)를 이용하였다. 염기서열 다양도(π)와 단상체형 다양도(Hd)는 Nei (1987)의 방법으로 산출하였고, 중립진화 분석을 위해 Tajima’s D 지수 (Tajima, 1989)와 Fu’s Fs 지수(Fu, 1997)를 산출하였다. 집단 사이의 유전적 분화와 유전자 흐름을 추정하기 위해 Hudson et al. (1992)의 Fst 지수를 산출하고, 이주율(migration rate, Nm)은 Wright’s Island Model (Nm=(1-Fst)/4Fst) (Wright, 1931)를 이용하여 계산하였다. 하늘다람쥐 D-loop 단상체형들의 계통 유연관계는 MEGA7 (Kumar et al., 2016) 프로그램을 이용하여 살펴보았다. 개별 단상체형 서열들 사이에서 최적 유전적 거리지수는 Bayesian Information Criterion 점수가 가장 낮은 모델을 염기치환 양상을 가장 잘 설명할 수 있는 모델로 간주하여 Tamura and Nei (1993) 방법으로 산출하고, Gamma 이산 분포를 적용하여 서열 간 진화 속도의 불균일성을 완화한 후 Neighbor-joining tree (Saitou and Nei, 1987)를 작성하였다. 집단들 사이에서의 평균 유전적 거리지수 역시 Tamura and Nei (1993)의 방법으로, Gamma 이산 분포를 적용하여 산출하였다. 계통수 분지의 신뢰성을 제공하기 위한 Bootstrap (Felsenstein, 1985)은 1,000회 반복하였고, 분지 형태의 최적화와 공통조상 추적을 위한 외군으로 다람쥐과 동물들에서 보고된 서열들(NC_031847, Hylopetes alboniger; KY410849, Callosciurus finlaysonii; AB239459, Petaurista grandis; AB043814, P. leucogenys; AB239355, P. lena; AB100911, P. albiventer)를 포함하여 계통수를 작성하였다.
소백산국립공원 내 3개 지역(삼가, 단산, 고치령)에서 수집한 166개의 분변에서 추출한 241개 DNA 시료를 분석하여 201개 D-loop 서열을 수집하였다. 이후 MS 유전자형에 따라 52개체를 확인하였다(Table 1). 전체 52개 D-loop 서열들은 46개의 다형적 염기서열에 의해 18개 단상체형으로 구분되었고, 염기서열 다양도(π)는 0.0267±0.0015, 단상체형 다양도(Hd)는 0.919±0.020이었다. 단산 집단은 18개체의 서열들이 36개의 다형적 염기서열에 의해 6개 단상체형으로 구분되고, 1개의 고유 단상체형이 출현했으며, π는 0.0276 ±0.0025, Hd는 0.817±0.054였다. 고치령 집단은 16개체의 서열들이 41개의 다형적 염기서열에 의해 9개 단상체형으로 구분되고, 5개 고유 단상체형이 출현했으며, π는 0.280±0.0023, Hd는 0.833±0.061이었다. 삼가 집단은 18개체의 서열들이 32개의 다형적 염기서열에 의해 11개 단상체형으로 구분되고, 6개 고유 단상체형이 출현했으며, π는 0.0208±0.0024, Hd는 0.915±0.046이었다(Table 2).
이번 연구에서 확인된 소백산 하늘다람쥐 D-loop 서열의 π 값(0.0267±0.0015)과 Hd 값(0.919±0.020)은 북서 유럽의 핀란드(π =0.00156±0.00040, Hd=0.642±0.103), 에스토니아(π=0.00048±0.00007, Hd=0.305±0.047), 러시아 Karelia (π=0.00127±0.00048, Hd=0.700±0.218) 집단들에서 보고된 연구결과(Nummert et al., 2020)보다 더 높았다. 우리나라 멸종위기종의 유전적 다양성에 대한 선행연구에서 설악산 산양(Naemorhedus caudatus)은 π 0.007, Hd 0.886 (Kim et al., 2025), 대구-경상북도 수달(Lutra lutra)은 π 0.01041 ±0.0012, Hd 0.9202 (Hwang and Cho, 2018)로 이번 연구에 비해 π는 낮지만, Hd는 0.8 이상으로 모두 높은 수준이었다. 우리나라-북한의 삵(Prionailurus bengalensis)에서 cytB의 π 값은 0.0016 (Kim et al., 2010)로 하늘다람쥐 cytB의 π 값 0.00616±0.00070 (Lee et al., 2008b)보다 낮았다. 국내에 서식하는 설치류 중 다람쥐(Tamias sibiricus)는 π 0.01886±0.0008, Hd 0.944±0.007 (Lee et al., 2008a), 등줄쥐(Apodemus agrarius coreae)는 π 0.00683±0.00065, Hd 0.967±0.012 (Kim and Park, 2015a), 흰넓적다리붉은쥐(A. peninsulae)는 π 0.00844±0.00042, Hd 0.980±0.010 (Kim and Park, 2015b)로 하늘다람쥐보다 π는 더 높으나, Hd는 낮았다. 이번 연구에서 소백산 집단은 π는 0.005 이상, Hd는 0.8 이상이었고, 이 결과는 유전적으로 안정된 수준으로 간주된다(Nei, 1987; Rogers and Harpending, 1992). 또한, 국내에 서식하는 다른 설치류 종들이나 멸종위기 포유동물에 비해서도 유전적 다양성이 비교적 좋은 상태였다. 한편, 하늘다람쥐 mtDNA cytB 유전자의 π는 극동 아시아 0.0095±0.00246, 일본(Hokkaido) 0.0022±0.0007, 북서 유라시아 계통에서 0.0062 ±0.0055였다(Oshida et al., 2005). Lee et al. (2008b)은 우리나라 하늘다람쥐 집단은 극동 아시아 계통군에 속하며, 중국, 러시아, 일본(Hokkaido)과는 다른 단상체형을 나타내고, π는 0.00616±0.00070으로 북부 유라시아(0.00591±0.00318)보다 높으나, 극동 러시아(0.00950±0.00246)보다 낮다고 하였다. 이번 연구결과에서 또 하나의 흥미로운 점은 소백산 집단에서 D-loop의 π 값(0.0269± 0.0014)은 선행연구의 cytB π 값(0.00616±0.00070) (Lee et al., 2008b)에 비해 매우 높았다는 점인데, 이러한 경향은 북서 유럽 3개 집단(핀란드, 에스토니아, 러시아의 Karelia)에서도 D-loop의 π 값(0.00048-0.00156)이 cytB 유전자의 π 값(0.00027-0.00097)에 비해 훨씬 높았다(Nummert et al., 2020). 이러한 결과들은 하늘다람쥐 mtDNA에서 비암호화영역인 D-loop 영역이 단백질 암호화 유전자인 cytB에 비해 더 빠르게 진화하고 있음을 보여주고 있다.
중립진화 분석결과(Table 2)에서 전체 집단의 Fu’s Fs 지수는 1.360, Tajima’s D 지수는 0.717을 나타내었고, 유의적인 수준을 보이지 않았다(P<0.05). 지역 집단 중에서 삼가, 고치령 집단의 Fu’s Fs와 Tajima’s D 지수는 모두 유의적인 수준을 나타내지 않아(P<0.05), 중립진화 단계에 있다고 할 수 있다. 반면, 단산 집단은 Fu’s Fs가 7.527로 양의 값(+)이면서 높은 유의적인 수준(P=0.003, P<0.01)을 나타내어, 희귀 유전자형의 소실 단계로 보이며, 유전적 병목현상이 진행 중이거나, 다른 지역 집단의 아집단일 가능성을 나타내고 있다(Fu, 1997). 하지만, 단산 집단의 Tajima’s D 지수가 0.717로 양의 값이지만, 유의적인 수준은 아니므로(P>0.10), 유전적 병목현상이 강하지는 않다고 할 수 있다(Tajima, 1989). 선행연구에서 에스토니아 집단은 Tajima’s D 지수가 유의적이지 않고, 핀란드, 러시아(Karelia) 집단에 비해 유전적 분화가 낮다고 하였다(Nummert et al., 2020). 우리나라의 낙동강 수계와 동해안 지역에서 수집한 잔가시고기(Pungitius kaibarae) 집단들은 Fu’s Fs와 Tajima’s D 모두 유의적이지 않았으나(Kim et al., 2023), 서남부 지역 남생이(Mauremys reevesii) 집단들은 양의 값이고, 유의적 수준을 보여 유전적 병목현상이나 균형선택이 진행 중으로 추정하였다(Moon et al., 2025). 병목현상이나 균형선택은 규모가 작고, 지리적으로 격리되어 유전적 흐름이 차단된 집단에서 주로 발생하기 때문에(Tajima, 1989; Fu, 1997), 단산 지역 개체군의 성장과 관련한 모니터링과 유전적 다양성 분석이 지속되어야 한다.
소백산 집단에서 결정된 D-loop 서열들과 선행연구에서 보고된 서열들을 종합했을 때, 총 23개의 단상체형들(SK01-SK23)이 발견되었다. 선행연구에서 강원도 강릉(MH212330)과 평창(MW722950), 전라남도 구례(MW722951)의 서열들과 장소에 대한 정보가 없는 서열들(MT430951, NC_019612)은 모두 개별 단상체형으로 구분되었고, 소백산 집단들과 일치하지 않았다. 이들 중 2개 지역 이상에서 공통으로 검출된 단상체형은 5개(SK01, SK05, SK10, SK12, SK15)였으며, 나머지 18개는 지역-고유 단상체형이었다. SK05, SK10, SK12는 소백산 내 단산-고치령-단산 지역에서 모두 발견되었고, SK01은 고치령-삼가, SK15는 단산-삼가에서 공통이었다. 지리적 거리가 가까운 단산-고치령 사이에서는 3가지 단상체형이 공통으로 관찰되나, 상대적으로 거리가 먼 삼가-단산, 삼가-고치령은 4가지 단상체형을 공유하였다(Table 3).
단상체형 분포 분석결과는 소백산 내 3개 지역 집단들이 모계가 서로 유전적으로 연관되어 있지만, 고유 단상체형이 많다는 점은 지역별 단상체형 형성과정이 서로 일치하지 않는다는 점을 나타낸다. 대부분의 야생동물 집단의 단상체형 분포는 난자형성과정에서 돌연변이가 생긴 후 모계를 통해 다음 세대로 전달되는 유전적 분화(Brown et al., 1979; Giles et al., 1980; Clayton, 1982), 외부 집단과의 교류에 의한 유입/유출, 또는 분화와 교류가 동시에 적용되어 형성된 사례(Larson et al., 2005; De Jong et al., 2011; Sexton et al., 2014), 이외에도 지리적 장벽, 환경에 대한 적응, 빙하기와 같은 역사적인 과정에 의한 변형 등 다양한 경로로 해석되고 있다(Oshida et al., 2005; Pope et al., 2006; Li et al., 2013; Meng et al., 2021). 소백산은 백두대간 중간에 위치하며, 동북쪽은 강원도 영월군, 북쪽과 서쪽은 충청북도 단양군, 동쪽은 경상북도 봉화군, 남쪽은 경상북도 영주시와 접하고 있다. 이들 지역들은 모두 하늘다람쥐가 서식할 수 있는 높은 산림지역을 포함하고 있어(Won, 1967; Yoon et al., 2004; Kim, 2010; Lim et al., 2021), 외부 산림들과 소백산은 생태적으로 연결되어 있다고 볼 수 있다(Fig. 1). 하지만 소백산 집단은 적어도 강원도 강릉과 평창, 전라남도 구례에서 보고된 서열들과는 다른 모계 집단이었다. 단상체형 분포와 형성과정을 보다 명확하게 이해하기 위해서는 유전자 흐름을 보다 면밀하게 파악할 수 있는 핵 DNA 정보가 필요하며, 또한 소백산 인근 여러 지역의 유전자 정보가 필요하다고 하겠다.
Table 3. Haplotype distribution of the mtDNA D-loop sequences in the Sobaeksan NP and other locations reported in South Korea
테이블
Sobaeksan NP indicates the Sobaeksan National Park, South Korea.
소백산 3개 집단과 선행연구에서 보고된 강원도(강릉, 평창) 집단 사이에서 유전적 분화를 조사하였다. 소백산 집단들 사이에서 유전적 분화 지수 Fst 값은 0.044 (단산-삼가)-0.117 (고치령-삼가)의 범위였으며, 이주율 Nm 값은 1.89 (고치령-삼가)-5.43 (단산-삼가)의 범위였다. 강원도 집단과 비교했을 때 Fst는 0.196 (삼가-강원도)-0.227 (고치령-강원도), Nm은 0.85 (고치령-강원도)-1.03 (삼가-강원도) 수준이었다(Table 4). 이러한 결과는 소백산 내 집단들은 유전적 분화(Fst<0.10)가 적고, 이주율이 높은(Nm>3.0) 관계이나, 삼가-고치령은 Fst>0.1, Nm<2.0을 나타내어 상대적으로 유전적 교류가 적다고 할 수 있다. 단산 집단은 삼가, 고치령 집단들과 Fst<0.1, Nm>2.0을 나타내어 이들 지역들과는 유전적 분화가 낮고, 이주율이 높아 유전자 흐름이 매우 활발한 집단으로 판단된다. 한편, 강원도 집단은 소백산 3개 지역과 Fst>0.15, Nm<1.5를 나타내어, 유전적 분화가 심하고, 이주율이 낮아 유전자의 흐름이 매우 제한적이라고 판독된다(Wright, 1951; Hudson et al., 1992).
유전적 분화와 유전자 흐름이 소백산-강원도 사이에서 현저한 차이를 보이는 점은 집단 간 유전적 분화와 유전자 흐름이 지리적 거리에 의한 분리와 상관있음을 보여주고 있다. 하지만, 소백산 내부에서는 지리적으로 멀리 떨어져 있는 단산-삼가의 Fst가 0.044로 가장 낮고, Nm는 5.43으로 가장 높은 수준이라는 점에서 유전적 분화와 유전자 흐름이 단순히 지리적 거리에 의해 결정되는 것이 아니라, 다른 요인들도 영향을 준다는 선행연구 결과들을 지지하고 있다. 호주의 멸종위기종 Hoplocephalus bungaroides는 집단 간 유전적 분화가 적고, 지리적 거리에 비례한다고 보고되었다(Dubey et al., 2011). 반면, 바하마에 서식하는 박쥐류인 Tadarida brasiliensis는 섬 지역 사이에서 유전적 연속성이 적고, 유전적 분화가 매우 높은 수준이어서, 섬 지역들 사이에서 유전적으로 단절되어 있다고 하였다(Speer et al., 2017). 포유동물뿐만 아니라 조류와 곤충까지도 울창한 산림과 협곡 등 지리적 장벽이 유전자 흐름에 영향을 준다고 하였고(Matthee and Robinson 1997; Lehmann et al. 2000; Girman et al., 2001), 반대로 중국의 tufted deer의 높은 이주율은 Yangtze River가 집단 간 유전자 흐름에 장벽이 되지 않는다는 근거로 제안되었다(Wang et al. 2025). 결론적으로 소백산 하늘다람쥐 지역 집단들은 모계가 유전적 연관성이 나타내지만, 단상체형의 분포는 거리적 거리뿐만 아니라 다른 요인들에 의한 영향도 고려되어야 하며, 이를 설명할 수 있는 추가 연구가 뒤따라야 할 것이다.
Table 4. Genetic differentiation (Fst, lower left) and migration rate (Nm, upper right) among D-loop haplotypes of the Eurasian flying squirrels in Sobaeksan NP and other locations from South Korea
테이블
Sobaeksan NP indicates the Sobaeksan National Park, South Korea.
Tamura and Nei 방식(1993)으로 산출한 단상체형 간 유전적 거리지수는 최소 0.0030 (SK01-SK02, SK12-SK14, SK15-SK16, SK06SK19)에서 최고 0.0838 (SK06-SK23)을 나타내었다(자료 미기재). 한편, 집단 간 유전적 거리지수는 소백산 내에서 0.0273-0.0321로 소백산-강원도(0.0422-0.0495), 소백산-전라남도(0.0513-0.0551)보다 낮았다(Table 5). 계통 유연관계 분석 결과에서 소백산 집단의 단상체형 18개와 선행연구에서 보고된 5개 서열들은 계통수 상에서 6개 아계통군(subclade I-VI)으로 구분되었고, 전체적으로 우리나라 계통군(South Korea clade)에 위치하였다. 반면, 핀란드, 에스토니아, 러시아(Karelia)에서 보고된 D-loop 서열들은 북서 유럽 계통군(Northwestern Europe clade)을 형성하였고, 우리나라의 서열들과는 뚜렷하게 분지되었다. 분지 순서는 아계통군 VI이 공통조상에 더 가깝고, 아계통군 VI와 나머지 단상체형들의 분지는 100% 지지도를 보였다. 우리나라 아계통군 대부분(II-IV)은 북서 유럽 계통군에 비해 먼저 분지된 양상을 보였고, 아계통군들 아계통군 I, II는 북서 유럽 계통군과 동일한 분지점에서부터 분지되었다(Fig. 2). 이러한 결과들은 Oshida et al. (2005), Lee et al. (2008b)의 연구보고와 유사한 양상이었고, Oshia et al. (2015)이 제안한 유라시아-일본(Hokkaido)은 약 20-40만 년 전, 극동아시아-북서 유라시아 집단들이 약 7-14만 년 전에 분지되었을 가능성에 대한 가설을 지지하고 있다.
Table 5. Estimates of evolutionary genetic distance (lower left) and standard error (upper right) over sequence pairs between populations
테이블
Sobaeksan NP indicates the Sobaeksan National Park, South Korea.
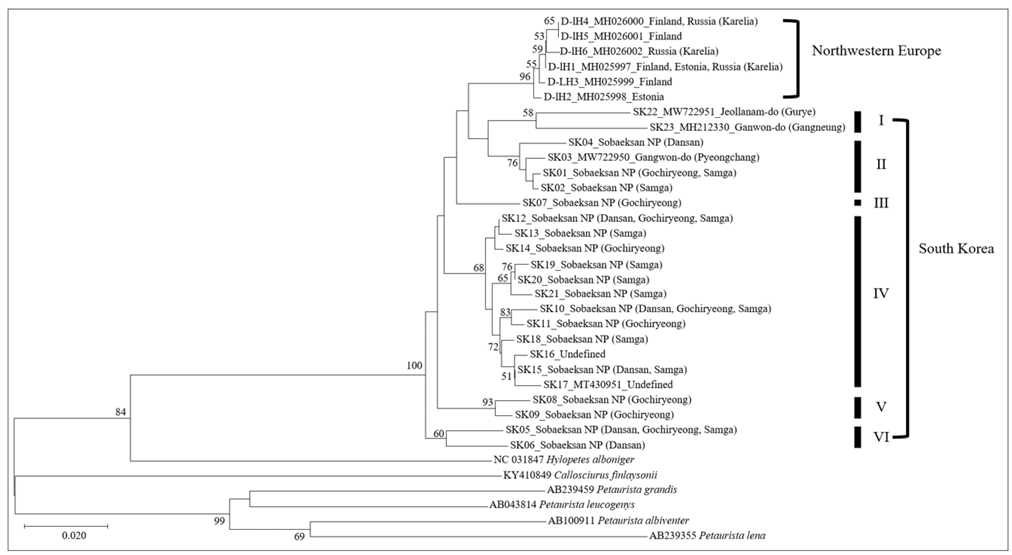
Figure 2. Neighbor-joining tree of phylogenetic relationships among the 23 D-loop haplotypes (SK01-SK23) identified from the Eurasian flying squirrels (Pteromys volans) in South Korea, based on the Tamura and Nei (1993) with gamma distribution model. Numbers represents bootstrap p values (1000 bootstrap replications). The Bootstrap supports are given on branches. Haplotype codes are given in the Table 3. D-loop haplotypes D-lH1 to D-lH6 were designated by Nummert et al. (2020). Sobaeksan NP is the Sobaeksan National Park, South Korea.
이번 연구에서 소백산 집단(52개체)에서 18개의 단상체형이 검출되어, 북서 유럽(130개체)의 단상체형 6개(Nummert et al., 2020)보다 많았다. 이 결과는 cytB 유전자에 대한 연구결과들(Oshida et al., 2005; Lee et al., 2008b)과도 유사한 양상이었다. Oshida et al. (2005)은 우리나라-동북아시아 32개체에서 단상체형 29개, Lee et al. (2008b)은 우리나라의 14개체에서 단상체형 7개, 중국-극동 러시아 12개체에서 단상체형 10개를 보고하였다. 이처럼 시료 수에 비해 상대적으로 많은 단상체형이 발견되는 결과는 시료들의 모계 연관성이 매우 낮음을 나타내며, 한 지역에서 오랜 시간동안 진화하면서, 다른 지역과의 유전자 교류가 매우 빈번했음을 의미한다(Brown et al., 1979; Clayton, 1982). 이 결과들과 비슷한 양상은 동북아시아에서 청설모, 다람쥐, 등줄쥐, 흰넓적다리붉은쥐 등 여러 설치류에서도 보고되었다(Oshida et al., 2005; Koh et al., 2006; Kim and Park 2015a, b; Lee et al., 2008a, b). 이상의 결과들은 우리나라, 일본을 포함한 동북아시아 지역이 제4빙하기 동안 포유동물의 피난처로써 빙하기-간빙기 동안 다양한 유전자형을 보유한 개체들이 유입되었고, 유입 이후 지역적인 진화적 분화가 활발히 진행된 것으로 추정하고 있다(Oshida et al., 2005; Sakka et al., 2010; Oh et al., 2013). 결론적으로 소백산 집단을 포함한 우리나라 하늘다람쥐 집단 역시 아시아 대륙에서 여러 차례에 걸쳐 모계들이 유입되었고, 유입된 모계들이 오랜 시간동안 진화적 분화를 진행해 왔다고 해석된다.
소백산 내 3개 지역(삼가, 단산, 고치령)에서 수집한 하늘다람쥐 분변을 이용한 비침습적 유전자 분석으로 집단의 유전적 다양성과 지역 간 분화, 유전자 흐름과 계통 유연관계을 분석하였다. 각 집단들은 높은 수준의 유전적 다양성을 나타내는 안정적인 상태였으며, 지역 간 공통 단상체형들이 출현하여 3개의 집단들이 서로 모계에 의해 연관된 형태임을 확인하였다. 이들 중 단산지역은 다른 지역들과 연관성이 높아 중요한 역할을 담당하는 것으로 추정되었다. 소백산 집단과 강원도 집단은 모계의 유전적 분화가 심하고 유전자 흐름도 낮은 상태였다. 계통 유연관계 분석결과는 소백산 집단의 단상체형들이 우리나라 계통군에 속하고, 여러 아계통군을 형성한다는 점에서 이들 단상체형들은 오랜 시간동안 여러 차례에 걸쳐 유입되었고, 유입된 이후에 또 다시 지역적 분화를 통한 진화가 진행되어왔음을 나타내었다. 향후 이번 조사 지역뿐만 아니라 소백산 인근과 다른 지역의 시료들을 수집하여 유전적 다양성, 지역 간 유전자 흐름과 연결성, 계통 유연관계 등을 파악한다면, 집단의 형성과정과 진화적 분화, 개체군의 유전적 안정성 등을 설명할 수 있는 좋은 설명 자료가 될 것이다. 특히, MS 마커 분석을 보완하면, 지역별로 현재 서식하고 있는 최소 개체수, 연도별 변동, 지역 간 이동 여부 등을 판독할 수 있을 것이다. 이번 연구결과는 멸종위기인 하늘다람쥐의 비침습적 서식 확인과 개체수 산출을 통한 지속적 자연 서식지 발굴과 유전적 안정성 평가, 지역 간 유전적 상관성 해석에 있어 매우 중요한 정보를 제공하고 있다. 또한 개체와 서식지 보호, 유전적 다양성 증진과 서식지 간 연결성 확보 등 관리방안 마련에 필수적인 자료가 될 것이다.
No potential conflict of interest relevant to this article is reported.
이 연구는 환경부의 지원을 받아 국립공원공단 국립공원야생생물보전원의 “생물자원보전시설운영사업”에 의해 수행된 결과에 일부입니다. 연구비 지원에 감사드립니다.
Brown WM, George MJr, Wilson AC. 1979. Rapid evolution of animal mitochondrial DNA. Proceeding of the National Academy of Science of the United States of America 76(4):1967-1971.
Caragiulo A, Dias-Freedman I, Clark JA, Rabinowitz S, Amato G. 2014. Mitochondrial DNA sequence variation and phylogeography of Neotropic pumas (Puma concolor). Mitochondrial DNA 25(4):304-312.
Cho HJ, Kim DH, Kang TH, Kim IK, Lee JW. 2013. Basic research on the habitat characteristics of endangered species Pteromys volans. Korean Journal of Environment and Ecology 27(5):544-549.
Cultural Heritage Administration. 2006. Yearbook of Cultural Heritage 2006. Seoul, Republic of Korea. Cultural Heritage Administration, pp.51-59.
Clayton DA. 1982. Replication of animal mitochondrial DNA. Cell 28:693-705.
De Jong MA, Wahlberg N, van Eijk M, Brakefield PM, Zwaan BJ. 2011. Mitochondrial DNA signature for range-wide populations of Bicyclus anynana suggests a rapid expansion from recent refugia. PLoS One 6(6):e21385.
DeSalle R, Amato G. 2004. The expansion of conservation genetics. Nature Reviews Genetics 5(9):702-712.
Dubey S, Summer J, Pike DA, Keogh JS, Webb JK, Shine R. 2011. Genetic connectivity among populations of an endangered snake species from southeastern Australia (Hoplocephalus bungaroides, Elapidae). Ecology and Evolution 1:218-227.
Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39:783-791.
Fu YX. 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics 147(2):915-925.
Ghildiyal K, Nayak SS, Rajawat D, Sharma A, Chhotaray S, Bhushan B, Dutt T, Panigrahi M. 2023. Genomic insights into the conservation of wild and domestic animal diversity: A review. Gene 886:147719.
Giles RE, Blanc H, Cann HM, Wallace DC. 1980. Maternal inheritance of human mitochondrial DNA. Proceeding of the National Academy of Science of the United States of America 77:6715-6719.
Girman DJ, Vila C, Geffen E, Creel S, Millis MGL, McNutt JW, Ginsberg J, Kat PW, Mamiya KN, Wayne RK. 2001. Patterns of populations subdivision, gene flow and genetic variability in the African wild dog (Lycaon pictus). Molecular Ecology 10:1703-1723.
Gonzalez-Oliver A, Pineda-Vazquez D, Garfias-Morales E, La Cruz-Laina I, Medrano-Gonzalez L, Marquez-Morfin L, Ortega-Munoz A. 2018. Genetic overview of the Maya populations: mitochondrial DNA haplogroups. Human Biology 90(4):281-300.
Hajkova P, Zemanova B, Bryja J, Hjek B, Roche K, Tkadlec E, Zima J. 2006. Factors affecting success of PCR amplification of microsatellite loci from otter faeces. Molecular Ecology Notes 6:559-562.
Han CW, Lim SJ, Park HB, Park YC. 2018. Seasonal characteristics of fecal sites of the Siberian flying squirrel Pteromys volans. Journal of Forest and Environmental Science 34(2):184-187.
Harris DJ, Arnold EN, Thomas RH. 1998. Relationships of lacertid lizards (Reptilia: Lacertidae) estimated from mitochondrial DNA sequences and morphology. Proceeding of the Royal Society B Biological Sciences 265:1939-1948.
Hwang JY, Cho GJ. 2018. Identification of novel haplotypes and interpretation of gene flow of mitochondrial DNA control region of Eurasian otter (Lutra lutra) for the effective conservation. The Journal of Veterinary Medical Science 80(11):1791-1800
Hudson RR, Slatkin M, Maddison WP. 1992. Estimation of levels of gene flow from DNA sequence data. Genetics 132:583-589.
Ibouroi MT, Arnal V, Cheha A, Dhurham SAO, Montgelard C, Besnard A. 2021. Noninvasive genetic sampling for flying foxes: a valuable method for monitoring demographic parameters. Ecosphere 12(7):e03327.
Iverson JB, Brown RM, Akre TS, Near TJ, Le M, Thomson RC, Starkey DE. 2007. In search of the tree of life for turtles. Chelonian Research Monographs 4:85-106.
Kalinowski ST, Taper M, Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology 16:1099-1106.
Kim EK. 2010. Terrestrial mammalia of the Korea national parks in Baekdudaegan. Journal of National Park Research 1(2):104-107.
Kim HR, Park YC. 2015a. Genetic diversity and genetic structure of the striped field mouse Apodemus agrarius coreae (Muridae, Rodentia) in Korea. Gene 572(2):292-297.
Kim HR, Park YC. 2015b. Genetic isolation of Korean populations of Apodemus peninsulae (Rodentia: Muridae) from their neighboring populations. Genes & Genomics 37:999-1005.
Kim JW. 2018. Study on morphology, ecology and genetics of Siberian flying squirrel (Pteromys volans) in Korea. Ph. D. Thesis, Seoul National University.
Kim KR, Sung MS, Hwang Y, Lee MS, Jeong JH, Kim H, Yu JN. 2023. Analysis of genetic diversity across newly occupied habitats within the Goryeong population of Pungitius kaibarae using the mitochondrial Cytb gene. Korean Journal of Ichthyology 35(4):217-223.
Kim YR, Kim HR, Kim DH, Jang JE, Son JI, Han SH, Lee HJ. 2025. Phylogenetic status and population genetic structure of the long-tailed goral, Naemorhedus caudatus inhabiting the Seoraksan National Park in Korea. Korean Journal of Environmental Biology 43(1):28-41.
Kim YS, Yoo MH, Jung BD, Kim JT. 2010. Genetic diversity in Korean leopard cats (Prionailurus bengalensis euptilura), based on mitochondrial DNA cytochrome b gene sequence analysis. Korean Journal of Veterinary Service 33(4):353-359.
Kobayashi F, Fukui D, Kojima E, Masuda R. 2012. Population genetic structure of the Japanese large-footed bat (Myotis macrodactylus) along three rivers on Hokkaido Island, northern Japan. Mammal Study 37:227-235.
Koh HS, Zhang M, Wang J, Shin DS, Lee BG. 2006. Mitochondrial DNA variation in the red squirrel (Sciurus vulgaris manchuricus) from Korea and northeast China. Acta Theriologica Sinica 26(1):1-7.
Kumar S, Stecher G, Tamura K. 2016. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution 33:1870-1874.
Lampa S, Gruber B, Henle K, Hoehn M. 2008. An optimisation approach to increase DNA amplification success of otter faeces. Conservation Genetics 9:201-210.
Larson G, Dobney K, Albarella U, Fang M, Matisoo-Smith E, Robins J, Lowden S, Finlayson H, Brand T, Willerslev E, Rowley-Conwy P, Andersson L, Cooper A. 2005. Worldwide phylogeography of wild boar reveals multiple centers of pig domestication. Science 307(5715):16181621.
Laxmivandana R, Vashi Y, Kalita D, Banik S, Sahoo NR, Naskar S. 2022. Genetic diversity in mitochondrial DNA D-loop region of indigenous pig breeds of India. Journal of Genetics 101:5.
Lee MY, Lissovsky AA, Park SK, Obolenskaya EV, Dokuchaev NE, Zhang Y, Yu L, Kim YJ, Voloshina I, Myslenkov A, Choi TY, Min MS, Lee H. 2008a. Mitochondrial cytochrome b sequence variations and population structure of Siberian chipmunk (Tamias sibiricus) in Northeastern Asia and Population Substructure in South Korea. Molecules and Cells 26(6):566-575.
Lee MY, Park SK, Hong YJ, Kim YJ, Voloshina I, Myslenkov A, Saveljev AP, Choi TY, Piao RZ, An JH, Lee MH, Lee H, Min MS. 2008b. Mitochondrial genetic diversity and phylogenetic relationships of Siberian flying squirrel (Pteromys volans) populations. Animal Cells and Systems 12:269-277.
Lehmann T, Hawley WA, Grebert H, Danga M, Atieli F, Collins FH. 1999. The Rift Valley complex as a barrier to gene flow for Anopheles gambiae in Kenya. Journal of Heredity 90(6):613-621.
Li L, Wu X, Yu Z. 2013. Genetic diversity and substantial population differentiation in Crassostrea hongkongensis revealed by mitochondrial DNA. Marine Genomics 11:31-37.
Lim SJ, Kim KY, Kim EK, Han CW, Park YC. 2021. Distribution and habitat use of endangered Siberian flying squirrel Pteromys volans (Rodentia: Sciuridae). Journal of Ecology and Environment 45:17.
Matthee CA, Robinson TJ. 1997. Mitochondrial DNA phylogeography and comparative cytogenetics of the spring hare, Pedetes capensis (Mammalia: Rodentia). Journal of Mammalian Evolution 4:53-73.
Mellya RVK, Hopcraft JGC, Mwakilema W, Eblate EM, Mduma S, Mnaya B, Chuma IS, Macha ES, Wambura D, Fyumagwa RD, Kilbride E, Ijaz UZ, Mable BK, Khan A. 2025. Natural dispersal is better than translocation for reducing risks of inbreeding depression in eastern black rhinoceros (Diceros bicornis michaeli). Proceeding of the National Academy of Sciences of the United States of America 122(23):e2414412122.
Meng X, Liu T, Zhang L, Jin L, Sun K, Feng J. 2021. Effects of colonization, geography and environment on genetic divergence in the intermediate leaf-nosed bat, Hipposideros larvatus. Animals 11(3):733.
Ministry of Environment. 2005. Picture Book of Endangered Wild Animals and Plants. Seoul, Republic of Korea. Ministry of Environment. p.78.
Moon KS, Lee YG, Go CH, Kim TW, Lee SH, Yang DH, Han SH, Sung HC. 2025. Genetic diversity based on the mitochondrial cytochrome B gene polymorphisms in the Reeves’ turtle populations in southwestern region of South Korea. Journal of Animal Breeding and Genomics 9(1):39-47.
Mumma MA, Zieminski C, Fuller TK, Mahoney SP, Waits LP. 2015. Evaluating noninvasive genetic sampling techniques to estimate large carnivore abundance. Molecular Ecology Resources 15(5):1133-1144.
Nei M. 1987. Molecular Evolutionary Genetics. New York, NY: Columbia University Press.
Nowak RM. 1999. Walker’s Mammals of the World vol. 2. 6th Ed. Johns Hopkins University Press, Baltimore and London, pp.1247-1250.
Nummert G, Aaspollu A, Kunings K, Timm U, Hanski IK, Maran T. 2020. Genetic diversity in Siberian flying squirrel (Pteromys volans) in its western frontier with a focus on the Estonian population. Mammal Research 65:4.
Oh DJ, Kim TW, Chang MH, Han SH, Oh HS, Kim SJ. 2013. Migration route estimation of the Jeju striped field mouse Apodemus agrarius chejuensis (Rodentia, Muridae). Mitochondrial DNA 24(2):137-144.
Oshida T, Abramov A, Yanagawa H, Masuda R. 2005. Phylogeography of the Russian flying squirrel (Pteromys volans): implication of refugia theory in arboreal small mammal of Eurasia. Molecular Ecology 14(4):1191-1196.
Painter JN, Selonen V, Hanski IK. 2004. Microsatellite loci for the Siberian flying squirrel, Pteromys volans. Molecular Ecology Notes 4:119-124.
Pope LC, Domingo-Roura X, Erven K, Burke T. 2006. Isolation by distance and gene flow in the Eurasian badger (Meles meles) at both a local and broad scale. Molecular Ecology 15:371-385.
Pritchard JK, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data. Genetics 155(2):945-959.
Reed JZ, Tollit DJ, Thompson PM, Amos W. 1997. Molecular scatology: the use of molecular genetic analysis to assign species, sex and individual identity to seal faeces. Molecular Ecology 6:225-264.
Rodriguez-Castro KG, Saranholi BH, Bataglia L, Blanck DV, Galetti PM. 2018. Molecular species identification of scat samples of South American felids and canids. Conservation Genetics Resources 12(1):61-66.
Rogers AR, Harpending H. 1992. Population growth makes waves in the distribution of pairwise genetic differences. Molecular Biology and Evolution 9(3):552-569.
Rozas J, Ferrer-Mata A, Sanchez-DelBarrio JC, Guirao-Rico S, Librado P, Ramos-Onsins SE, Sanchez-Garcia A. 2017. DnaSP v6: DNA sequence polymorphism analysis of large datasets. Molecular Biology and Evolution 34:3299-3302.
Ryu SH, Kwak MJ, Hwang UW. 2013. Complete mitochondrial genome of the Eurasian flying squirrel Pteromys volans (Sciuromorpha, Sciuridae) and revision of rodent phylogeny. Molecular Biology Reports 40(2):1917-1926.
Saitou N, Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution 4:406425.
Sakka H, Quere JP, Kartavtseva I, Marina PK, Chelomina G, Atopkin D, Bogdanov A, Michaux JH. 2010. Comparative phylogeography of four Apodemus species (Mammalia: Rodentia) in the Asian Far East: evidence of Quaternary climate changes in their genetic structure. Biological Journal of the Linnean Society 100(4):797-821.
Saremi NF, Supple MA, Byrne A, Cahill JA, Coutinho LL, Dalén L, Figueiró HV, Warren E. Johnson WE, Milne HJ, O’Brien SJ, O’Connell B, Onorato DP, Riley SPD, Sikich JA, Stahler DR, Villela PMS, Vollmers C, Wayne RK, Eizirik E, Corbett-Detig RB, Green RE, Wilmers CC, Shapiro B. 2019. Puma genomes from North and South America provide insights into the genomic consequences of inbreeding. Nature Communications 10:4769.
Sexton JP, Hangartner SB, Hoffmann AA. 2014. Genetic isolation by environment or distance: which pattern of gene flow is most common? Evolution 68(1):1-15.
Shar S, Lkhagvasuren D, Henttonen H, Maran T, Hanski I. 2016. Pteromys volans (errata version published in 2017). The IUCN Red List of Threatened Species 2016: e.T18702A115144995.https://dx.doi.org/10.2305/IUCN.UK.2016-3.RLTS.T18702A22270935.en. Accessed on 15 August 2025.
Shin HS. 2023. Effectiveness and improvement on wildlife crossing structures of National Park, South Korea. Ph. D. Thesis, Sangji University.
Speer KA, Petronio BJ, Simmons NB, Richey R, Magrini K, Soto-Centeno JA, Reed DL. 2017. Population structure of a widespread bat (Tadarida brasiliensis) in an island system. Ecology and Evolution 7:7587-7598.
Taberlet P, Griffin S, Goossens B, Questiau S, Manceau V, Escaravage N, Waits LP, Bouvet J. 1996. Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic Acids Research 24(16):3189-3194.
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics 123:585-595.
Tamura K, Nei M. 1993. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Molecular Biology and Evolution 10:512-526.
Tende T, Hansson B, Ottosson U, Akesson M, Bensch S. 2014. Individual identification and genetic variation of lions (Panthera leo) from two protected areas in Nigeria. PLoS ONE 9(1):e84288.
Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: improving the sensitivity of progressive sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research 22:4673-4680.
Tian X, Shi L, Bai X, Wang Z. 2025. Genetic analysis of the Siberian flying squirrel population in the northern Changbai Mountains, Northeast China: Insights into population status and conservation. Open Life Science 20(1):20251128.
Untergasser A, Cutcutache I, Koressaar T, Ye J, Faircloth BC, Remm M, Rozen SG. 2012. Primer3 – new capabilities and interfaces. Nucleic Acids Research 40(15):e115.
Wang F, Yang C, Xiong Y, Cui X, Peng J. 2025. Genetic diversity and population structure of tufted deer (Elaphodus cephalophus) in Chongqing, China. Animals 15:2254.
Won C, Smith KG. 1999. History and current status of mammals of the Korean Peninsula. Mammal Review 29:3-33.
Won PH. 1967. Illustrated Encyclopedia of Fauna & Flora of Korea, Vol. 7 Mammals. Seoul, Republic of Korea. Ministry of Education. pp.180-187.
Won PO. 1968. Notes on the first propagating record of the Palaearctic flying squirrel, Pteromys volans aluco (Thomas) from Korea. Journal of The Mammalogical Society of Japan 4:40-43.
Wright S. 1931. Evolution in Mendelian populations. Genetics 16(2):97-159.
Wright S. 1951. The genetical structure of populations. Annals of Eugenics 15(1):323-354.
Xu H, Yang X, Ni W, Liu F, Chen H, Zhu X, Liu X. 2023. Genetic diversity and genetic structure of selected generations of Chinese pond turtle (Mauremys reevesii) based on microsatellite DNA. Journal of Dalian Ocean University 38(5):812-818.
Yoon MH, Han SH, Oh HS, Kim JG. 2004. The Mammals of Korea. Dongbangmedia, Seoul, pp.111-113.
Zittlau KA, Davis CS, Stobeck C. 2000. Characterization of microsatellite loci in northern flying squirrels (Glaucomys sabrinus). Molecular Ecology 9:826-827.