Kwang-Seon Moon1,2, Young-Gu Lee3, In-Yeob Kim4, Tae-Wook Kim5, Sa-Hyun Lee5, Doo-Ha Yang5, Sang-Hyun Han5*, Ha-Cheol Sung6*
1Mudeungsan National Park Nature Center, Korea National Park Service, Gwangju 61146, Republic of Korea
2Department of Biological Science and Biotechnology Graduate School, Chonnam National University, Gwangju 61186, Republic of Korea
3Western Regional Headquarters, Korea National Park Service, Jeonju 54843, Republic of Korea
4Mudeungsan National Park Office, Korea National Park Service, Gwangju 61494, Republic of Korea
5National Park Institute for Wildlife Conservation, Korea National Park Service, Yeongju 36015, Republic of Korea
6Department of Biological Science, Chonnam National University, Gwangju 61186, Republic of Korea
*Correspondence to Sang-Hyun Han, Ha-Cheol Sung E-mail: shcol2002@jnu.ac.kr, hansh04@knps.or.kr
Volume 9, Number 2, Pages 79-90, June 2025.
Journal of Animal Breeding and Genomics 2025, 9(2), 79-90. https://doi.org/10.12972/jabng.2025.9.2.3
Received on June 02, 2025, Revised on June 24, 2025, Accepted on June 24, 2025, Published on June 30, 2025.
Copyright © 2025 Korean Society of Animal Breeding and Genetics.
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0).
To understand the genetic diversity and population structure this study were examined using microsatellite (MS) genotypes in two Reeves’ turtle populations in South Korea. Mean observed and expected heterozygosities and polymorphic information content were 0.555, 0.595 and 0.564 in Geumgok population and 0.550, 0.633 and 0.591 in Gangjin-Yeongam population, respectively. Genetic diversity levels of Korean turtles showed higher than those of Chinese and Taiwanese captive breeding populations but lower than those of two Chinese populations collected from broad geographic range. Among nine MS loci tested, six loci in Geumgok population and two loci in Gangjin-Yeongam population showed significantly departed from Hardy-Weinberg equilibrium (PHW<0.05). Positive inbreeding coefficient (FIS) values were found, estimating that inbreeding occurred. On the other hand, negative FIS estimated to be introgression of exotic gene pools at the past. The genetic structure exhibited a fitting constant K=2, along with separation between both populations. The results from FCA, structure and phylogenetic analyses showed that these populations shared almost alleles except for several individuals but clustered into different branches on the phylogenetic tree, indicating that both populations had the similar ancestral origins but current populations formed by different reproduction processes. Our findings suggest that the two populations have experienced severe genetic bottlenecks in the past and have been growing their populations through a breeding process including inbreeding since the introgression of exotic gene pools. This study will provide important information for developing management programs for enhancing the genetic diversity and the population stability of the endangered Reeves’ turtle in South Korea.
endangered, genetic diversity, population structure, microsatellite, Reeve’s turtle
중형 담수거북인 남생이(Mauremys reevesii) (Reeves’ turtle, Chinese three-keeled pond turtle)는 중국, 대만의 Kimmen Island, 한반도에 서식하고, 일본과 대만 본섬, 홍콩 등 동아시아와 미국에는 도입된 것으로 알려져 있다(Chen and Lue, 2010; Lovich et al., 2011; van Dijk, 2011; Chang et al., 2012). 우리나라에는 제주도를 비롯한 도서지방을 제외한 한반도 전역에 분포하였으나(Song, 2007; NIBR, 2011), 20세기 동안 서식지 파괴, 환경오염, 식용이나 약용으로 이용하기 위한 남획 등 다양한 이유로 개체수가 급격히 감소하여(Lovich et al., 2011; van Dijk, 2011; Kim et al., 2013), 국제자연보전연맹(International Union for the Conservation of Nature, IUCN)의 적색목록의 멸종위기(endangered, EN) 등급, 멸종위기에 처한 야생 동식물종의 국제거래에 관한 협약(Convention on International Trade in Endangered Species of Wild Fauna and Flora, CITES)의 부속서 Ⅲ에 등재되었고(van Dijk, 2011; CITES, 2015), 국내에서는 천연기념물과 멸종위기야생생물 Ⅱ급으로 지정되었다(CHA, 2006; NIBR, 2011; MOE, 2017). 이외에도 1970년대 이후 종교활동, 관상용, 식용, 약용을 목적으로 붉은귀거북(Trachemys scripta elegans) 등 다양한 외래 거북류과 함께 중국 원산의 남생이도 상당수 도입되었고, 일부는 자연생태계에 유출되어 우리나라 거북류의 생존을 위협하고 있다(Koo et al., 2015; Jo et al., 2017). 남생이에 관한 연구는 2000년대 이후 본격적으로 진행되고 있으며, 중국과 대만, 일본 등 동북아시아에서는 남생이를 포함한 육지거북들의 원산지나 종내 또는 종간 교잡을 추적하기 위한 종 식별과 계통유전학 연구들이 주로 진행되었고(Fong and Chen, 2010; Suzuki et al., 2011; Xia et al., 2011), 국내에서는 남생이의 서식지, 성장, 번식과 관련된 연구들이 주로 보고되었다(Kim et al., 2013; Jung et al., 2016; Koo et al., 2015, 2019).
핵 DNA에서 발견되는 미세부수체(microsatellite, MS)는 빠른 돌연변이를 통해 다양한 대립유전자형의 다형성을 나타내어(Weber and Wong 1993; Payseur et al., 2011), 이를 토대로 같은 종 내에서 집단 간 유연관계 해석, 집단의 유전적 혈통 구조에 대한 해석뿐만 아니라, 친자확인, 동일성검사 등 법의학적 분석, 가축의 이력 추적, 야생동물 개체군 크기 측정과 유전적 다양성 평가 등 다양한 분야에서 활용되고 있다(Rosenberg et al., 2002; Lim et al., 2009; Zhao et al., 2017; Kim et al., 2025; Myung et al., 2025). 남생이에서는 MS 마커 분석을 통해 중국 내에서 지역별 집단의 유전적 다양성과 집단 간 분화도가 비교적 높은 수준이지만, 근친도는 지역별로 다르다고 보고되었고(Ye et al., 2009; Bu et al., 2019; Xu et al., 2023), 대만의 자연집단들은 서식지별로 사람 활동에 의한 유전적 교잡이나 역교배가 발생하는 것으로 추정하였다(Lee et al., 2019). 현재까지 우리나라 남생이 집단에 대한 유전자 수준에서의 연구는 미토콘드리아 DNA (mitochondrial DNA, mtDNA) 다형성에 근거하여 한반도 고유 개체와 중국 기원 개체들도 모두 서식함을 확인하였고(Oh et al., 2017), mtDNA와 핵 DNA 유전자 연구를 통해 국내 자연생태계에는 중국에서 도입된 남생이뿐만 아니라 중국줄무늬목거북(M. sinensis)이 발견되며, 심지어 남생이×중국줄무늬목거북의 교잡 후손들도 발견된다고 보고되었다(Baek et al., 2024). 최근 mtDNA cytochrome B 유전자의 다형성을 근거로 유전적 다양성이 비교적 낮은 수준임을 보고하였으나(Moon et al. 2025), 현재까지 부모로부터 물려받은 유전적 특성을 모두 설명할 수 있는 핵 DNA 마커를 이용한 유전적 다양성 연구결과는 보고되지 않았다.
이 연구에서는 우리나라 서남부권에 위치한 광주광역시, 전라남도 영암군-강진군에서 수집한 남생이 두 집단에서 MS 대립유전자형의 다형성에 근거한 유전적 다양성을 살펴보고자 하였다.
연구에 이용한 남생이 시료는 2019년부터 2024년까지 전라남도 영암군과 강진군에서 수집한 강진-영암 집단(n=21), 광주광역시 북구에서 수집한 금곡 집단(n=92)을 이용하였다(Table 1). 유전체 DNA 추출은 수집한 남생이의 등갑을 긁거나 절개하여 얻은 피부 조직에서 DNeasy Blood & Tissue Kit (Qiagen, Hilden, Germany)를 이용하여 공급자의 안내에 따랐고, 추출한 DNA는 중합효소연쇄반응(polymerase chain reaction, PCR)의 주형으로 이용하였다.
Table 1. Genetic diversities, inbreeding coefficient (FIS) and Hardy-Weinberg equilibrium test results for each locus in two Reevesi’ turtle populations in South Korea
| Locus | Geumgok (n=92) | Gangjin-Yeongam (n=21) | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| k | Hobs | Hexp | PIC | FIS | HWE | k | Hobs | Hexp | PIC | FIS | HWE | |||
| PHW | Significance | PHW | Significance | |||||||||||
| Cre9 | 6 | 0.633 | 0.607 | 0.529 | 0.007 | 0.006 | ** | 6 | 0.714 | 0.790 | 0.738 | 0.098 | 0.252 | N.S. |
| Cre14 | 7 | 0.378 | 0.504 | 0.465 | 0.298 | 0.000 | *** | 6 | 0.571 | 0.674 | 0.608 | 0.155 | 0.075 | N.S. |
| Cre22 | 5 | 0.413 | 0.671 | 0.617 | 0.386 | 0.000 | *** | 5 | 0.286 | 0.569 | 0.507 | 0.504 | 0.002 | ** |
| Cre37 | 14 | 0.826 | 0.814 | 0.787 | -0.015 | 0.000 | *** | 10 | 0.857 | 0.895 | 0.862 | -0.044 | 0.129 | N.S. |
| Cre46 | 4 | 0.272 | 0.265 | 0.252 | -0.025 | 0.025 | ** | 3 | 0.238 | 0.220 | 0.199 | -0.081 | 0.224 | N.S. |
| Mdw01 | 6 | 0.413 | 0.419 | 0.394 | 0.015 | 0.119 | N.S. | 7 | 0.667 | 0.591 | 0.546 | -0.131 | 0.881 | N.S. |
| Mdw03 | 10 | 0.791 | 0.869 | 0.849 | 0.089 | 0.002 | ** | 9 | 0.476 | 0.826 | 0.786 | 0.429 | 0.000 | *** |
| Mdw05 | 10 | 0.880 | 0.852 | 0.838 | -0.034 | 0.007 | ** | 10 | 0.905 | 0.875 | 0.836 | -0.035 | 0.238 | N.S. |
| Mdw08 | 2 | 0.391 | 0.354 | 0.354 | 0.105 | 0.158 | N.S. | 3 | 0.238 | 0.260 | 0.237 | 0.087 | 0.338 | N.S. |
| Mean±SD | 7.1±3.7 | 0.555±0.229 | 0.595±0.224 | 0.564±0.220 | 0.070±0.165 | 6.4±2.6 | 0.550±0.258 | 0.633±0.252 | 0.591±0.245 | 0.118±0.218 | ||||
k, number of alleles found; Hobs, observed heterozygosity; Hexp, expected heterozygosity; PIC, polymorphic information contents; HWE, Hardy-Weinberg equilibrium test; PHW, P-value for heterozygote deficit for Hardy-Weinberg test.
1, *, **, and *** indicate significantly different at PHW<0.05, PHW<0.01, and PHW<0.001, respectively; N.S., not significant.
개체별 핵 DNA 유전자형 정보를 얻기 위해 Ye et al. (2009)과 Liu et al. (2012)에 의해 제안된 15종의 MS 마커 중 사전 시험을 통해 최종 9종(Cre9, Cre14, Cre22, Cre37, Cre46, Mclw01, Mclw03, Mclw05, Mclw08)을 선정하여 대립유전자형을 결정하였다. 각각의 유전자 좌위에 대한 순방향 프라이머는 FAM, NED, VIC, PET으로 표지하였다. PCR은 주형 DNA와 프라이머를 Multiplex Plus PCR Kit (Qiagen, Hilden, Germany)와 혼합하여 10 ul로 반응하였고, Mastercycler Nexus Gradient (Eppendorf, Hamburg, Germany)를 이용하여 touch-down PCR 기법으로 증폭하였다. PCR 반응의 초기 변성은 94℃에서 5분, 프라이머-DNA 결합은 65℃에서 55℃까지, 가닥 신장 72℃에서 1분으로 이어지는 touch-down cycle을 10회 수행한 후, 94℃ 1분, 55℃ 1분, 72℃ 1분으로 이어지는 연쇄반응을 30회 추가하고, 72℃에서 10분간 최종 신장하였다. 증폭이 끝난 PCR 산물은 1.5% agarose 겔 상에서 전기영동하여 확인하고, 5-10배 희석한 후 GeneScan 500 LIZ dye Size Standard (Life Technologies Ltd., Woolston, WA, UK)와 혼합하여 자동염기서열 분석장치 ABI Automated DNA Sequencer 3730XL (Applied Biosystems, Foster City, CA)을 이용하여 전개하였다. 대립유전자형은 GeneMapper ver. 5 (Applied Biosystems, Foster City, CA) 프로그램을 이용하여 PCR 산물의 길이와 형광표지자의 종류별로 분류하였다.
각각의 유전자 좌위에서 발견된 대립유전자형을 근거로 남생이 집단의 유전적 다양성을 분석하였다. 대립유전자의 수(k), 관찰이형접합율(observed heterozygosity, Hobs), 기대이형접합율(expected heterozygosity, Hexp), 다형정보량(polymorphic information contents, PIC)은 CERVUS ver. 3.0.3 (Kalinowski et al., 2007)으로 분석하였다. Lee et al. (2019)에 의해 보고된 대만 남생이 집단(n=9)과 중국줄무늬목거북(n=11)의 MS 대립유전자형은 저자의 자료를 CERVUS 프로그램으로 분석하였다. 분석 프로그램 간 입력자료의 변환은 Microsatellite Analyser (MSA) ver. 4.05 (Dierginger and Schlotterer, 2003)를 이용하였다. Weir and Hill (2002) 추정법에 의한 근교계수(FIS)는 FSTAT ver 2.9.4 (Goudet, 2001)과 GENEPOP ver. 4.7.3 (Raymond and Rousset, 1995)을 사용하여 계산하였다. 유전적 평형은 GENEPOP을 이용하여 MS 마커 대립유전자형의 분포에 대한 최소 기대빈도, χ2 값, P 값을 산출하고 Hardy-Weinberg test를 수행하였다. 집단의 유전적 구조 분석은 StructureSelector (Li and Liu, 2018)를 이용하여 Bayesian 접근법으로 K=2-10으로 설정하고 Evanno et al. (2005)의 방법으로 최적화된 ΔK 값을 확인하고 STRUCTURE HARVESTER (Earl and Von-Holdt, 2012)으로 도식화하였다. 집단 간 유연관계 분석은 GENETIX version 4.05 (Belkhir et al., 2004)로 개체별 요인대응분석(factorial correspondence analysis, FCA)을 수행하였다. MS 대립유전자형을 근거로 개체 간 근연관계를 확인하기 위해 MSA로 변환한 자료를 PHYLIP ver. 3.698 (Felsenstein, 2004)을 이용하여 neighbor-joining (NJ) (Saitou and Nei, 1987)의 방법으로 계통수를 작성하고 SplitsTree 5 (Hudson and Bryant, 2006)로 도식화하였다.
남생이 두 집단(금곡, 강진-영암)에서 다형성을 나타내는 MS 유전자 좌위를 이용하여 유전적 다양성을 확인하였다(Table 1). 금곡 집단은 마커의 대립유전자(k)가 7.1±3.7개 발견되었고, 관찰이형접합율(Hobs)은 0.272-0.880 (0.555±0.229), 기대이형접합율(Hexp)은 0.265-0.869 (0.595±0.224), 다형정보량(PIC)은 0.252-0.849 (0.564±0.272)의 범위를 나타내었고, 강진-영암 집단은 k=6.4±2.6개, Hobs는 0.238-0.905 (0.550±0.258), Hexp는 00.220-0.0.895 (0.633±0.252), PIC는 0.199-0.862 (0.591±0.245)의 범위를 나타내었다. 금곡 집단이 강진-영암 집단보다 대립유전자의 수는 많고, Hobs는 높게 나타났으나, Hexp, PIC는 강진-영암 집단이 다소 높은 수준을 보였다. 야생동물에서 집단의 크기는 이형접합율 등 유전적 다양성과 높은 상관성을 나타낸다(Frankham, 1996; Pruett and Winker, 2008). 하지만 이번 연구에서 금곡 집단은 강진-영암 집단보다 4배 이상 크지만, 금곡 집단이 Hobs가 다소 높으나, Hexp, PIC는 강진-영암 집단이 더 높았다. 이는 남생이 집단의 유전적 다양성이 집단의 크기 이외의 요인에 의해 영향을 받고 있음을 추정할 수 있겠다. Moon et al. (2025)은 금곡, 강진-영암 집단 모두 중국 기원 남생이들이 함께 발견되고, 특히 강진-영암 집단은 전체의 약 36%는 중국 기원으로, 금곡 집단(26.1%)보다 많았다. 이 결과는 두 집단에 비교적 최근에 유입된 외래 유전자 공급원(gene pool)에 의해 유전적 다양성이 영향을 받았을 가능성을 보여주고 있음을 추정할 수 있다. 반면, Bao et al. (2007)은 집단 내 Hobs가 집단 크기에 강한 영향을 받는다고 하였는데, 이번 연구결과도 Hobs는 집단의 크기가 큰 금곡 집단에서 더 높은 수준을 보였다. PIC는 집단의 유전적 다양성을 평가하는 데 있어 핵심적인 요인으로 간주하며(Botstein et al., 1980), 금곡 집단과 강진-영암 집단은 모두 0.5 이상인 높은 수준의 유전적 다양성을 보였다.
개체군의 지역적인 격리나 유전적 병목현상을 거친 개체군 밀도가 낮은 야생동물에서의 근친교배이나 유전적 흐름의 저하가 이형접합율의 소실의 주된 원인으로 간주되고 있다. 미국 California 해달 집단은 18-19세기 동안 모피용 사냥으로 인한 유전적 병목현상을 거친 이후 지역 집단 내에서 근친교배가 발생함에 따라 Hexp에 비해 Hobs가 낮은 수준을 보이고(Lidicker and McCollum, 1997), Arizona의 야생 집쥐(Mus musculus domesticus) 집단은 약 45%의 유전자 좌위에서 관찰되는 이형접합체 결핍은 집단 내 근친교배, 다른 집단과의 낮은 유전자 흐름으로 인해 것으로 기술되었다(Laurie et al., 2007). 스코틀랜드의 St Kilda 군도에 분포하는 야생 양 집단에서의 낮은 이형접합율은 지리적 격리로 인해 발생한 높은 근친교배와 낮은 개체군 밀도로 인한 것으로 분석되었다(Pemberton et al., 2017). 반면, 외래 유전자 공급원의 유입에 의한 Hobs의 증가는 외부 유전자원의 유입이나 이형접합체 우위(heterozygote advantage)에 의한 결과로 알려져 있다. 인도양에 위치한 프랑스령 Kerguelen 제도에 도입된 mouflon (Ovis orientalis musimon) 개체군은 단 한 쌍으로 시작하여 수 십 년 동안 개체수가 지속적으로 증가했으나, Hobs는 Hexp보다 더 높은 양상을 보였는데, 이는 외래 유전자원의 유입에 의해 Hobs의 초과적인 증가는 이루어진 것으로 제안하였다다(Kaeuffer et al., 2007). 슬로바키아의 야생 붉은사슴(Cervus elaphus)에서 관찰된 높은 Hobs 역시 외래 유전자 유입이 주된 요인으로 제안되었다(Moravcikova et al., 2023). 한편, 핀란드에 도입된 흰꼬리사슴(Odocoileus virginianus) 집단은 창시자가 암컷 3마리와 수컷 1마리에서 시작되었고 외래 유전자원의 유입이 없음에도 불구하고 수 십 년 후 개체수의 증가와 함께 Hobs와 Hexp가 일치하는 수준을 보였는데, 이는 선택적인 이형접합체 우위에 의한 결과로 추정하였다(Kekkonen et al., 2012).
금곡과 강진-영암 두 집단에서 유전적 평형을 HWE 시험으로 평가하고, MS 마커 대립유전자형 분포에 따른 근교계수(FIS)를 산출하였다(Table 1). 금곡 집단은 시험된 마커 9개 중 6개(Cre9, Cre14, Cre22, Cre37, Mclw03, Mclw05)에서 대립유전자형의 분포가 HWE 시험에서 유의적인 차이를 나타내었고(PHW<0.05), 나머지 3개의 마커는 유의적인 차이가 확인되지 않았다(PHW>0.05). 이들 중 Cre14, Cre22, Mclw03 마커들에서는 관찰이형접합 빈도가 기대이형접합 빈도보다 유의적으로 낮았고(Hobs<Hexp) (PHW<0.05), 근계계수 FIS가 Cre14 0.298, Cre22 0.386, Mclw03 0.102로 0.0보다 큰 양의 값을 나타내었다. 한편, 강진-영암 집단은 2개의 마커(Cre22, Mclw03)에서만 대립유전자형의 분포가 HWE 시험에서 유의적인 차이를 나타내었고(PHW<0.05), 나머지 7개의 마커는 유의적인 차이가 확인되지 않았다(PHW>0.05). 이들 중 Cre22, Mclw03 마커들에서는 관찰이형접합 빈도가 기대이형접합 빈도보다 유의적으로 낮았고(Hobs<Hexp) (PHW<0.05), FIS가 0.0보다 매우 큰 양의 값(Cre22 0.0504, Mclw03 0.429)을 나타내었다. 일반적으로 개체군 내에서 Hobs가 Hexp보다 낮고, FIS가 0.0보다 높은 양(+)의 값을 나타내면, 근친교배가 활발하거나, 집단 구조화로 인해 유전자 흐름이 제한된 것으로 간주한다. 반대로 Hobs가 Hexp보다 높고, FIS가 0.0보다 낮은 음(-)의 값을 나타내면, 외부 개체군과의 교류 또는 유전자 공급원의 유입을 의미한다고 하였다(Wright 1951, 1965; Hedrick, 2005; Parreira and Chikhi, 2015). 금곡 집단은 HWE가 분석된 9개의 MS 중에서 6개에서 유의적인 차이를 보이고(PHW<0.05), Cre14, Cre22의 FIS가 0.298, 0.386으로 다소 높은 양의 값을 보이므로, 집단 내 근친교배에 의한 유전적 불평형과 함께 다양성이 감소하고 있다고 하겠다. 반면, Cre37, Mclw05는 Hobs>Hexp (PHW<0.05), FIS<0.0인 음의 값을 나타내어, 외부 개체군과의 교류 또는 외래 유전자 공급원의 유입을 생각할 수 있겠다. 전체적인 FIS가 0.070±0.165를 나타내어 외부 유전자원의 유입뿐만 아니라 근친교배에 의해 개체군이 성장하고 있는 것으로 보인다. 강진-영암 집단 역시 HWE 시험에서 유의적인 차이를 나타낸 MS 마커 2개(Cre22, Mclw03)에서 FIS가 높은 양의 값을 보이므로, 근친교배가 발생하고 있는 것으로 추정된다. 하지만, 일부 MS 마커들에서 FIS가 음의 값을 보이는 부분은 외부 유전자 공급원의 유입도 있었던 것으로 추정된다. 전반적으로 금곡 집단과 강진-영암 집단 모두 20세기 동안 병목현상으로 개체군 크기가 감소한 이후, 사람의 활동에 의한 외래 개체들(중국 기원 남생이)이 유입되었고, 근친교배를 포함한 번식활동을 통해 개체군이 성장하고 있는 단계로 파악되며, 이 결과들은 Moon et al. (2025)의 연구결과에서 제안한 중국 기원의 남생이들이 유입되었거나, 근처 다른 집단과의 혼합에 의한 결과와 유사한 결과라 하겠다.
전 세계적으로 남생이가 자연적으로 서식하고 있는 지역은 중국, 대만, 한반도에 국한되어 있다. 이번 연구에서 확인된 우리나라 남생이 두 집단의 유전적 다양성과 근교계수를 중국, 대만 집단에서 보고된 결과들(Bu et al., 2019; Ye et al., 2019; Xu et al., 2023)과 비교하였다(Table 2). 이 중 Z-J-S (Zhejiang-Jiangxi-Shanghai) 집단은 중국의 중남부 3개 성 지역에서, H-A 집단은 중국 중부의 HubeiAnhui 2개 성 지역에서 수집되어 상대적으로 넓은 분포에서 수집된 집단들이며, 반면 다른 집단들은 도시 단위로 수집된 집단이다(Bu et al., 2019; Ye et al., 2019; Xu et al., 2023). MS 마커에 대한 대립유전자의 수는 Z-J-S 집단이 k=10.9로 우리나라 k=9.3보다 많았고, 다른 집단들은 우리나라보다 적었다. Hobs가 우리나라(0.554)보다 높은 곳은 Z-J-S (0.708), Guangzhou (0.568), Kinmen (0.572) 등이었고, Hexp는 Z-J-S (0.815), Guangzhou (0.717), Nanning (0.640) 등이 우리나라(0.629)보다 높았고, PIC의 수준도 자료가 제시되지 않은 Z-J-S 집단을 제외하면, Guangzhou 집단은 0.664로 우리나라 집단보다 높은 수준이었으나, 다른 지역 집단들은 0.441-0.541로 낮은 수준을 나타내었다. FIS는 개체수가 적어서 산출되지 않은 대만 Kinmen 집단을 제외한 중국의 모든 집단들(FIS0.119-0.285)이 우리나라(0.105)에 비해 높은 수준을 나타내었다. 전체적으로 분석 시료의 수집 면적이 넓은 집단들(Z-J-S, Guangzhou)의 유전적 다양성이 더 높은 양상을 보였고, 이는 서식지 면적이 넓을수록 높은 유전적 다양성을 나타낸다는 기존의 연구결과(Frankham, 1996)에 일치하는 경향이라 하겠다.
우리나라 남생이 집단의 유전적 다양성은 중국줄무늬목거북(M. sinensis)에 대한 연구결과(Lee et al., 2019)보다 높으나, 노랑연못거북(M. mutica) (Zhang et al., 2010), 붉은바다거북(Caretta caretta) (Monzon-Arguello et al., 2008)보다 낮은 수준을 보였다. 대만에서 중국줄무늬목거북은 1998년 처음 기록되었으나, 최근 수십 년 동안 이후 사람에 의한 도입과 사육, 자연계 방사 등에 의해 남생이 자연 개체군에 위협을 초래하고 있다고 알려져 있다(Chen and Lue, 2010; Lin et al., 2015; Lee et al., 2019). 따라서 대만에서 수집된 중국줄무늬목거북의 낮은 유전적 다양성은 중국으로부터 유입된 소수 개체군에서 증식된 개체들이 자연생태계로 유입된 데에서 기인한 결과로 추정된다. 해양성 거북류인 붉은바다거북 집단은 Canary Islands 등 다양한 지역에서 수집된 개체들에 대한 시험결과이므로, 유전적 다양성의 높게 나타냈으며(Monzon-Arguello et al., 2008), 이는 유전적 다양성과 서식 면적이 비례적인 관계라는 기존 연구결과(Frankham, 1996)와 일치한다. 결과적으로 우리나라 남생이 집단의 유전적 다양성은 중국으로부터 대만으로 도입된 중국줄무늬목거북 대만 집단보다는 높지만, 다른 야생 거북류 집단들에 비해 낮다고 할 수 있다. 이는 20세기 동안 식용, 약용 등 여러 가지 목적을 위한 야생 남생이 남획, 대규모 도로 건설과 현대화된 주택단지 조성 등 국토 개발에 따른 서식지의 파괴, 산업화에 따른 환경(수질) 오염과 농약 사용량 증가에 따른 피해 등 다양한 이유에 의해 남생이 개체군이 급감한 데에서 초래된 결과라 하겠다(Lovich et al., 2011; van Dijk, 2011; Kim et al., 2013). 이러한 결과는 모계 유전 마커인 미토콘드리아 DNA 서열의 유전적 다양성이 낮은 수준임을 기술한 선행 연구결과(Moon et al., 2025)와도 일치한다.
중국에서 포획되어 증식중인 5개의 남생이 집단들(Guangzhou, Nanning, Haikou, Wuhu 1, Wuhu 2; Table 2)에서 FIS값이 0.119-0.265 사이로 양의 값을 나타내고, 집단 내에서 Hobs가 부족한 형태를 하고 있다는 점에서 다양한 정도에서 근친교배가 발생하고 있다고 판단하였다(Bu et al., 2019). 또 다른 포획-증식 집단인 Hubei 남생이 집단 역시 평균 FIS가 0.170으로 근친교배가 진행되고 있다고 추정하였고(Xu et al., 2023), 대만 Kinmen Island의 야생에서 포획된 남생이에서도 Hobs가 Hexp보다 매우 낮아, 근친교배에 의한 유전적 다양성이 감소하고 있다고 하였다(Lee et al., 2019). 대부분의 중국 남생이 집단에서 유전적 다양성의 감소가 포획과정보다는 증식과정에서 발생한다고 제안하였다(Zhang et al., 2010). 집단 내에서 근친교배의 발생은 결과적으로 이형접합체의 소실에 따른 유전적 다양성의 감소뿐만 아니라 집단의 환경 적응력의 감소를 초래할 수 있다고 하였다(Edwards et al., 2014). 이러한 연구결과들은 현재 우리나라 남생이의 두 집단에서도 근친교배가 활발하게 발생하고 있다는 점에서 향후 유전적 다양성의 증진을 위한 적절한 보호 프로그램이 마련되어야 함을 시사하고 있다.
Table 2. Comparison of genetic diversity and inbreeding coefficient values obtained from two South Korean populations with those reported in Chinese and Taiwanese populations, and other species
| Species | Country | Population1 | La | Na | k | Genetic diversity | Reference | |||
|---|---|---|---|---|---|---|---|---|---|---|
| Hobs | Hexp | PIC | FIS | |||||||
| M. reevesii | South Korea | G-GY | 9 | 113 | 9.3 | 0.554 | 0.629 | 0.592 | 0.105 | This study |
| China | H-A | 12 | 300 | 8.6 | 0.479 | 0.581 | 0.541 | 0.170 | Xu et al. 2023 | |
| China | ZJ-S | 8 | 29 | 10.9 | 0.708 | 0.815 | n.d. | 0.285 | Ye et al. 2009 | |
| China | Wuhu1 | 12 | 20 | 4.8 | 0.521 | 0.589 | 0.493 | 0.119 | Bu et al. 2019 | |
| China | Wuhu2 | 12 | 22 | 3.6 | 0.436 | 0.538 | 0.441 | 0.194 | Bu et al. 2019 | |
| China | Guangzhou | 12 | 22 | 7.9 | 0.568 | 0.717 | 0.664 | 0.211 | Bu et al. 2019 | |
| China | Naning | 12 | 8 | 4.2 | 0.479 | 0.640 | 0.537 | 0.265 | Bu et al. 2019 | |
| China | Haikou | 12 | 15 | 3.4 | 0.461 | 0.557 | 0.453 | 0.177 | Bu et al. 2019 | |
| Taiwan | Kinmen | 13 | 9 | 5.2 | 0.572 | 0.602 | 0.532 | n.d. | Lee et al., 2019 | |
| M. sinensis | Taiwan | Kinmen | 13 | 11 | 3.9 | 0.392 | 0.504 | 0.429 | n.d. | Lee et al., 2019 |
| M. mutica | China | Jinhua | 14 | 30 | 10.4 | 0.646 | 0.823 | 0.785 | 0.114 | Zhang et al. 2010 |
| Caretta caretta | Spain | Cape Verde | 12 | 50 | 7.3 | 0.613 | 0.640 | 0.595 | n.d. | Monzon-Arguello et al. 2008 |
남생이 두 집단(금곡, 강진-영암)의 MS 대립유전자형에 근거한 FCA 분석 결과 제 1분산치는 24.6%, 제 2분산치 12.3%, 제 3분산치 10.7%로, 총 47.3%로 확인되었다(Fig. 1A). 금곡 집단과 강진-영암 집단은 뚜렷한 2개의 계통으로 군집화되지 않았고, 대부분 중첩된 양상을 보였다. 단, 금곡 집단의 일부가 중심에서 벗어나 독립적으로 위치하였으며, 이들은 두 집단에서 관찰된 보편적인 대립유전자형의 범주를 벗어나는 독특한 대립유전자형을 보유한 개체들로 판단되지만, 이들이 보유한 독특한 대립유전자형이 남생이에서 기원한 것으로 단정할 수는 없다. 이뿐만 아니라 Baek et al. (2024)은 한반도에서 남생이와 중국줄무늬목거북의 교잡 후손들이 서식하고 있음을 보고하였고, 중국, 일본, 대만 등에서도 Mauremys 종간의 교잡은 빈번하게 보고되고 있다. 따라서 이번 FCA 분석에서 독자적인 위치를 차지한 개체들의 경우, 중국줄무늬목거북을 포함한 다른 종과의 교잡 연구가 필요하다고 하겠다.
Bayesian 접근법을 활용한 대립유전자의 단상체형 분포를 기준으로 집단의 유전적 구조를 분석한 결과, 유전적 계보 수(K)는 K=2 model에서 ΔK=124.6으로 최적의 상태를 나타내었고(Fig. 1B), K=1부터 K=10까지 ΔK의 범위는 0.639-124.6이었다. 두 가지 유전적 계보들은 금곡 집단과 강진-영암 집단에서 모두 출현하였으나, 두 집단에서 출현하는 양상에는 다소 큰 차이를 보였다. 금곡 집단의 경우 푸른색으로 표현된 계보가 더 많은 분포를 나타내지만, 강진-영암 집단은 거의 주황색에 해당하는 계보가 대부분의 영역을 차지하고 있다. 대립유전자형의 분포를 기준으로 개체별 유연관계를 분석한 결과, NJ tree 상에서 몇 개의 cluster를 형성하였고, 금곡 집단과 강진-영암 집단 내에서는 여러 개체가 하나의 분지를 형성하는 형태를 보였으나, 금곡 집단과 강진-영암 집단 개체들이 하나의 분지 내에서 동시에 관찰되지는 않았다(Fig. 1C). 이 결과는 미토콘드리아 DNA 서열의 다형성이 집단 내에서 각각 두 가지 단상체형으로 구분되는 연구결과(Moon et al., 2025)와 일치하지만, 핵 DNA의 MS 대립유전자형의 집단별 구성은 서로 다른 진화적 역사를 보유한 것으로 판단된다.
한반도 육상 포유류 중 너구리와 산양의 경우는 K≥2인 구조를 나타내었고, 유전적으로 지역적인 분지 양상을 나타내기도 하였으나(Hong et al., 2013; Choi et al., 2015), 고라니와 삵은 K=1 모델에서 최적인 유전적 집단 구조를 보이면서 지역별 유전적인 차이가 없는 단일 집단이라고 제안되었다(Lee et al., 2011; Ko et al., 2018). 멧토끼의 경우 K=2 model에서 최적인 구조를 나타내었으나, 강원도와 경상북도 집단에서 집단-특이적인 대립유전자 단상체형이 없어, 두 집단 사이에서 지리적 장벽이 없다고 추정되었다(Kim et al., 2012). 반면, 수생태계에서 서식하지만 땅 위로 이동하기도 하는 수달은 유라시아 대륙에서 유전적 다양성은 낮지만, K=3인 유전적 구조를 형성하였고(Li et al., 2023), 국내에서 경상남도 수달 집단이 크게 진주, 거제도 지역으로 구분되는데, 지역 간 차이는 수계에 따라 유전적으로 고정되어 있다고 보고되었다(Park et al., 2011). 담수어류인 배가사리(Microphysogobio longidorsalis)는 한강 수계에서 남한강, 북한강, 임진강에 서식하며, 여울마자(Microphysogobio rapidus)는 낙동강 수계에 서식하며, 두 종 모두 K=2의 유전적 구조를 나타내고, 하류의 합수부나 호수 등을 통해 연결되어 있어, 유전적 연결성이 유지되어 지역 집단 사이의 유의적인 유전적 차이가 없다고 보고되었다(Hong et al., 2023; Kim et al., 2024). 반면, 감돌고기(Pseudopungtungia nigra)는 수계가 지리적으로 분리된 금강과 만경강 사이에서 유전적으로 분화된 양상을 보였다(Kim et al., 2023). 이상의 연구결과들을 종합해 볼 때, 남생이 금곡 집단과 강진-영암 집단은 K=2의 유사한 유전적 구조를 보이고, FCA 분석에서 대립유전자형들 대부분이 중복되어 있으나, 각각의 집단 형성에 이바지한 유전적 계보에는 다르다고 판단된다. 또한 이들 두 집단은 같은 영산강 수계에 속하기는 하지만, 지리적으로 금곡 집단의 서식지는 양산강의 중상류의 지류인 중암천의 상부인 무등산 인근에 있고, 강진-영암 집단은 영산강의 하류에 있는 학산천, 영암천 두 지류의 상부인 월출산 인근이라는 점을 고려했을 때, 두 지역의 지리적 연결성은 거의 없다. 남생이의 계절별 최대 이동거리 196m, 행동권 크기 0.256-6.138 ha로 알려져 있고(Song et al., 2014; Jo et al., 2017), 두 지역의 직선거리가 50km 이상 떨어져 있다는 점에서 단일 번식기 내에서 두 지역이 유전적으로 연결된 가능성은 없다. 다시 말해서, 현재의 이들 두 집단은 인위적인 중재가 없는 상태에서는 유전적으로 완전히 독립적인 상태라 판단된다.
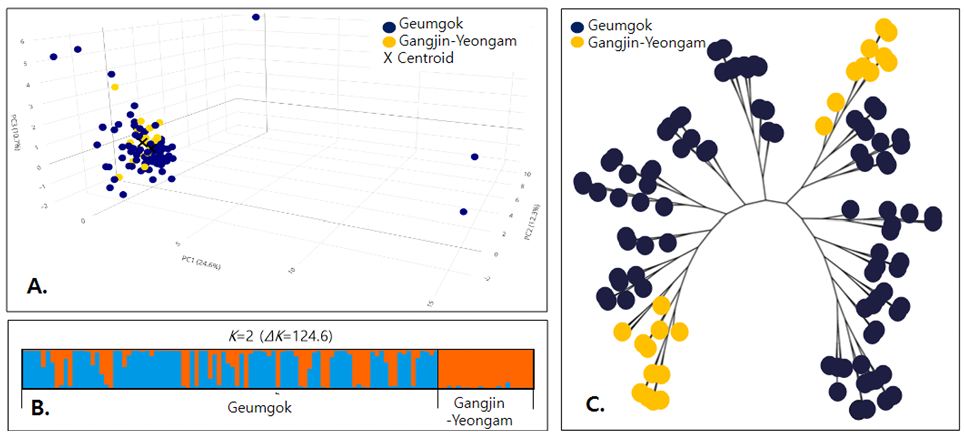
Figure 1. Results of factorial correspondence analysis (A), genetic structure using Bayesian model-based clustering (B) and unrooted neighbor-joining (NJ) tree for microsatellite genotype-based haplotypes of the Mauremys reevesii in two Reevesi’ turtle populations in South Korea (C).
이번 연구에서 우리나라의 서남부권에서 수집된 남생이 2개 집단(금곡, 강진-영암)은 20세기 동안 급격한 개체수 감소를 거치는 과정을 통해 유전적 다양성이 감소하였으나, 이후 외래 유전자 공급원의 유입이 발생하였고, 이후 번식과정을 통해 개체군 크기가 증가하고 있으나, 그 과정에서 근친교배가 발생하고 있었다. 두 집단 모두 두 개의 유전적 계보로 형성되었으나, 현재의 집단 형성에 이바지한 계보는 두 집단이 각각 다른 양상을 보였다. 근친교배의 비율과 번식을 주도하고 있는 유전적 계보들을 추적하기 위해서는 성별과 나이 정보를 수집한 후 유전자 수준의 친자확인을 통해 가계도를 작성하고, 이를 바탕으로 혈통 구조를 분석해야 할 것이다. 특히, 우리나라에 도입된 중국 기원 남생이들과 기존의 한반도 기원 남생이들과의 교잡 여부에 대한 판독은 멸종위기종, 천연기념물로 지정된 고유 개체군의 보호에 필수적인 선결과제이다. 또한 FCA 분석에서 독립적인 위치를 나타낸 개체들은 다른 종과의 교잡 시험이 필요하며, 국내 다른 지역에서 서식하는 남생이 집단들과의 유전적 분화와 연결성도 시험하여야 한다. 이번 연구에서 시험한 금곡, 강진-영암 집단의 경우 지리적으로 떨어져 있어 자체적인 연결이 불가능하다는 점에서 두 집단의 유전적 다양성 증진을 위해서는 각각의 집단들의 인근 지역에서 지속적으로 새로운 서식지와 서식 개체들을 발굴하고, 서식지 간 연결성의 확보, 향후 국가 차원의 개체군 관리에 필요한 메타 개체군의 마련 등 종합적인 검토가 요구된다.
No potential conflict of interest relevant to this article is reported.
이 연구는 환경부의 지원을 받아 국립공원공단 국립공원야생생물보전원의 “멸종위기종증식복원및반달가슴곰공존문화조성사업”에서 수행된 결과의 일부입니다. 연구비 지원에 감사드립니다.
Baek HJ, Cheong E, Kim Y, Koo KS, Kim SH, Park CD and Yoon JD. 2024. Geographical distribution of Mauremys sinensis, Mauremys reevesii, and their hybrids in South Korea. Animals 14:2626.
[DOI][PubMed][PMC]
Bao WB, Shu JT, Xu SH and Li HF. 2007. Effects of sample size and sec ratio on various genetic diversity measures with microstallite markers. Chinese Journal of Animal Science 43(1):6-9.
Belkhir K, Borsa P, Chikhi L, Raufaste N and Bohomme F. 2004. GENETIX 4.05, logiciel sous Windows TM pour la genetique des populations. Université de Montpellier II, Montpellier. France.
Botstein K, Borsa P, Chikhi L, Raufaste N and Bonhomme F. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphism. American Journal of Human Genetics 32(3):314-331.
Bu X, Wang X, Lu W, Cai W, Xia X and Nie L. 2019. Genetic diversity of the captive Chinese pond turtle (Mauremys reevesii) populations in China assessed by microsatellite markers. The Journal of Animal & Plant Sciences 29(4):1160-1168.
CHA (Cultural Heritage Administration). 2006. Yearbook of Cultural Heritage 2006. Cultural Heritage Administration. Seoul, Republic of Korea. pp.51-59.
Chang MH, Song JY and Koo KS. 2012. The status of distribution for native freshwater turtles in Korea, with remarks on taxonomic position. Korean Journal of Environmental Biology 30(2):151-155.
Chen TH and Lue KY. 2010. Population status and distribution of freshwater turtles in Taiwan. Oryx 44(2):261-266.
[DOI]
Choi SK, Chun S, An J, Lee M, Kim HJ, Min M, Kwon S, Choi TY, Lee H and Kim KS. 2015. Genetic diversity and population structure of the long-tailed goral, Naemorhedus caudatus, in South Korea. Genes & Genetic Systems 90:31-41.
[DOI][PubMed]
CITES (Convention on International Trade in Endangered Species of Wild Fauna and Flora). 2015. Appendices I, II and III. Available from https://cites.org/eng/app/appendices.php
Dierginger D and Schlotterer C. 2003. Microsatellite analyser (MSA): a platform independent analysis tool for large microsatellite data sets. Molecular Ecology Notes 3(1):167-169.
[DOI]
Earl DA and Von-Holdt BM. 2012. Structure Harvester: A website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources 4:359-361.
[DOI]
Edwards T, Cox EC, Buzzard V, Weise C, Hillard LS and Murphy RW. 2014. Genetic assessments and parentage analysis of captive Bolson tortoises (Gopherus flavomarginatus) inform their “rewilding” in New Mexico. PLoS One. 9:e102787.
[DOI][PubMed][PMC]
Evanno G, Regnaut S and Goudet J. 2005. Detecting the number of clusters of individuals using the software structure: a simulation study. Molecular Ecology 14:2611-2620.
[DOI][PubMed]
Felsenstein J. 2004. PHYLIP (phylogeny inference package). Version 3.6. Distributed by the author, Department of Genome Sciences, University of Washington, Seattle. Available from https://phylipweb.github.io/phylip/
Fong JJ and Chen TH. 2010. DNA evidence for the hybridization of wild turtles in Taiwan: possible genetic pollution from trade animals. Conservation Genetics 11:2061-2066.
[DOI]
Frankham R. 1996. Relationship of genetic variation to population size in wildlife. Conservation Biology 10:1500-1508.
[DOI]
Goudet J. 2001. FSTAT, a program to estimate and test gene diversities and fixation indices (ver. 2.9.3). Accessed in https://www2.unil.ch/popgen/softwares/fstat.htm on 01 April 2025.
Hedrick PW. 2005. A standardized genetic differentiation measure. Evolution 59(8):1633-1638.
[DOI][PubMed]
Hong Y, Kim K, Lee H and Min M. 2013. Population genetic study of the raccoon dog (Nyctereutes procyonnoides) in South Korea using newly developed 12 microsatellite markers. Genes & Genetic Systems 88:69-76.
[DOI][PubMed]
Hong YK, Kim KR, Kim KS and Bang IC. 2023. The impact of weir construction in Korea’s Nakdong river on the population genetic variability of the endangered fish species, rapid small gudgeon (Microphysogobio rapidus). Genes 14:1611.
[DOI][PubMed][PMC]
Hudson DH and Bryant D. 2006. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution 23(2):254-267.
[DOI][PubMed]
Jo SI, Na S, An CK, Kim H, Jeong YJ, Lim YM, Kim SD, Song JY and Yi H. 2017. Comparison analysis for using the habitat pattern between the Korean endangered species, Mauremys reevesii, and the exotic species, Trachemys scripta elegans. Korean Journal of Environment and Ecology 31:397-408.
[DOI]
Jung YJ, Kim IH and Park D. 2016. The possibility assessment of age estimation of the endangered Reeves’ turtle (Mauremys reevesii) based on the number if growth lines on dorsal scutes. Korean Journal of Environment and Ecology 30(6):970-976.
[DOI]
Kalinowski ST, Taper M and Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology 16:1099-1106.
[DOI][PubMed]
Kaeuffer R, Coltman DW, Chapuis JL, Pontier D and Reale D. 2007. Unexpected heterozygosity in an island mouflon population founded by a single pair of individuals. Proceedings of the Royal Society B 274:527-533.
[DOI][PubMed][PMC]
Kekkonen J, Wikstro M and Brommer JE. 2012. Heterozygosity in an isolated population of a large mammal founded by four individuals is predicted by an individual-based genetic model. PLoS One 7(9):e43482.
[DOI][PubMed][PMC]
Kim SI, An J, Choi SK, Lee YS, Park HC, Kimura J, Kim KS, Min MS and Lee H. 2012. Development and characterization of nine microsatellite loci from the Korean hare (Lepus coreanus) and genetic diversity in South Korea. Animal Cells and Systems 16(3):230-236.
[DOI]
Kim KR, Kwak YH, Sung MS, Cho SJ and Bang IC. 2023. Population structure and genetic diversity of the endangered fish black shinner Pseudopungtungia nigra (Cyprinidae) in Korea: a wild and restoration population. Scientific Reports 13:9692.
[DOI][PubMed][PMC]
Kim KR, Sung MS, Hwang Y, Jeong JH and Yu JN. 2024. Assessment of the genetic diversity and structure of the Korean endemic freshwater fish Microphysogobio longidorsalis (Gobioninae) using microsatellite markers: a first glance from population genetics. Genes 15:19.
[DOI][PubMed][PMC]
Kim SR, Lee JH, Song JY, Chang MH, Sung HC and Cho DG. 2013. A study on the habitat restoration model for Chinemys reevesii. Journal of the Korean Society of Environmental Restoration Technology 16(2):115-125.
[DOI]
Kim YL, Kim HR, Kim DH, Jamg JE, Son JI, Han SH and Lee HJ. 2025. Phylogenetic status and population genetic structure of the long-tailed goral, Naemorhedus caudatus inhabiting the Seoraksan National Park in Korea. Korean Journal of Environmental Biology 43(1):28-41.
[DOI]
Ko BJ, An J, Seomun H, Lee MY and Eo SH. 2018. Microsatellite DNA analysis reveals lower than expexted genetic diversity in the threatened leopard cat (Prionailurus bengalensis) in South Korea. Genes & Genomics 40:521-530.
[DOI][PubMed]
Koo KS, Jang MH, Kim DI, Kim SH, Baek HJ and Sung HC. 2019. Report on the large population and habitat status of endangered species, Mauremys reevesii Gray 1831 (Reptilia; Testudines; Geomydidae) in South Korea. Journal of the Korean Society of Environmental Restoration Technology 33(4):402-407.
[DOI]
Koo KS, Song JY and Chang MH. 2015. Mating behavior of Reeves’ turtle, Mauremys reevesii. Journal of National Park Research 6(3):111-114.
Laurie CC, Nickerson DA, Anderson AD, Weir BS, Livingston RJ, Dean MD, Smith KL, Schadt EE and Nachman MW. 2007. Linkage disequilibrium in wild mice. PLoS Genetics 3(8):e144.
[DOI][PubMed][PMC]
Lee Y, Lin JW, Tseng SP, Chen TS and Lin SM. 2019. Human disturbance as a possible cause of genetic introgression from exotic into native Mauremys turtles. Animal Conservation 22(6):556-567.
[DOI]
Lee YS, Choi SK, An J, Park HC, Kim SI, Min MS, Kim KS and Lee H. 2011. Isolation and characterization of 12 microsatellite loci from Korean water deer (Hydropotes inermis argyropus) for population structure analysis in South Korea. Genes & Genomics 33:535-540.
[DOI]
Li SH, Yeh C, Jang-Liaw NH, CHang SW, Lin YH, Tsai CE, Chiu CC, Chen CW, Ke HR, Wang Q, Lu Y, Zheng K, Fan P, Zhang L and Liu Y. 2023. Low but highly geographically structured genomic diversity of East Asian Eurasian otters and its conservation implications. Evolutionary Applications 17:e13630.
[DOI][PubMed][PMC]
Li YL and Liu JX. 2018. StructureSelector: A web based software to select and visualize the optimal number of clusters using multiple methods. Molecular Ecology Resources 18:176-177.
[DOI][PubMed]
Lidicker WZJr and McCollum FC. 1997. Allozymic variation in California Sea otters. Journal of Mammalogy 78(2):417-425.
[DOI]
Lim HT, Seo BY, Jung EJ, Yoo CK, Zhong T, Cho IC, Yoon D, Lee JG and Jeon JT. 2009. Establishment of a microsatellite marker set for individual, pork brand and product origin identification in pigs. Journal of Animal Science & Technology 51(3):201-206.
[DOI]
Lin SM, Lee Y, Chen TH and Lin JW. 2015. Habitat preference and management of a Chinese pond turtle population protected by the demilitarized Kinmen Islands. Journal of Herpetology 49:399-404.
[DOI]
Liu L, Nie L, Bu X, Xia X, Huang Z, Jing W, Jiang Y and Wang L. 2012. Isolation and characterization of ten novel polymorphic microsatellites loci in the Chinese pond turtle (Mauremys reevesii) and cross-species amplification in other Cryptodira species. Anhui Normal University Master Thesis, Anhui, China.
Lovich JE, Yasukawa Y and Ota H. 2011. Mauremys reevesii (Gray 1831) – Reeves’ Turtle, Chinese Three-keeled Pond Turtle. In: Rhodin AGJ, Pritchard PCH, van Dijk PP, Saumure RA, Buhlmann KA, Iversion JB, Mittermeier RA. (Eds.). Conservation Biology of Freshwater Turtles and Tortoises: A Compilation Project of the IUCN/SSC Tortoise and Freshwater Turtle Specialist Group. Chelonian Research Monographs No. 5, pp.050.1-050.10.
[DOI]
MOE (Ministry of Environment). 2017. Wildlife Conservation and Management Act.
Moon KS, Lee YG, Go CH, Kim TW, Lee SH, Yang DH, Han SH and Sung HC. 2025. Genetic diversity based on the mitochondrial Cytochrome B gene polymorphisms in the Reeves’ turtle populations in southwestern region of South Korea. Journal of Animal Breeding and Genomics 9(1):39-47.
[DOI]
Monzon-Arguello C, Munoz J, Marco A, Lopez-Jurado LF and Rico C. 2008. Twelve new polymorphic microsatellite markers from the loggerhead sea turtle (Caretta caretta) and cross-species amplification on other marine turtle species. Conservation Genetics 9:1045-1049.
[DOI]
Moravcikova N, Kasarda R, Zidek R, McEwan JC, Brauning R, Landete-Castillejos T, Chonco L, Ciberej J and Pokoradi. 2023. Traces of humanmediated selection in the gene pool of red deer populations. Animals 13(15):2525.
[DOI][PubMed][PMC]
Myung CH, Kim TW, Lee SH, Jeong WJ, Yang JJ, Kang HN, Kang HC, Kim JY, Lim HT and Han SH. 2025. Novel microsatellite markers for the identification of the Asiatic black bears being restored in South Korea. Journal of Agriculture & Life Science 59(1):83-89.
[DOI]
NIBR (National Institute of Biological Resources). 2011. Red data book of endangered amphibians and reptiles in Korea. p.125.
Oh HS, Park SM and Han SH. 2017. Mitochondrial haplotype distribution and phylogenetic relationship of an endangered species Reeve’s turtle (Mauremys reevesii) in East Asia. Journal of Asia-Pacific Biodiversity 10:27-31.
[DOI]
Park MS, Lim HT, Oh KC, Moon YR, Kim JG and Jeon JT. 2011. A phylogenetic analysis of otters (Lutra lutra) inhabiting in the Gyeongnam area using D-loop sequence of mtDNA and microsatellite markers. Journal of Life Science 21(3):385-392.
[DOI]
Parreira BR and Chikhi L. 2015. On some genetic consequence of social structure, mating systems, dispersal, and sampling. Proceedings of the National Academy of Sciences USA 112(26):E3318-E3326.
[DOI][PubMed][PMC]
Payseur BA, Jing P and Haasl RJ. 2011. A genomic portrait of human microsatellite variation. Molecular Biology and Evolution 28(1):303-312.
[DOI][PubMed][PMC]
Pemberton JM, Ellis PE, Pilkington JG and Berenos C. 2017. Inbreeding depression by environment interactions in a free-living mammal population. Heredity 118:64-77.
[DOI][PubMed][PMC]
Pruett CL and Winker K. 2008. The effects of sample size on population genetic diversity estimates in song sparrows Melospiza melodia. Journal of Avian Biology 39:252-256.
[DOI]
Raymond M and Rousset F. 1995. GENEPOP (Version 1.2): Population genetics software for exact tests and ecumenicism. Journal of Heredity 86(3):248-249.
[DOI]
Rosenberg NA, Pritchard JK, Weber JL, Cann HM, Kidd KK, Zhivotovsky LA and Feldman MW. 2002. Genetic structure of human populations. Science 298:2381-2385.
[DOI][PubMed]
Saitou N and Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution 4:406-425.
Song JY. 2007. Current status and distribution of reptiles in the Republic of Korea. The Korean Journal of Environmental Biology 25(2):124-138.
Song JY, Chang MH and Koo KS. 2014. Distribution and movement of Reeve’s turtle (Mauremys reevesii) in South Korea. Chinese Journal of Wildlife 35(3):239-334.
Suzuki D, Ota H, Oh HS and Hikida T. 2011. Origin of Japanese populations of Reeves’ pond turtle, Myauremys reevesii (Reptilia: Geoemydidae), as inferred by a molecular approach. Chelonian Conservation Biology 10(2):237-249.
[DOI]
van Dijk PP. 2011. Mauremys reevesii. The IUCN Red List of Threatened Species 2011:e.T170502A97431862.
Weber JL and Wong C. 1993. Mutation of human short tandem repeats. Human Molecular Genetics 2:1123-1128.
[DOI][PubMed]
Weir BS and Hill WG. 2002. Estimating F-statistics. Annual Reviews of Genetics 36:721-750.
[DOI][PubMed]
Wright S. 1951. The genetical structure of populations. Annals of Eugenics 15:323-354.
[DOI][PubMed]
Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution 19:395-420.
[DOI]
Xia X, Wang L, Nie L, Huang Z, Jiang Y, Jing W and Liu L. 2011. Interspecific hybridization between Mauremys reevesii and Mauremys sinensis: evidence from morphology and DNA sequence data. African Journal of Biotechnology 10(35):6716-6724.
Xu H, Yang X, Ni W, Liu F, Chen H, Zhu X and Liu X. 2023. Genetic diversity and genetic structure of selected generations of Chinese pond turtle (Mauremys reevesii) based on microsatellite DNA. Journal of Dalian Ocean University 38(5):812-818.
Ye R, Zheng R, Wang L and Du W. 2009. Polymorphic microsatellite loci in the Chinese pond turtle (Chinemys reevesii). Conservation Genetics 10:1045-1048.
[DOI]
Zhang Y, Zhang JY and Zheng RQ. 2010. Isolation and characterization of 14 polymorphic microsatellite loci in the Asian yellow pond turtle, Mauremys mutica(Cantor, 1842). Aquaculture Research 41:e353-e356.
[DOI]
Zhao J, Zhu C, Xu Z, Jiang X., Yang S and Chen A. 2017. Microsatellite markers for animal identification and meat traceability of six beef cattle breeds in the Chinese market. Food Control 78:469-475.
[DOI]