Kwang-Seon Moon1,2, Young-Gu Lee3, Chang-Hyun Go4, Tae-Wook Kim5, Sa-Hyun Lee5, Doo-Ha Yang5, Sang-Hyun Han5*, Ha-Cheol Sung6*
1Mudeungsan National Park Nature Center, Korea National Park Service, Gwangju 61146, Republic of Korea
2Department of Biological Science and Biotechnology Graduate School, Chonnam National University, Gwangju 61186, Republic of Korea
3Western Regional Headquarters, Korea National Park Service, Jeonju 54843, Republic of Korea
4Mudeungsan National Park Office, Korea National Park Service, Gwangju 61494, Republic of Korea
5National Park Institute for Wildlife Conservation, Korea National Park Service, Yeongju 36015, Republic of Korea
6Department of Biological Science, Chonnam National University, Gwangju 61186, Republic of Korea
*Correspondence to Sang-Hyun Han, Ha-Cheol Sung E-mail: hansh04@knps.or.kr, shcol2002@jnu.ac.kr
Volume 9, Number 1, Pages 39-47, March 2025.
Journal of Animal Breeding and Genomics 2025, 9(1), 39-47. https://doi.org/10.12972/jabng.2025.9.1.4
Received on March 04, 2025, Revised on March 27, 2025, Accepted on March 28, 2025, Published on March 31, 2025.
Copyright © 2025 Korean Society of Animal Breeding and Genetics.
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0).
endangered, genetic diversity, haplotype, mitochondrial DNA, Reeve’s turtle
남생이(Mauremys reevesii) (Reeves’ turtle, Chinese three-keeled pond turtle)는 중형 담수거북으로, 중국의 중부와 동부, 대만의 Kimmen Island, 한반도에 걸쳐 동아시아에 분포하며, 대만 본섬과 일본, 인도네시아, 팔라우, 티모르, 미국 등은 인위적으로 도입된 것으로 추정되고 있다(Lovich et al., 1985; Chen and Lue, 2010; van Dijk, 2011). 우리나라에서는 주로 오염되지 않은 일부 하천, 강이나 호수 등 유속이 느린 담수에 서식하며, 제주도를 비롯한 도서지방을 제외한 한반도 전역에 분포한다(Song, 2007; NIBR, 2011). 1970년대 이후 종교활동, 애완용, 식용, 약재용으로 남생이와 붉은귀거북(Trachemys scripta elegans) 등 다양한 거북류가 해외에서 도입되었고, 상당수가 자연생태계로 유출되어 생태적 습성이 비슷한 우리나라 남생이의 생존을 위협하고 있다(Oh and Hong, 2007; Koo et al., 2015; Jo et al., 2017). 남생이는 천연기념물(CHA, 2006), 멸종위기야생생물 Ⅱ급(NIBR, 2011; MOE, 2017)으로 지정되었고, 국제자연보전연맹(International Union for the Conservation of Nature, IUCN)의 적색목록, Convention on International Trade in Endangered Species of Wild Fauna and Flora (CITES)의 부속서 Ⅲ에 포함되어 국가 간 거래가 규제되고 있다(van Dijk, 2011; IUCN, 2015; CITES, 2015).
20세기 말부터 분자유전학적 실험기법을 이용한 다양한 연구들이 시도되었고, 동물의 계통발생과 진화, 유전적 다양성 연구에도 DNA를 이용한 연구성과들이 보고되고 있다(Harris et al., 1998; Iverson et al., 2007; Xu et al., 2023). 특히, 미토콘드리아는 단일 세포 내에 수백에서 수 만 개까지 존재하여 적은 시료에서도 다수의 DNA 분자들을 확보할 수 있고, 미토콘드리아 DNA (mitochondrial DNA, mtDNA)가 모계유전을 하고 핵 DNA 보다 돌연변이율이 높아 진화 속도가 빠르기 때문에(Brown et al., 1979; Giles et al., 1980; Clayton, 1982), 종이나 집단 수준에서 유전적 다양성 연구에 활발히 이용되고 있다(Carter, 2007; Nabholz et al., 2009; Caragiulo et al., 2014; Gonzalez-Oliver et al., 2018; Laxmivandana et al., 2022).
국내에서 남생이에 대한 연구는 서식지 분포와 모델링, 연령 측정, 성장, 번식과 관련된 연구결과들이 주로 보고되었다(Kim et al., 2013; Jung et al., 2016; Koo et al., 2015, 2019; Koo and Song, 2018). 반면, 동북아시아에서는 남생이를 포함한 여러 육지 거북류들이 고유 개체인지, 이입 개체인지를 판독하거나, 종 내 또는 종 간 교잡을 추적하는 등 종 식별과 계통학적 기원에 대한 연구결과들이 주로 보고되었다(Fong and Chen, 2010; Xia et al., 2011; Suzuki et al., 2011; Oh et al., 2017). 국내에서는 mtDNA 분석을 통해 한반도 고유 개체뿐만 아니라 중국 기원인 개체들도 서식하고 있으며(Oh et al., 2017), 중국줄무늬목거북(M. sinensis)과 남생이×중국줄무늬목거북의 교잡종도 서식하고 있다고 보고되었다(Baek et al., 2024). 하지만, 아직까지 우리나라 여러 지역에 서식하는 남생이 집단의 유전적 특성을 설명할 수 있는 연구결과는 없다.
이 연구에서는 우리나라 서남부권에 위치한 전라남도 구례군, 강진군과 영암군, 광주광역시 북구에서 수집한 남생이 3개 집단에서 mtDNA cytB 염기서열의 다형성에 근거하여 유전적 다양성을 살펴보고, 유전적 다양성 증진을 위한 기초자료를 제공하고자 하였다.
연구에 이용한 남생이 시료는 2019년부터 2024년까지 전라남도 구례군에서 수집한 구례 집단(n=14), 영암군과 강진군에서 수집한 강진-영암 집단(n=22), 광주광역시 북구 금곡동에서 수집한 금곡 집단(n=92)을 이용하였다(Table 1). 수집된 남생이에서 등갑의 표피를 긁거나 절개하여 DNA 추출에 이용하였다. DNA 추출은 DNeasy Blood & Tissue Kit (Qiagen, Hilden Germany)를 이용하여 공급자의 안내에 따랐고, 추출한 DNA는 중합효소 연쇄반응(polymerase chain reaction, PCR)의 주형으로 이용하였다.
추출한 DNA를 주형으로 mtDNA cytB 유전자를 PCR로 증폭하였다. PCR에 사용된 primer는 Oh et al. (2017)에 의해 고안된 프라이머 쌍을 이용하였다. PCR은 Multiplex Plus PCR Kit (Qiagen, Germany)와 혼합하여 20 ul로 반응하였고, Mastercycler Nexus Gradient (Eppendorf, Germany)를 이용하여 증폭하였다. PCR 반응은 94℃에서 5분간 초기 변성 후, 94℃ 30초, 58℃ 45초, 72℃ 45초로 이어지는 연쇄반응을 35회 수행한 후, 72℃에서 5분간 최종 신장하였다. 증폭이 끝난 PCR 산물은 1.5% agarose 겔 상에서 전기영동하여 확인한 후, DNA 염기서열 분석에 이용하였다. PCR 산물은 정제 후 dye-termination method를 이용하여 반응하고, ABI Automated DNA Sequencer 3730XL (Applied Biosystems, CA)을 이용하여 DNA 서열을 결정하였다. 결정된 염기서열은 NCBI의 nucleotide database에서 basic local alignment search tool (BLAST, https://blast.ncbi.nlm.nih.gov/Blast.cgi) 검색을 통해 기존에 서열 중 최고 유사서열을 탐색하여 남생이 종을 확인하고, 선행연구에서 보고된 서열의 정보를 수집하였다.
이 연구를 통해 결정된 mtDNA cytB 서열들과 선행연구에서 보고된 우리나라(SK 집단, k=44), 중국(k=39), 대만(k=10), 일본(k=133), 미국(k=2) 집단의 서열들(Table 2)을 CLUSTAL W (Thompson et al., 1994)로 다중 정렬하였다. 정렬된 서열들에 대한 유전적 변이 확인과 단상체형 결정, 변이유전적 다양성, 중립성 시험, 유전적 분화 시험은 DnaSP6 프로그램(Rozas et al., 2017)을 이용하였다. 염기서열 다양도(π)와 단상체형 다양도(Hd)는 Nei (1987)의 방법으로 산출하였고, 중립성 시험을 위해 Tajima’s D (Tajima, 1989)와 Fu’s Fs (Fu, 1997) 지수를 산출하였다. 집단 사이의 유전적 분화는 Nei (1987)의 Dxy 지수와 Hudson et al. (1992)의 Fst 지수를 산출하여 추정하였다.
검출된 단상체형 서열들의 계통 유연관계를 살펴보기 위해 MEGA7 (Kumar et al., 2016) 프로그램을 이용하여 계통수를 작성하였다. 최적의 유전적 거리지수는 Bayesian Information Criterion 점수가 가장 낮은 모델을 염기치환 양상을 가장 잘 설명할 수 있는 모델로 간주하여 Kimura’s two parameter method (Kimura, 1980)의 방식으로 산출하였고, Gamma 이산 분포를 사용하여 서열 간 진화 속도의 불균일성을 완화한 neighbor-joining (NJ) tree (Saitou and Nei, 1987)를 작성하였다. 계통수 분지의 신뢰성을 제공하기 위한 Bootstrap (Felsenstein, 1985)은 1,000회 반복 수행하였고, 분지 형태의 최적화를 위해 Mauremys 속의 거북류(M. caspica, AY434594; M. mutica, AY434622; M. annamensis, AY434564)를 포함하였고, 공통조상 추적을 위한 외군은 Cuora amboinensis (FJ763736)를 이용하였다.
우리나라 서남부권에 수집한 남생이 3개 집단에서 128개의 cytB 서열과 선행연구에서 보고된 우리나라 남생이 서열(SK 집단 44개 서열)을 종합하여 172개 서열에서 유전적 다양성을 분석하였다(Table 1). 전체에서 확인된 염기변이의 수(S)=13개, π=0.00233 ±0.00019, 단상체형의 수(nH)=11개, Hd=0.530±0.033이었다. 강진-영암 집단은 S=5개, π=0.00207±0.00029, nH=2개, Hd=0.515 ±0.081, 금곡 집단은 S=5개, π=0.00171±0.00019, nH=2개, Hd=0.390±0.044, 구례 집단은 S=12개, π=0.00457±0.00051, nH=5개, Hd=0.824±0.070, SK 집단은 S=13개, π=0.00307±0.00040, nH=8개, Hd=0.681±0.053이었다. 분석된 집단들 중 구례 집단은 π와 Hd가 가장 높은 수준이어서 유전적 다양성이 가장 좋은 상태로 추정되며, 강진-영암 집단은 π는 낮은 수준, Hd는 다소 높은 수준을 보였고, 금곡 집단은 π와 Hd가 모두 낮은 상태를 나타내었강진-영암, 금곡, 구례 집단과 SK 집단을 종합하여 분석한 결과, 우리나라 전체 집단의 π=0.00233±0.00019로 낮은 수준(π<0.005)을 보였고, Hd=0.530±0.033로 중간 정도(0.5≤Hd≤0.8)의 수준을 나타내었다(Nei, 1987; Rogers and Harpending, 1992). 국내 담수어류에 대한 연구에서 잔가시고기는 서식지에 따라 π=0.00000-0.00291, Hd=0.000-0.755로 매우 폭넓게 나타났고(Kim et al., 2023), 강릉 연곡천의 참갈겨니 집단은 π=0.0003, Hd=0.0904로 매우 낮은 수준이었으며(Kim et al., 2020b), 충남 예당호의 붕어 집단은 π=0.0018 ±0.0004로 매우 낮은 수준을 보였다(Kim et al., 2020a). 이러한 결과들은 우리나라 담수 척추동물의 유전적 다양성이 전반적으로 낮은 수준이며, 이번 연구에서 확인된 남생이의 낮은 유전적 다양성과도 유사한 양상이었다.
강진-영암, 금곡, 구례 집단들에 대한 중립성 시험 결과, Fu’s Fs, Tajima’s D의 수준은 강진-영암, 금곡, 구례 집단 모두 양의 값을 나타내었다(Table 1). 또한 강진-영암 집단은 Fu’s Fs, Tajima’s D 모두 유의적인 수준을 보였고(P<0.05), 금곡 집단은 Fu’s Fs는 고도의 유의적 수준(P<0.0001), Tajima’s D는 유의적인 수준을 보였다(P<0.05). 이상의 결과에서 Fu’s Fs, Tajima’s D 지수가 모두 유의적인 수준이고, 양의 값을 보인다는 점은 강진-영암 집단과 금곡 집단들은 오래지 않은 과거에 유전적인 병목(genetic bottleneck)이나 균형적 선택(balancing selection)을 겪은 이후, 현재는 집단이 성장하고 있는 것으로 추정된다. 또한 구례 집단 역시 유전적 병목이나 균형적 선택이 진행되었으나, 유의적인 수준을 보이지는 않았다(Tajima, 1989; Fu, 1997). 반면, 우리나라 전체 집단에서는 Fu’s Fs, Tajima’s D 모두 0에 가까운 값을 나타내어, 전체적으로는 유전적으로 큰 변동은 없다고 추정되지만, 서남부권에 해당하는 전라남도 강진-영암 집단과 광주광역시 금곡 집단은 유전적인 중립성이 다소 역동적인 상태라고 하겠다. Kim et al. (2023)은 낙동강 수계와 동해안 지역에서 수집한 잔가시고기 집단들은 Fu’s Fs, Tajima’s D 모두 유의적인 수준은 아니라고 하였다.
Table 1. Genetic diversity for mtDNA cytB gene sequences and haplotypes in the Reeves’ turtle populations in southwestern region of South Korea
| Population | n | S | π±SD | nH | Hd±SD | Fu’s Fs | Tajima’s D |
|---|---|---|---|---|---|---|---|
| Overall | 172 | 13 | 0.00233 ± 0.00019 | 8 | 0.530 ± 0.033 | 0.042 | 0.10149 |
| Gangjin-Yeongam | 22 | 5 | 0.00207 ± 0.00029 | 2 | 0.515 ± 0.081 | 4.043* | 2.14970* |
| Geumgok | 92 | 5 | 0.00171 ± 0.00019 | 2 | 0.390 ± 0.044 | 8.177** | 2.12884* |
| Gurye | 14 | 12 | 0.00457 ± 0.00051 | 5 | 0.824 ± 0.070 | 1.651 | 1.51491 |
| SK1 | 44 | 13 | 0.00307 ± 0.00040 | 8 | 0.681± 0.053 | -0.429 | 0.13001 |
Sample size (n), number of variable sites (S), nucleotide diversity (π), number of haplotypes (nH), haplotype diversity (Hd) and standard deviation (SD).
* Significant Tajima’s D and Fu’s Fs values (P<0.05).
** Significant Tajima’s D and Fu’s Fs values (P<0.0001).
1, mtDNA cytB sequences of the South Korean Reeves’ turtles previously reported by Suzuki et al. (2011), Oh et al. (2017), and Han et al. (unpublished, NCBI direct submission).
서부권역 남생이 3개 집단과 SK 집단 사이에서의 유전적 분화도를 분석한 결과(Table 2), 금곡 집단은 구례 집단과는 중간 정도의 분화(Fst=0.10526, 0.05≤Fst≤0.15)를 보였고, 강진-구례 집단, SK 집단과는 유전적 차이가 적은 상태였다(Hudson et al. 1992). 강진-영암 집단 역시 구례 집단과 중간 정도의 분화(Fst=0.14761, 0.05≤Fst≤0.15)를 나타내었으나, 다른 집단들과의 유전적 분화가 낮은 상태로 확인되었다. Dxy (Nei, 1987)에 대한 분석 결과에서도 전체적으로 Dxy 값이 낮은 수준을 보여(Dxy<0.05), 모든 집단들 사이에서 유의적인 분화는 없으나, 구례와 금곡 간(0.00041±0.00118), 구례와 강진-영암 간(0.00059±0.00122)에는 다소 높은 수준을 보였다. 이 결과들은 남생이 3개 집단들이 우리나라의 서남부권에 위치하고, 지리적으로 서로 가까운 장소인데서 기인한 것으로 추정된다. 붕어의 경우 한국과 거리적으로 거리가 먼 중국, 일본 집단들과 유전적 분화도가 높은 수준을 보였고(Kim et al., 2020a), 잔가시고기는 지리적 거리와 수계의 영향이 모두 반영된 결과를 나타내었다(Kim et al., 2023). 한편, 중국 해안에 서식하는 굴(Crassostrea hongkongensis)에서는 지리적 거리뿐만 아니라, 해류와 반도-섬에 의한 지리적 장벽도 집단 간 유전적 분화에 영향을 준다고 제안되었다(Li et al., 2013).
Table 2. MtDNA cytB gene divergence between populations in the Reeves’ turtle populations in southwestern region of South Korea
| Geumgok | Gangjin-Yeongam | Gurye | SK1 | |
|---|---|---|---|---|
| Geumgok | -0.00002 ± 0.00053 | 0.00041 ± 0.00118 | 0.00010 ± 0.00098 | |
| Gangjin-Yeongam | -0.00984 | 0.00059 ± 0.00122 | 0.00016 ± 0.00052 | |
| Gurye | 0.10526 | 0.14761 | 0.00010 ± 0.00098 | |
| SK1 | 0.00744 | 0.06617 | 0.02605 |
Upper diagonal, average number of nucleotide substitutions per site between populations (Dxy) and its standard deviation. Lower diagonal, values represent that Fst (Hudson et al., 1992) between populations.
1, mtDNA cytB sequences of the South Korean Reeves’ turtles previously reported by Suzuki et al. (2011), Oh et al. (2017), and Han et al. (unpublished, NCBI direct submission).
이번 연구에서 새로 결정된 128개의 cytB 서열과 동북아시아, 미국에서 보고된 서열들을 모두 종합했을 때, 전체 356개 서열에서 단상체형 12개가 검출되었다(Table 3). 이중 우리나라에서는 8개, 중국과 대만은 각각 5개, 일본은 3개, 미국은 2개의 단상체형들이 검출되었다. 단상체형 중 8개(MrcytB02, MrcytB05-MrcytB09, MrcytB11-MrcytB12)는 한 나라에서만 확인되었고, 나머지 4개는 여러 나라에서 검출되었다. 이 중 MrcytB01과 MrcytB04는 우리나라, 중국, 대만, 일본, 미국 등 분석에 이용된 모든 나라에서 검출되었고, MrcytB01은 우리나라, 중국, 대만, MrcytB10은 우리나라, 일본, 대만에서 검출되었다. Oh et al. (2017)은 우리나라와 중국, 일본, 대만 등에서 수집한 cytB 서열(k=226)에서 6개의 단상체형을 확인하였으나, 이번 연구결과에서는 12개의 단상체형이 검출되고 나라별 단상체형의 수도 증가하였다. 단상체형 중에서 MrcytB01, MrcytB03, MrcytB04, MrcytB05, MrcytB10, MrcytB11는 Oh et al. (2017)의 연구결과에서 제안된 Hap04, Hap05, Hap01, Hap02, Hap03, Hap06과 일치하였고, 다른 6개는 새롭게 확인되었다. 이러한 차이는 Oh et al. (2017)의 보고 외에도 여러 나라에서 유전자 정보가 추가로 공개되었고(Spinks et al., 2004; Fong and Chen, 2010; Suzuki et al., 2011; Fuji et al., 2014; Lee et al., 2019), 이번 연구에서 분석한 염기서열의 길이가 늘어난 데에서 기인한 결과로 보인다. 그럼에도 불구하고 일본, 대만, 미국에 비해, 중국과 우리나라에서 많은 단상체형이 검출되고, 고유 단상체형도 높은 빈도(중국 2개, 40%; 우리나라 4개, 50%)를 보이는 이 연구결과는 중국과 우리나라에는 고유 개체들 서식해왔다는 선행연구결과들(Avise 2000; Suzuki et al., 2011)을 뒷받침하는 근거라 하겠다. 반면, 일본과 대만에서 각각 1개씩 고유 단상체형이 발견된 것은 시료 수집의 오류에서 기인한 결과라 추정되나, 향후 폭넓은 시료 분석을 통해 연구되어야할 부분이다. 또한 이번 연구결과 역시 우리나라와 중국, 일본, 대만, 동남아시아, 미국에 이르기까지 여러 가지 목적을 위해 남생이의 국제간 교류가 예전부터 활발히 이루어져왔다는 선행 연구결과들(Spinks et al., 2004; Fong and Chen, 2010; Suzuki et al., 2011; van Dijk et al., 2012)과 유사한 결과이다.
Table 3. Geographical distribution of the mtDNA cytB haplotypes found in this study
| Haplotype | k | South Korea | China | Taiwan | Japan | USA | Oh et al. (2017) | |||
|---|---|---|---|---|---|---|---|---|---|---|
| Gangjin-Yeongam | Geumgok | Gurye | SK1 | |||||||
| MrcytB01 | 6 | 3 | 1 | 1 | 1 | Hap04 | ||||
| MrcytB02 | 1 | 1 | ||||||||
| MrcytB03 | 9 | 1 | 1 | 3 | 1 | 2 | 1 | Hap05 | ||
| MrcytB04 | 117 | 8 | 24 | 4 | 15 | 30 | 6 | 28 | 1 | Hap01 |
| MrcytB05 | 4 | 1 | Hap02 | |||||||
| MrcytB06 | 1 | 4 | ||||||||
| MrcytB07 | 2 | 2 | ||||||||
| MrcytB08 | 3 | 1 | 2 | |||||||
| MrcytB09 | 1 | 1 | ||||||||
| MrcytB10 | 212 | 14 | 68 | 5 | 21 | 1 | 103 | Hap03 | ||
| MrcytB11 | 1 | 1 | Hap06 | |||||||
| MrcytB12 | 1 | 1 | ||||||||
k, number of sequences found in each haplotype.
1, mtDNA cytB sequences of the South Korean Reeves’ turtles previously reported by Suzuki et al. (2011), Oh et al. (2017), and Han et al. (unpublished, NCBI direct submission).
남생이 cytB 12개의 단상체형들의 계통 유연관계를 살펴보기 위해 Mauremys 속의 다른 종들과 함께 NJ tree를 작성하였다(Fig. 1). 전체적으로 15개의 단상체형들은 크게 4개의 계보(MRE01-MRE04)로 구분되었고, MRE01은 3개(MrcytB01-03), MRE02는 2개(MrcytB10, MrcytB11), MRE03은 2개(MrcytB08-09), MRE04는 5개(MrcytB04-07, MrcytB12)의 단상체형들을 포함하였다. Mauremys 속의 다른 동물들과 진화적으로 가장 근연으로 보이는 MRE01 계보에는 우리나라, 중국, 대만, 일본, 미국 등 모든 나라에서 보고된 서열들이 포함되어 있었고, MRE02에는 우리나라, 일본의 서열들과 대만의 서열 1개, MRE03에는 우리나라 서열들로 구성된 단상체형 2개, MRE04에는 중국, 대만, 일본, 미국, 우리나라의 단상체형 서열들이 모두 확인되었다.
우리나라와 중국의 단상체형들을 비교했을 때, 두 나라에서 모두 확인된 단상체형은 MrcytB01, MrcytB03-04 등 3개였고, 이들 단상체형에 해당하는 서열들을 보유한 남생이 개체들은 한반도에서 수집되었더라도, 아마도 중국의 남생이와 연관된 개체 또는 이들의 후손일 가능성이 높다고 하겠다. Oh et al. (2017) 역시 우리나라-중국이 연관된 단상체형이 3개이며, 이들 중 Hap01 (Table 1, MrcytB04)은 중국 계보로 판독하였다. 일본과 대만, 미국 모두 여러 개의 단상체형이 검출되고(Table 1), 계통수 상에서 다양한 위치에서 발견되고 있다(Fig. 1). 하지만, 일본, 대만, 미국은 화석이나 과거의 서식 기록이 없고, 일본은 중세 이후 조선과 중국에서, 대만은 오랜 역사 동안 수차례에 걸쳐 주로 중국에서 남생이를 비롯한 다양한 담수거북들이 유입되었고, 미국은 20세기에 아시아에서 전파된 것으로 추정하고 있다(Spinks et al., 2004; Suzuki et al., 2011; Fuji et al., 2014;). 이번 연구에서 분석된 모계 계보는 Oh et al. (2017)이 제안한 2개보다 많지만, 전체적인 나라별 분포는 유사한 양상을 보였다. MRE02의 경우 고유 개체군인 우리나라와 이주 개채군인 일본과 대만에서 확인되었는데, 일본의 남생이들은 중국, 한반도에서 19세기 이전부터 도입되었다는 연구결과들(Suzuki et al., 2011, 2014)과 동일한 결과이다. 대만 본섬의 남생이는 고유 개체군이 아닐 가능성이 더 높으나, 중국 대륙과 2km 거리인 Kinmen Island의 개체군은 고유 개체군으로 강력한 보호를 받고 있다. 하지만, 한약재로 많은 거북류를 중국과 동아시아 등지에서 수입하고 있으며, 인위적인 도입과 다른 종과의 교잡 등이 고유 개체군 보호에 걸림돌로 간주되고 있다(Chen et al., 2009; Chen and Lue, 2010; Fong and Chen, 2010; Lee et al., 2019). 계통수 상에서 나라별 단상체형의 위치를 살펴볼 때, MRE01과 MRE04 계보는 중국 기원의 모계, MRE02, MRE03은 한반도 기원의 모계 계보로 보이나, MRE02에서 MrcytB10 단상체형(대만 1개 서열)이 함께 발견된다는 점은 향후 동아시아 전체의 자연 집단들을 대상으로 한 개체군 이주에 대한 광범위한 유전자 정보 분석이 필요함을 시사하고 있다.
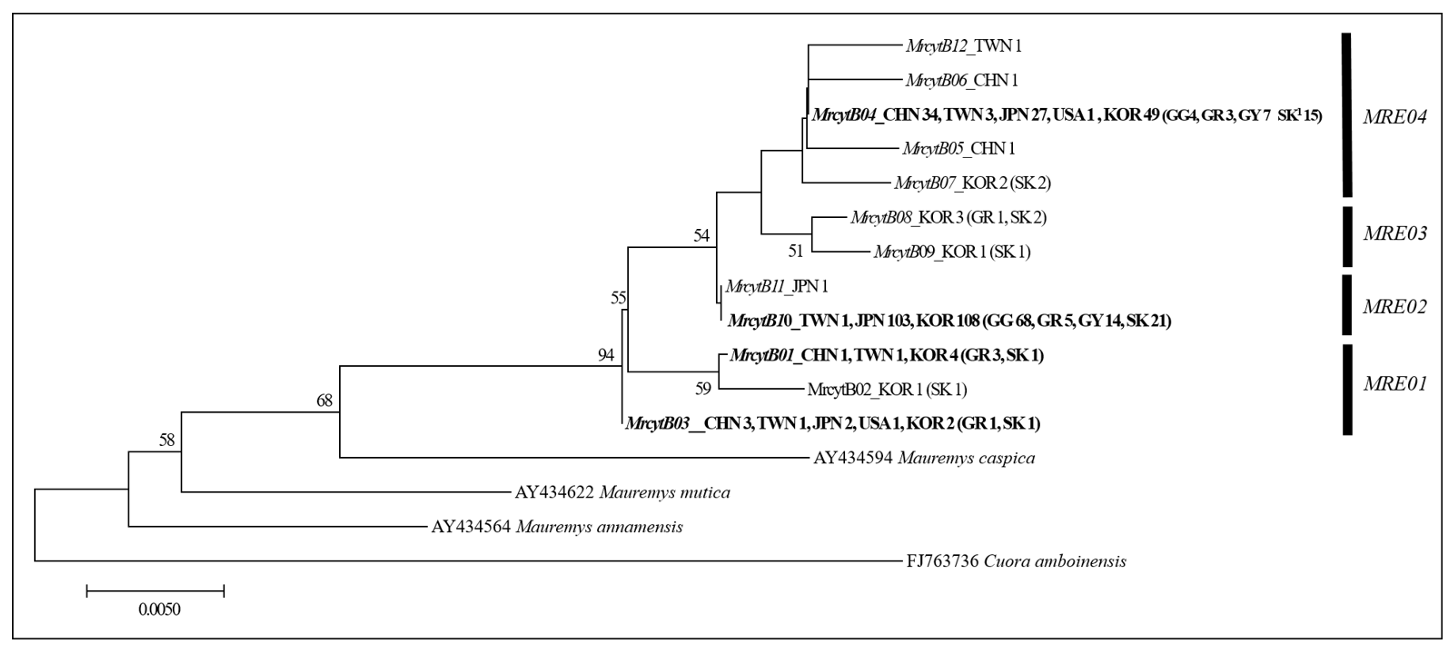
Figure 1. Neighbor-joining tree for twelve haplotype sequences of the Mauremys reevesii mtDNA cytB gene newly determined in this study and those previously reported in NCBI nucleotide database. Haplotype codes are given in the Table 3. Maternal lineages MRE01-MRE04 were designated in this study. Abbreviations of country: CHN, China; JPN, Japan; KOR, South Korea; TWN, Taiwan; USA, the United States of America. Abbreviations of population: GG, Geumgok; GR, Gurye; GY, Gangjin-Yeongam; SK, mtDNA cytB sequences of the South Korean Reeves’ turtles previously reported by Suzuki et al. (2011), Oh et al. (2017), and Han et al. (unpublished, NCBI direct submission).
우리나라 서남부권 남생이 3개 집단(강진-영암, 금곡, 구례)의 유전적 다양성은 염기서열 다양도와 단상체형 다양도가 중간 정도이거나 낮은 수준이었고, 중립성 시험에서도 좋지 않은 상태임을 확인하였다. 한편, 이번 우리나라 집단들에서 일부이지만 중국 기원인 단상체형들이 발견되기 때문에, 이들을 제외하고 한반도 고유 단상체형을 보유한 개체들의 유전적 다양성을 평가한다면, 강진-영암, 금곡 집단 모두 염기서열 다양도와 단상체형 다양도가 0.000에 가까워, 유전적 다양성이 매우 낮은 상태라고도 평가할 수 있다. 구례 집단 역시 한반도 기원 단상체형은 2개이므로 유전적 다양성이 낮은 상태라 하겠다. 다시 말해서, 한반도 기원 단상체형만을 놓고 봤을 때, 우리나라 서남부권 남생이의 유전적 다양성은 매우 낮으며, 중국 기원인 개체(또는 그 후손)들의 유전적 이입(genetic introgression)에 의해 교란되어 있는 상태라고도 할 수 있다. 이번 연구에서 cytB 유전자의 다형성만을 살펴보았기 때문에 보다 포괄적인 유전적 다양성을 언급하기 위해서는 향후 mtDNA 내에서 다형성이 더 높다고 알려진 control region 등 다른 유전자뿐만 아니라, 핵 DNA 서열, 미세부수체 정보에 대한 추가적인 정보들을 이용한 유전적 다양성, 집단 구조, 혈연관계 등도 분석되어야 할 것이다. 이후 남생이 개체군의 유전적 다양성의 증진을 위해 한반도 기원의 단상체형을 보유한 개체의 발굴, 서식지 간 연결성의 확보, 다양성을 증가시킬 수 있는 개체나 유전자 공급원(gene pool)의 활용 등에 대한 종합적인 검토가 진행되어야할 것이다.
No potential conflict of interest relevant to this article is reported.
이 연구는 환경부의 지원을 받아 국립공원공단 국립공원야생생물보전원의 “멸종위기종증식복원및반달가슴곰공존문화조성사업”에서 수행된 결과의 일부입니다. 연구비 지원에 감사드립니다.
Avise JC. 2000. Phylogeography: The History and Formation of Species. Cambridge, MA: Harvard University Press. p.447.
[DOI]
Baek HJ, Cheong E, Kim Y, Koo KS, Kim SH, Park CD, Yoon JD. 2024. Geographical distribution of Mauremys sinensis, Mauremys reevesii, and their hybrids in South Korea. Animals 14:2626.
[DOI][PubMed][PMC]
Brown WM, George MJr, Wilson AC. 1979. Rapid evolution of animal mitochondrial DNA. Proceeding of the National Academy of Science of the United States of America 76(4):1967-1971.
[DOI][PubMed][PMC]
Caragiulo A, Dias-Freedman I, Clark JA, Rabinowitz S, Amato G. 2014. Mitochondrial DNA sequence variation and phylogeography of Neotropic pumas (Puma concolor). Mitochondrial DNA 25(4):304-312.
[DOI][PubMed]
Carter RW. 2007. Mitochondrial diversity within modern human populations. Nucleic Acids Research 35(9):3039-3045.
[DOI][PubMed][PMC]
CHA (Cultural Heritage Administration). 2006. Yearbook of Cultural Heritage 2006. Cultural Heritage Administration. Seoul. pp.51-59.
Chen TH, Chang HC, Lue KY. 2009. Unregulated trade in turtle shells for Chinese medicine in East and Southeast Asia: the case of Taiwan. Chelonian Conservation and Biology 8(1):11-18.
[DOI]
Chen TH, Lue KY. 2010. Population status and distribution of freshwater turtles in Taiwan. Oryx 44(2):261-266.
[DOI]
Clayton DA. 1982. Replication of animal mitochondrial DNA. Cell 28:693-705.
[DOI][PubMed]
CITES (Convention on International Trade in Endangered Species of Wild Fauna and Flora). 2015. Appendices I, II and III. <https://cites.org/eng/app/appendices.php>
Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39:783-791.
[DOI][PubMed]
Fong JJ and Chen TH. 2010. DNA evidence for the hybridization of wild turtles in Taiwan: possible genetic pollution from trade animals. Conservation Genetics 11:2061-2066.
[DOI]
Fu YX. 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics 147:915-925.
[DOI][PubMed][PMC]
Fuji R, Ota H, Toda M. 2014. Genetic andmorphological assessments of hybridization between two non-native Geomydid turtles, Mauremys reevesii and Mauremys mutica, in Northcentral Japan. Chelonian Conservation and Biology 13(2):191-201.
[DOI]
Giles RE, Blanc H, Cann HM, Wallace DC. 1980. Maternal inheritance of human mitochondrial DNA. Proceeding of the National Academy of Science of the United States of America 77:6715-6719
[DOI][PubMed][PMC]
Gonzalez-Oliver A, Pineda-Vazquez D, Garfias-Morales E, La Cruz-Laina I, Medrano-Gonzalez L, Marquez-Morfin L, Ortega-Munoz A. 2018. Genetic overview of the Maya populations: mitochondrial DNA haplogroups. Human Biology 90(4):281-300.
[DOI][PubMed]
Harris DJ, Arnold EN, Thomas RH. 1998. Relationships of lacertid lizards (Reptilia: Lacertidae) estimated from mitochondrial DNA sequences and morphology. Proceeding of the Royal Society B Biological Sciences 265:1939-1948.
[DOI][PubMed][PMC]
Hudson RR, Slatkin M, Maddison WP. 1992. Estimation of levels of gene flow from DNA sequence data. Genetics 132:583-589.
[DOI][PubMed][PMC]
IUCN (International Union for the Conservation of Nature). 2015. Mauremys reevesii. The IUCN red list of threatened species. version 2015.2.
Iverson JB, Brown RM, Akre TS, Near TJ, Le M, Thomson RC, Starkey DE. 2007. In search of the tree of life for turtles. Chelonian Research Monographs 4:85-106.
Jo, SI, Na S, An CK, Kim H, Jeong YJ, Lim YM, Kim SD, Song JY, Yi H. 2017. Comparison analysis for using the habitat pattern between the Korean endangered species, Mauremys reevesii, and the exotic species, Trachemys scripta elegans. Korean Journal of Environment and Ecology 31:397-408.
[DOI]
Jung YJ, Kim IH, Park D. 2016. The possibility assessment of age estimation of the endangered Reeves’ turtle (Mauremys reevesii) based on the number if growth lines on dorsal scutes. Korean Journal of Environment and Ecology 30(6):970-976.
[DOI]
Kim GW, Joe SD, Kim HY, Park HB. 2020a. Phylogenetic analysis of Carassius auratus and C. cuvieri in Lake Yedang based on variations of mitochondrial CYTB gene sequences. Journal of Life Science 30(12):1063-1069.
Kim KR, Sung MS, Hwang Y, Lee MS, Jeong JH, Kim H, Yu JN. 2023. Analysis of genetic diversity across newly occupied habitats within the Goryeong population of Pungitius kaibarae using the mitochondrial Cytb gene. Korean Journal of Ichthyology 35(4):217-223.
[DOI]
Kim YR, Jang JE, Choi H, Lee HJ. 2020b. Phylogeographic and population genetic study of Korean endemic freshwater fish species, Zacco koreanus. Korean Journal of Environmental Biology 38(4):650-657.
[DOI]
Kim SR, Lee JH, Song JY, Chang MH, Sung HC, Cho DG. 2013. A study on the habitat restoration model for Chinemys reevesii. Journal of Korean Environment Research and Technology 16(2):115-125.
[DOI]
Kimura M. 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution 16:111-120.
[DOI][PubMed]
Kumar S, Stecher G, Tamura K. 2016. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular Biology and Evolution 33:1870-1874.
[DOI][PubMed][PMC]
Koo KS, Jang MH, Kim DI, Kim SH, Baek HJ, Sung HC. 2019. Report on the large population and habitat status of endangered species, Mauremys reevesii Gray 1831 (Reptilia; Testudines; Geomydidae) in South Korea. Korean Journal of Environment and Ecology 33(4):402-407.
[DOI]
Koo KS, Song JY. 2018. Effect of individual breeding and feeding on the growth of juvenile Mauremys reevesii. Korean Journal of Ecology and Environment 51(2):168-173.
[DOI]
Koo KS, Song JY, Chang MH. 2015. Mating behavior of Reeves’ turtle, Mauremys reevesii. Journal of National Park Research 6(3):111-114.
Laxmivandana R, Vashi Y, Kalita D, Banik S, Sahoo NR, Naskar S. 2022. Genetic diversity in mitochondrial DNA D-loop region of indigenous pig breeds of India. Journal of Genetics 101:5.
[DOI][PubMed]
Li L, Wu X, Yu Z. 2013. Genetic diversity and substantial population differentiation in Crassostrea hongkongensis revealed by mitochondrial DNA. Marine Genomics 11:31-37.
[DOI][PubMed]
Lee Y, Lin JW, Tseng SP, Chen TS, Lin SM. 2019. Human disturbance as a possibe cause of genetic intorgeression from exotic into native Mauremys turtles. Animal Conservation 22(6):556-567.
[DOI]
Lovich JE, Ernst CH, Gotte SW. 1985. Geographic variation in the Asiatic turtle Chinemys reevesii (Gray) and the status of Geoclemys grangeri Schmidt. Journal of Herpetology 19(2):238-245.
[DOI]
Lovich JE, Yasukawa Y, Ota H. 2011. Mauremys reevesii (Gray 1831)-Reeves’ Turtle, Chinese Three-keeled Pond Turtle. In: Rhodin AGJ, Pritchard PCH, van Dijk PP, Saumure RA, Buhlmann KA, Iversion JB, Mittermeier RA. (Eds.). Conservation Biology of Freshwater Turtles and Tortoises: A Compilation Project of the IUCN/SSC Tortoise and Freshwater Turtle Specialist Group. Chelonian Research Monographs No. 5, pp.050.1-050.10.
[DOI]
MOE (Ministry of Environment). 2017. Wildlife Conservation and Management Act.
Nabholz B, Glemin S, Galtier N. 2009. The erratic mitochondrial clock: variations of mutation rate, not population size, affect mtDNA diversity across birds and mammals. BMC Ecology and Evolution 9:54.
[DOI][PubMed][PMC]
Nei M. 1987. Molecular Evolutionary Genetics. New York, NY: Columbia University Press.
[DOI]
NIBR (National Institute of Biological Resources). 2011. Red data book of endangered amphibians and reptiles in Korea. p.125.
Oh HS, Hong C. 2007. Current conditions of habitat for Rana catesbeiana and Trachemys scripta elegans imported to Jeju-do, including proposed management plans. Korean Journal of Environment and Ecology 21(4):311-317.
Oh HS, Park SM and Han SH. 2017. Mitochondrial haplotype distribution and phylogenetic relationship of an endangered species Reeve’s turtle (Mauremys reevesii) in East Asia. Journal of Asia-Pacific Biodiversity 10:27-31.
[DOI]
Rozas J, Ferrer-Mata A, Sanchez-DelBarrio JC, Guirao-Rico S, Librado P, Ramos-Onsins SE, Sanchez-Garcia A. 2017. DnaSP v6: DNA sequence polymorphism analysis of large datasets. Molecular Biology and Evolution 34:3299-3302.
[DOI][PubMed]
Saitou N, Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution 4:406425.
Song JY. 2007. Current status and distribution of reptiles in the Republic of Korea. The Korean Journal of Environmental Biology 25(2):124-138.
Spinks PQ, Shaffer HB, Iverson JB, McCord WP. 2004. Phylogenetic hypotheses for the turtle family Geoemydidae. Molecular Phylogenetics and Evolution 32(1):164-182.
[DOI][PubMed]
Suzuki D, Ota H, Oh HS and Hikida T. 2011. Origin of Japanese populations of Reeves’ pond turtle, Myauremys reevesii (Reptilia: Geoemydidae), as inferred by a molecular approach. Chelonian Conservation Biology 10(2): 237-249.
[DOI]
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics 123:585-595.
[DOI][PubMed][PMC]
Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: improving the sensitivity of progressive sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research 22:4673-4680.
[DOI][PubMed][PMC]
van Dijk PP. 2011. Mauremys reevesii. The IUCN Red List of Threatened Species 2011:e.T170502A97431862.
van Dijk PP, Iversion JB, Shaffer HB, Bour R, Rhodin AGJ. 2012. Turtles of the World, 2012 update: annotated checklist of taxonomy, synonymy, distribution, and conservation status. Chelonian Research Monographs 5:243-328.
[DOI]
Xia X, Wang L, Nie L, Huang Z, Jiang Y, Jing W and Liu L. 2011. Interspecific hybridization between Mauremys reevesii and Mauremys sinensis: evidence from morphology and DNA sequence data. African Journal of Biotechnology 10(35):6716-6724.
Xu H, Yang X, Ni W, Liu F, Chen H, Zhu X and Liu X. 2023. Genetic diversity and genetic structure of selected generations of Chinese pond turtle (Mauremys reevesii) based on microsatellite DNA. Journal of Dalian Ocean University 38(5):812-818.