서론
한우 개량의 역사를 살펴보면 1960년대 전국 한우 챔피온 대회를 시작으로 1987년에 이르러 후대검정을 통해 씨수소를 선발하기 시작하면서 본격화 되었다(조 등, 2014). 이후 1995년 축산물등급판정제도의 전국 시행은 한우의 개량목표가 육량 위주에서 육량과 육질을 동시에 고려하게 되는 중요한 계기가 되었으며 이와 동시에 개체모형(Animal model)을 유전능력평가에 적용함으로써 육종가(breeding value)를 이용하여 씨수소를 선발하는 등 한우개량의 큰 전환점을 맞이하였다. 1999년부터는 일반 한우농가에서 능력을 고려하여 정액선택을 하도록 정액등급 제도를 시행하였으나 개체별 부족한 능력을 보완하기 보다는 단순히 등급위주로 정액을 선택하는 경향이 두드러졌고, 2000년대 후반에 들어 각 개량기관들에서 개체별 능력과 혈연관계에 근거한 근친도를 함께 고려한 계획교배 서비스를 실시함에 따라 농가 수준의 암소개량이 이뤄지기 시작하였다. 이에 발맞춰 2010년을 시작으로 지역별 한우 농가 조합을 중심으로 참여농가 암소의 혈통과 능력을 직접 관리하여 고능력 암소를 선발하고 저능력우를 도태할 수 있도록 지원하고, 암소의 유전능력과 근친도에 따라 정액을 선택할 수 있도록 계획교배 지침을 제공하는 한우암소검정사업이 운영되고 있으며, 현재 69개 한우조합 보유 암소 약 25만두 가량이 사업에 참여하고 있다(국립축산과학원, 2020). 이렇듯 한우 개량의 역사는 길지만 유전능력을 평가하여 씨수소를 선발하고 농가에서 씨수소와 암소의 능력에 따라 계획교배를 실시하는 등 집약적으로 개량이 이뤄진 기간은 그리 길지 않다고 할 수 있다.
한우는 당대검정을 거쳐 매년 66두의 후보씨수소를 선발하고 이 씨수소들의 자손의 능력검정을 통하여 최종 30두의 보증씨수소를 선발함으로써 능력이 우수한 씨수소 정액을 농가에 보급하고 있다. 또한 유전적 다양성 축소를 방지하기 위해 씨수소 한 마리당 생애 최대 총 10만 스트로(str) 미만으로 제한하고 있다. 그럼에도 불구하고 특정 씨수소 정액만을 선호하는 농가들이 늘고 있고 적은 두수의 씨수소 선발로 인해 세대 당 근친된 개체가 나타날 확률이 높아지고(Weigel, 2001) 있다는 문제가 제기되고 있다. 가축 개량에 있어서 반복적인 선발과 교배 과정은 집단의 유전자형 빈도를 지속으로 변화시키는 요인 중의 하나이다. 특히 한우와 같이 국내에서만 사육하고 있는 축종의 경우 한정된 집단 내에서 선발과 교배가 반복적으로 이뤄지기 때문에 유효집단크기 역시 작아져 유전적 다양성도 함께 축소될 가능성이 크고 이로 인해 근교퇴화가 일어나 성장, 비유, 생산 및 생존 능력 등의 감소가 일어날 가능성 또한 크기 때문에 지속적으로 모니터링하고 관리 할 필요가 있다(Leroy, 2014).
한우의 혈통 또는 유전체 정보를 이용하여 유전적 다양성을 구명하고 유효집단 크기를 추정하는 연구는 이미 여러 학자들에 여러 차례 수행된 바 있다(당 등, 2011; 송 등 2018, 조 등, 2014; Edea et al., 2020; Lee et al., 2011; Li and Kim, 2015; Kim et al., 2018). 하지만, 대부분 씨수소 또는 씨수소 선발을 위한 당·후대 검정집단 자료를 주로 연구에 이용하였고 농가단위 개량을 위한 암소 집단에 관한 연구는 조 등(2014)의 연구보고를 제외하고는 실시된 바가 없다. 조 등(2014)의 연구는 한우암소검정사업 초기의 자료를 이용하였기 때문에 본격적인 개량사업 이후에 지속적으로 축적된 혈통자료를 이용한 유전적 다양성 변화를 살펴보기에는 부족함이 많을 것으로 사료된다.
따라서, 본 연구는 한우암소검정사업이 시작한 지 10년이 지난 현 시점에서 개량사업에 참여하는 암소집단의 혈통정보를 이용하여 암소검정사업에 따른 혈통완성도, 근교계수 변화뿐만 아니라 한우 유효집단크기의 변화를 살펴봄으로써 향후 씨수소 선발 사업, 암소 개량방향 설정 등의 우리나라 한우개량 사업의 개량방향 설정을 위한 기초자료로 활용하기 위하여 실시하였다.
재료 및 방법
1. 공시자료
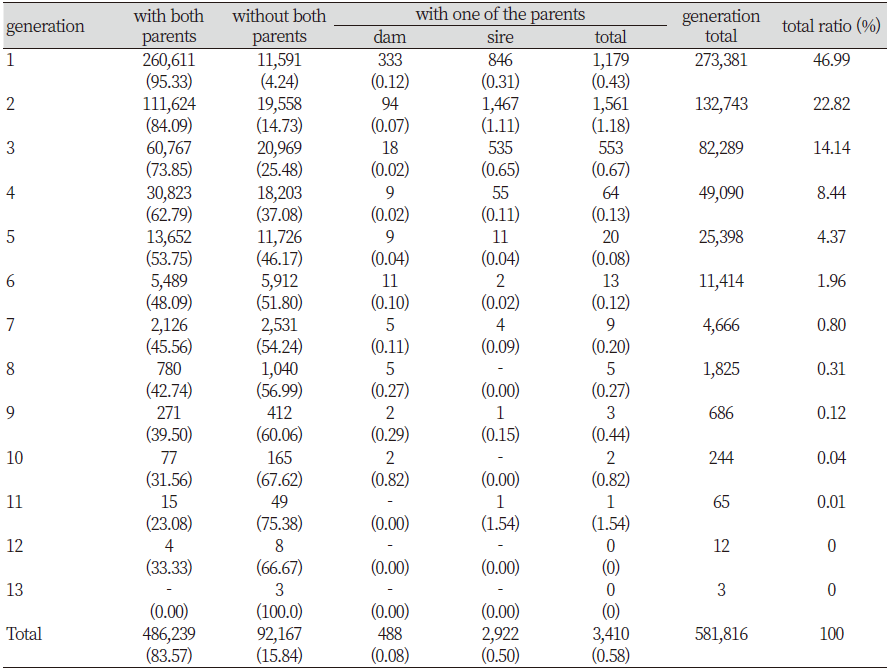
본 연구에는 2020년 현재 한우암소검정사업에 참여하고 있는 암소개체 274,347두의 자료중 혈통이 등록되어 혈통자료 조회가 가능한 273,381두를 이용하였으며, 최고 13세대까지의 혈통기록까지 조회하여 총 581,816개의 혈통자료를 분석에 이용하였다. 혈통완성도지수 분석에는 양쪽 부모(1세대)를 알고 있고, 부모는 적어도 하나의 부모를 알고 있는 개체들만 이용하였으며 총 232,461두에 대한 혈통정보를 이용하였다. 근교계수 계산에 이용한 개체는 혈통완성도지수가 떨어질 경우 불안정한 혈통으로 인해 근교계수가 0으로 나타나는 것을 감안하여 6세대까지의 혈통완성도지수(PCI5)가 0.7을 초과하는 개체들만 선별하여 총 181,853두의 혈통정보를 분석에 이용하였다. 세대간격 및 유효집단크기는 부모를 모두 알고 있는 개체들 260,611두에 대한 혈통 486,239개를 선별하여 분석에 이용하였다.
2. 분석방법
혈통완성도지수(Pedigree completeness index)는 지정한 세대까지 혈통이 얼마나 완전한지를 나타내는 지수로 아비와 어미의 혈통완성도지수에 대한 기여도로 나타낼 수 있으며 아래의 공식에 의해 계산하였다(MacCluer et al., 1983).
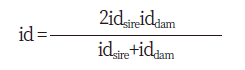
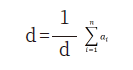
근교계수는 공통선조로부터 동일한 두 개의 유전자가 자손에게 전달될 확률로 정의를 하는데 혈연계수행렬을 이용하여 산출한다. 근교계수 알고리즘은 Meuwissen과 Luo (1992) 방법을 이용하였는데 본 방법은 혈연계수행렬의 하삼각행렬인 L 행렬에 대한 행별 원소를 계산하고 L의 각 행이 다른 행에 독립적으로 계산되게 구성함으로써 행렬 갱신에 적합하게 만드는 방법이다(조 등, 2014). 개체별 근교계수 계산은 김 등(2012)에 의해 개발된 ANGEL toolkit 프로그램을 이용하였다. 근교계수 결과가 0.0001 이상인 개체를 근친에 해당되는 개체로 구분하여 근친에 해당되는 개체들은 근친정도에 따라 세대별 근친정도 빈도를 비교 하였다.
세대간격은 자손이 태어났을 때 부모의 평균 나이로 아비에서 암소, 어미에서 암소까지의 경로를 이용하여 아래의 계산식으로 산출하였다.
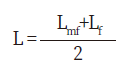
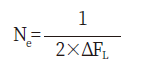
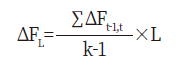 ×L
×L
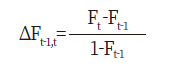
결과 및 고찰
본 연구에는 암소 273,381두 중 최대 13세대까지 혈통기록을 추적하여 자료가 조회된 581,816두에 대한 자료를 이용하였으며, 각 세대별 두수, 양친 부모를 아는 개체, 양친부모를 모르는 개체, 하나의 부모만 아는 개체들에 대한 두수를 분석하여 Table 1에 나타내었다.
세대별 두수 분포 분석결과를 보면, 1세대가 전체두수 대비 273,381두로 약 47%의 분포를 보였으며, 이 중 양친부모를 알고 있는 개체는 260,611두로 95.33%로 나타났으며, 양친부모를 모르는 개체는 11,591두 4.24%로 나타났다. 또한 한쪽 부모만 아는 개체는 1,179두였으며, 이 중 아비만 아는 개체는 846두로 0.31%, 어미만 아는 개체는 333두 0.12%로 나타났다. 2세대에서는 전체두수 대비 132,743두로 약 22.82%로 분포하고 있으며, 이 중 양친부모를 아는 개체는 111,624두로 84.09%로 나타났으며, 양친부모를 모르는 개체는 19,558두 14.73%로 나타났다. 한쪽 부모만 아는 경우도 1세대는 1,179두, 2세대 1,561두, 3세대 553두로 나타났으며, 이 중 1세대는 어미만 아는 개체 두수가 다른 세대보다 많은 것으로 나타났다.
이는 암소의 초종부 월령이 13~14개월령이고, 초산 분만 월령은 평균 28.7개월령으로 보고 된 것(Yang et al., 2017)과, 공태기간, 종부회수, 발정 재귀일수 등을 고려하면 1세대당 사육기간이 평균 3년 내외인 것으로 파악되어, 3세대는 약 10년 정도로 예측해볼 수 있다. 약 10년 전까지만 하더라도 한우 개량은 씨수소의 능력 위주의 개량으로 일반 농가에서 사육하고 있는 암소의 혈통에 대해서는 크게 중요하게 생각하지 않았다. 이에 따라 지난 10여 년간 한우암소검정사업과 혈통등록사업의 결과로 암소의 혈통자료 중 1~3세대 개체의 양친 부모를 아는 개체가 점차 증가해온 것으로 나타났고, 유전능력평가를 위한 육종가 추정(EBV)에서도 최근 출생한 암소 개체들의 혈통자료가 많이 포함된 것도 이러한 이유인 것으로 파악된다.
지정한 세대까지 혈통의 완성도를 나타내는 혈통완성도지수(PCI; Pedigree Completeness Index) 분석결과는 Table 2에 나타내었으며 1, 3, 5세대의 혈통완성도지수는 각각 PCI1, PCI3, PCI5으로 구분하여 표기하였다. PCI는 1세대(99.6%)가 가장 높았고, 그 다음으로 3세대(93.5%), 5세대(80.3%)로 순으로 분석되어 세대가 증가함에 따라 PCI가 감소하는 추세를 나타내었다. 또한 1세대의 평균 PCI는 50%를 차지한 것으로 분석되어 다른 세대 보다 높은 결과를 나타내었다. 일반적으로 이러한 결과는 모집단 간의 유전적 다양성을 측정하는데 사용되는 모수의 비교가 가능하다는 것을 나타낸다. 타 품종에 대한 연구보고와 비교해보면, Guersey 품종의 평균 PIC가 74~97%(Melka et al., 2013), 덴마크 젖소 품종의 평균 PIC가 94%(Sørensen et al., 2005)로 보고된 것에 비해서 약간 낮게 평가된 것으로 나타났다. 그리고 Holstein 품종에서 85%가 4세대 이상 완전한 혈통정보를 가지고 있다고 보고(Kearney et al., 2004)한 것과 비교하면, 한우 품종에서도 혈통완성도를 높이기 위한 노력이 필요하다고 사료된다.
|
Table 2. Pedigree completeness index(PCI) for each generation level of Hanwoo breeding population (n=232,461) 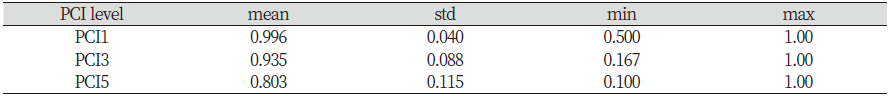
|
Table 3에는 출생연도에 따른 3세대와 5세대의 각 혈통완성도지수를 제시하였다. 출생 2010년에 3,732두로 시작하여 2018년까지 65,005두로 증가하는 것으로 추세를 나타내었으며, 2019년도에 27,073두로 개체수가 작은 것은 최근 출생한 개체들이 모두 포함되지 않았기 때문이다. 3세대와 5세대의 PIC를 비교한 결과 2010년에는 각각 85.4%, 61.1%로 나타났으며, 이후 계속 증가추세를 보여 2018년에는 각각 95.5%, 85.5%, 2019년에는 각각 95.5%, 86.9%까지 상당히 증가된 것으로 나타났다. 이는 꾸준한 혈통자료 수집을 통하여 근교계수 추정이나 유전능력평가 등의 가축개량 연구에 활용 가능한 양질의 혈통기록 자료가 축적된 결과로 판단된다.
|
Table 3. Pedigree completeness index(PCI) of PCI3 and PCI5 for each birth year of Hanwoo breeding population 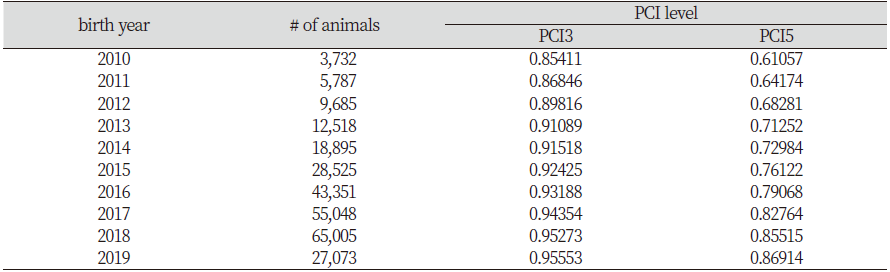
|
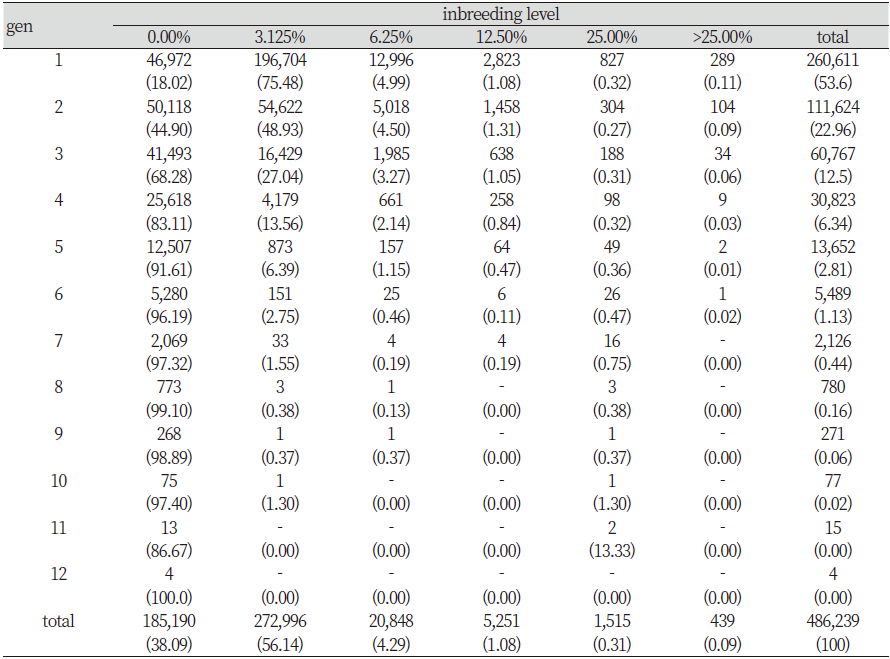
Table 4에는 부모를 모두 알고 있는 개체 260,611두에 대한 혈통 추적이 완료된 486,239두에 대하여 각 세대별 각 근교계수 구간별 근교계수 두수 및 비율을 나타내었다. 1세대부터 12세대까지 표기한 한우집단의 평균 근교계수 수준은 세대가 증가할수록 감소하는 경향을 보이고 있으며, 3세대까지 개체 두수가 전체두수 대비 89.06%로 대부분 3세대 이내의 혈통집단으로 구성되어 있는 것으로 나타났다. 3세대까지 개체 433,002두 중 근친이 0%인 개체는 138,583두로 32.01%로 나타났다. 1세대 260,611두 중 근교계수 0%인 개체는 46,972두 18.02%로 나타났고, 다음으로 근교계수 3.125% 구간에서 196,704두 75.48%로 가장 많은 두수를 나타내었다. 하지만, 고도의 근친이라 할 수 있는 25%이상 구간에서도 289두 0.11%가 나타나서, 아직까지 농가에서는 인기 정액을 이용한 지속적인 교배가 이루어지고 있음을 확인할 수 있다. 2세대는 111,624두 중 근교계수가 0% 개체는 50.118두로 44.90%로 1세대에 비해 근친이 걸리지 않은 개체의 비율이 많이 높아진 것으로 나타났고, 3세대이상 세대가 증가할수록 근친이 0%인 개체의 두수가 증가하였다. 다른 연구보고에서 시간이 지남에 따라 평균 근교수준이 증가한다고 보고한 결과와 비슷한 경향을 나타내었다(Nomura et al., 2001; Gutiérrez et al., 2003; Sørensen et al., 2005; Parland et al., 2007; Martinez et al., 2008)
Table 5에는 1980년부터 2019년까지 출생한 암소 중 부모의 혈통기록 및 생년월일 정보가 있는 486,239두에 대하여 추정한 출생년도별 한우 육종집단의 평균 근교계수, 근교된 개체에 대한 평균 근교계수 및 아비 또는 어미로부터 암송아지까지의 세대간격을 나타내었다.
|
Table 5. Change of inbreeding coefficient and generation interval for each year of Hanwoo breeding population 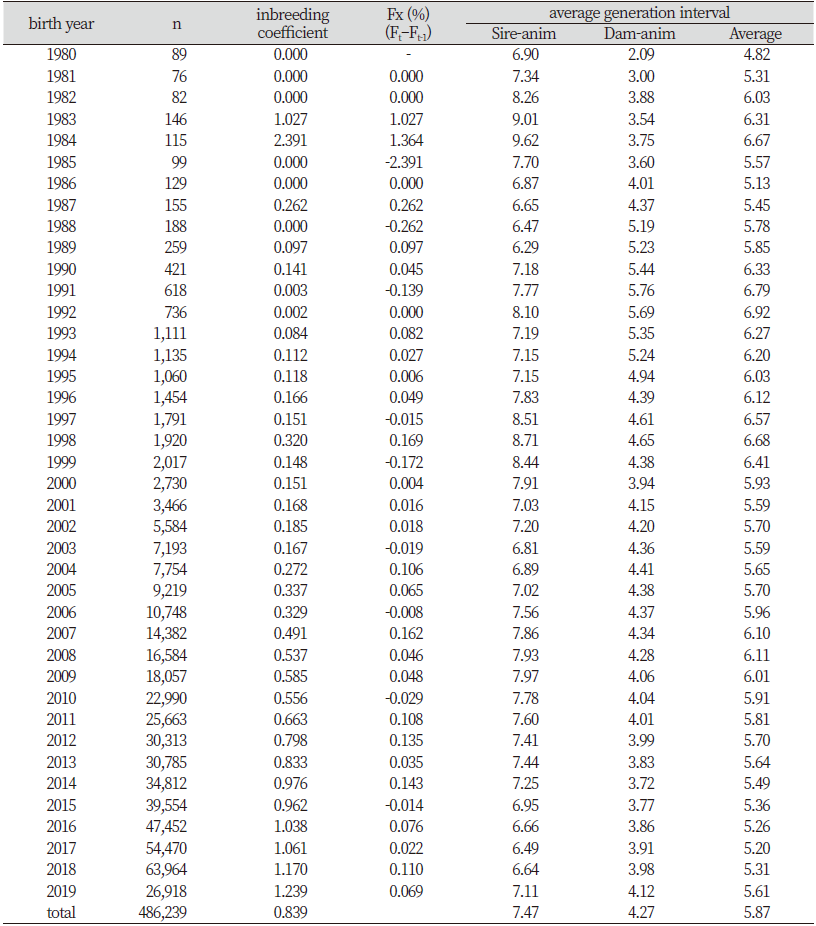
|
출생년도별 평균 근교계수는 출생년도가 지남에 따라 점차 증가하는 추세를 보였다. 출생년도에 따라 근교계수가 증가한다는 연구보고(당 등, 2011; 조 등, 2014)와 같은 결과를 나타내었다. 또한 오래전 출생한 개체일수록 혈통기록 부족으로 공통선조를 찾을 수 있는 확률이 작아진다는 연구보고와 유사한 결과를 나타내었다(Gutiérrez et al., 2003).
1990년대 이전에는 한우 혈통관리 및 자료관리가 부족하여 근교가 있어도 매우 낮게 추정되었으며 혈통등록사업(한국종축개량협회)을 통해 본격적인 혈통자료가 조사·축적되면서 1983년부터 근교계수를 확인할 수 있었다. 이와 같이 혈통정보를 이용한 근교계수는 공통선조의 혈통정보를 추적하여 이들 간 유전적 거리에 의해 추정되기 때문에 혈통완성도가 높을수록 근교계수를 정확히 추정할 수 있기 때문에 혈통정보 구축이 무엇보다 중요하다고 사료된다.
그리고 근교된 개체의 근교계수를 분석한 결과, 전체 개체를 분석한 평균 근교계수 추세와는 다르게 나타났다. 초기에는 혈통정보의 부족으로 근교가 있어도 추정되지 않거나 낮게 추정되었고, 이후 혈통정보 구축을 통해 혈통기록이 늘어남에 따라 농가에 혈통정보가 보급됨으로써 계획교배시 근친을 피할 수 있게 됨에 따라 근친을 피해 교배가 이루어져 근교계수가 크게 올라가지 않은 것으로 결과로 사료되며, 이에 따라 암소검정사업 참여농가 및 일반 한우농가에 혈통정보 및 계획교배 지침을 지속적으로 제공하는 것이 근교피해를 예방하는데 매우 중요한 부분으로 판단된다.
다음으로 아비에서 암송아지, 어미에서 암송아지, 평균에 대한 세대간격을 계산한 결과를 살펴보면, 아비에서 암송아지로 가는 세대간격은 평균 7.47년이고, 어미에서 암송아지로 가는 세대간격은 평균 4.27년으로 추정되었다. 아비에서 암송아지까지 세대간격은 현행 당·후대 검정기간이 약 5년이상 소요되며, 농가에서 인공수정할때까지 정액보관용 액체질소통에 정액을 보관하는 기간까지 고려하면 평균 7년 반 정도 소요되므로 이와 같은 결과가 나타난 것으로 판단되며, 어미에서 암송아지까지 세대간격이 짧게 나타난 것은 암소의 경우 씨수소와 같이 국가단위 능력검정을 하지 않기 때문에 비교적 낮게 추정된 것으로 사료된다(당 등, 2011; 조 등, 2014).
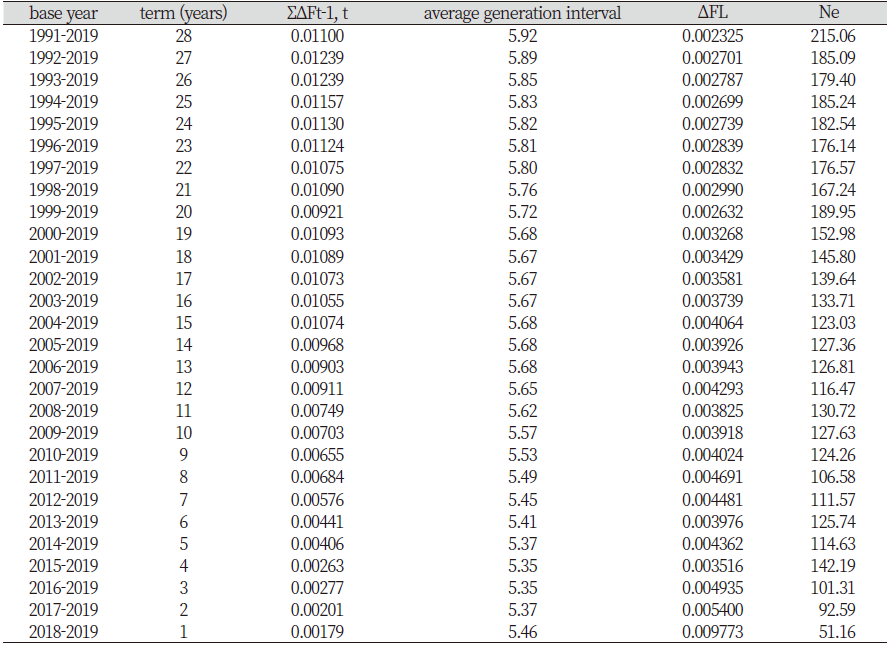
Table 6에는 한우 근교계수를 이용한 유효집단 크기의 변화를 나타내었다. 출생년도 범위 집단별 근교계수를 이용하여 유효집단의 크기를 추정한 결과, 과거 출생한 개체를 포함하는 집단에 비해 최근 출생한 개체를 포함하는 집단의 유효집단의 크기가 시간이 지남에 따라 감소하는 추세를 나타내었다. 이는 과거에서 최근으로 오면서 암소검정사업을 통해 혈통기록을 보유한 개체가 증가함에 따라 이전 혈통기록이 없어 계산되지 않았던 근교계수가 계산되어 유효집단이 감소한 것으로 사료되었으며, 제한된 농가내 개체를 활용한 국가단위 보증씨수소 선발로 인해 유전적 다양성이 감소되었다는 연구보고(조 등, 2014)와 유사한 결과를 나타내었다.
이번 연구에서 이용된 한우 암소의 유효집단크기가 51~215두 범위로 추정되어 일본 흑우의 유효집단크기 14~25두(Nomura et al., 2011), 스페인 8품종의 유효집단크기 21~123두(Gutiérrez et al., 2003), 미국 Hereford 품종에 대한 유효집단크기 85두(Cleveland. et al., 2005), 덴마크 Holstein 품종에 대한 유효집단크기 49~87두(Sørensen et al., 2005), 이탈리아 3품종 유효집단크기 122~138두(Bozzi et al., 2006), 아프리카 4품종에 대한 유효집단크기 108~165두(Maiwashe et al., 2006), 아일랜드 3품종에 대한 유효집단 64~127두(Parland et al., 2007), 콜롬비아 3품종에 대한 유효집단크기 27~143두(Martinez et al., 2008)보다 높게 추정되었다. 이는 보증 및 후보씨수소에 대해 근교를 피하기 위한 교배를 통해 평균 근교비율이 낮게 추정되어 유효집단크기가 높게 추정된 결과로 사료된다. 하지만 최근 2018에서 2019년도까지 유효집단크기는 약 51두로 낮게 추정되었는데 유효 집단크기를 씨수소 평균 근교비율로 추정하므로 최근 근교계수가 급격하게 증가했기 때문이라고 사료되며 다른 연구보고에서도 유사한 결과를 나타내었다(당 등, 2011; 조 등, 2014).
우리나라의 경우 젖소와 더불어 농가에서 유전능력이 우수하다고 알려진 특정 씨수소 정액을 선호하는 현상으로 인해 집단의 근교계수가 증가하여(Cho et al., 2017), 유효집단크기가 감소된 것으로 사료된다. 평균 근교율이 1% 증가되는 정도에서 유효집단의 크기가 50두 미만이거나 유효집단의 변이가 500두 일 때 멸종위험 수준이라는 연구보고(Franklin. 1980)에서 제안한 것을 고려하면, 그동안 국내 한우 유효집단크기와 관련된 연구보고에서 수차례 제안한 것과 같이 유효집단크기가 최근 급격하게 줄어들고 있어 한우 집단의 유전적 다양성을 유지하기 위해서는 선발 및 교배방법에 대한 검토가 반드시 이루어져야 할 것으로 사료된다.
현재 국가에서 암소검정사업을 통해 한우 농가에 개체별 혈통정보를 온·오프라인으로 지속적으로 컨설팅자료를 제공하여 근교피해를 예방하고 있으며, 특정 씨수소 정액 쏠림현상을 완화하고자 암소의 개체별 유전능력을 고려한 맞춤형 정액을 추천해주는 등 유전적 다형성에 대한 소실을 최소화하기 위한 노력을 지속적으로 기울이고 있다.
더불어 그동안의 육량, 육질형질 중심의 개량방향에서 번식형질, 사료효율, 정육율, 질병관련 형질 등으로 다양하게 수립하여 유전적으로 다양한 축군을 조성해나가면서, 단순히 보증씨수소의 두수만을 증가시키는 방법에서 나아가 암소의 경우에도 세대간격의 단축으로 유효집단크기를 높일 수 있는 암소 유전체 선발과 같은 추가 방안에 대한 검토 및 연구가 필요할 것으로 판단된다.