Introduction
돼지는 전통적으로 육류단백질의 주요공급원으로 사육되어 왔으나, 설치류모델에 비교하여 인간과의 생리학적 유사성이 높아 최근 의생명과학 분야에서의 활용성이 증가되고 있다(Bassols et al., 2014). 또한 동물자원의 국제적 이동 및 밀집사육에 의한 전염성 질환의 확산은 양돈을 포함한 동물산업 전반에 심각한 피해를 유발하고 있으며 이에 따른 개체 강건성 및 전염성 질환에 대한 가축의 저항성 향상 기술의 중요성 또한 커지고 있다(VanderWaal and Deen, 2018).
주조직적합성복합체(Major histocompatibility complex, MHC)는 적응성 면역반응을 조절하는 단백질로, 외부 항원을 인지하여 면역을 개시하는데 있어 핵심역할을 수행한다. MHC는 다양한 세포 내외부 항원을 면역 세포에 제시하여 CD8+ T 세포 혹은 CD4+ T 세포가 면역 연쇄 반응을 일으키는 데에 관여한다(Lunney et al., 2009). Zinkernagel과 Doherty (1997)에 따르면, T세포는 MHC가 제시하는 항원뿐만 아니라 항원을 결합한 MHC 분자를 또한 인지한다.
밀집사육 시스템의 현대 양돈에서 전염성 질환 확산으로 인한 생산성 및 가축 복지에 대한 문제발생, 그리고 추가적인 환경오염 등이 심각해짐에 따라 돼지 면역유전자의 유전적 특징 이해에 대한 연구의 필요성이 증가되었으며 MHC 유전자의 다형성에 대한 연구 역시 그 필요성이 점증되고 있다(Lumsden et al., 1993; Lunney et al., 2009; Lunney and Murrell, 1988).
돼지의 MHC는 Swine leukocyte antigen(SLA)이라고 하며, 크게 classⅠ과 classⅡ 분자로 분류된다(Chardon et al., 1999). SLA classⅠ은 모든 유핵 세포에서 발현되어 바이러스와 같이 세포질로부터 기원한 항원을 제시하여 주로 CD8+ T세포 면역반응을 유도한다. SLA classⅡ는 주로 대식세포나 B세포와 같은 항원제시세포(Antigen presenting cells, APCs)에서 발현되어 세포 외부에서 기원한 항원을 CD4+ T세포에 제시하여 항체생산 촉진 및 국지적 과민반응을 일으킨다(Lunney et al., 2009).
다양한 항원을 인식하고 제시하기 위하여 SLA 유전자 서열은 개체 간 변이가 매우 크다는 특성을 가진다. 이로 인해 특정 항원에 대한 개체 간 면역 반응의 차이가 발생할 수 있으며, 그 차이를 이해하기 위해서는 SLA 유전자의 다형성을 이해하는 것이 중요하다(Ando et al., 2003).
현재 전 세계 700여 종의 돼지로부터 450개의 major SLA 대립유전자 서열(class I 239개, classⅡ 211개)이 Immuno Polymrphism Database(IPD, )에 보고 되어있다(Scherf et al., 2015). 그러나 현재 인간 MHC 유전자인 Human leukocyte antigen(HLA)의 경우 총 30,039개의 대립 유전자 서열이 보고된(IPD-IMGT/HLA, ) 것과 비교할 때, 보고된 SLA 대립 유전자 데이터는 그 다형성을 이해하기에 여전히 제한적이다.
SLA 유전자 서열의 분석을 위해 다양한 방법이 개발되어왔지만 특히 유전자들 간의 서열 유사성이 높은 SLA class Ⅰ 유전자들의 경우, 원하는 유전자만을 특이적으로 증폭하는 데에 기술적 어려움이 존재했다(Shigenari et al., 2004). 뿐만 아니라, SLA class Ⅰ 유전자의 다형성을 이해하는데에 필수적인 엑손 2, 3 영역의 typing은 해당 영역의 서열 정보 부족과 더불어 매우 높은 다형성으로 인해 새로운 대립유전자의 명확한 규명에 한계가 있었다(Renard et al., 2001). 이에 본 연구팀은 앞서 SLA class Ⅰ 유전자 중 SLA-1, -2에 대한 DNA-based high-resolution typing(GSBT) 방법을 디자인하여 다양한 돼지 품종에서 유전자 다양성(gene diversity)을 입증하였다(Choi et al., 2015; Le et al., 2020). SLA class Ⅱ 유전자인 SLA-DQB1, DRB1, DQA1에 대한 comprehensive high-resolution typing의 개발과 이를 통해 식별한 새로운 대립유전자에 대한 정보 또한 보고하였다(Le et al., 2015; Le et al., 2012; Park et al., 2010; Thong et al., 2011).
본 연구에서는 기 개발된 SLA-2 타이핑 방법을 사용하여 10두의 개체로부터 추출된 염색체 DNA를 사용한 타이핑을 실시하고 그 결과를 보고하였다. 이를 통해 SLA class Ⅰ 유전자의 다형성 이해를 위한 대립유전자 데이터의 확장에 기여하고자 하였다.
Material and method
2. 1. 표본의 확보 및 DNA의 추출
PWG micropig, NIH miniature pig, Landrace, Seoul National University miniature pig (SNU)를 포함하는 총 10마리 개체의 귀, 폐, embryo 조직 약 0.5 g을 0.5% SDS와 1 mg/mL proteinase K을 함유한 lysis buffer (10 mM Tris-HCl [pH 8.0], 0.1 M EDTA) (Promega, Madison, WI, USA)와 함께 55℃에서 6시간 동안 배양한 후 phenol 추출(phenol extraction) genomic DNA를 추출하였다. 사용하기 전 모든 조직은 액체질소에 동결시킨 후 사용할 때까지 -80℃로 유지되었다. 모든 실험 절차는 건국대학교 동물보호 및 이용위원회(IACUC)의 승인 및 감독을 받았다(KU13101).
2. 2. SLA-2 유전자의 증폭 및 염기서열분석
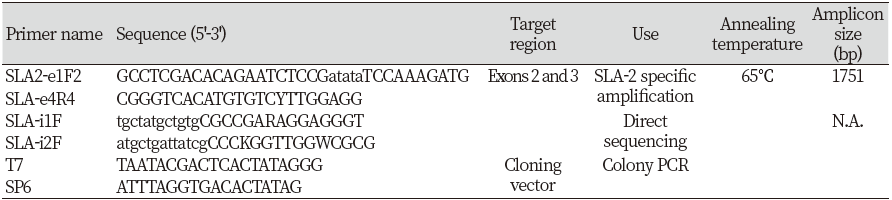
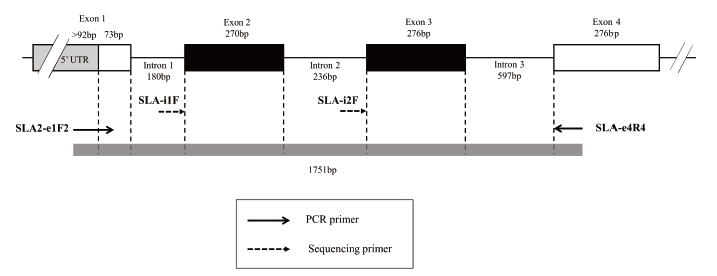
Choi 등(2015)에서 보고한 기존의 프라이머를 사용하여 SLA-2 유전자 증폭을 진행하였다(Table 1, Fig. 1). 프라이머 세트는 총 1,751 bp(base pair)를 증폭시키며 이는 인트론(intron) 1, 2, 3을 각각 180 bp, 236 bp, 597 bp와 엑손(exon) 2, 3 전체 영역을 포함한다(Fig. 1). 각 DNA 표본 당 총 반응물 20 μL 당 DNA template 40 ng, 프라이머 forward/reverse 각각 2 μL (10 pmol/μL), Taq 10X buffer 1 μL, 4X dye 2.5 μL, Supertherm Taq polymerase 0.1 μL (5 unit/μL) (JMR Holdings, Kent, UK), dNTP (1mM) 0.25μL, 나머지 용량은 3차 증류수를 첨가하여 실시하였다. 중합효소 연쇄 반응은 95℃에서 10분을 유지한 뒤 95℃에서 1분, 65℃에서 1분, 72℃에서 2분 수행하였다. 1분간 95℃를 유지하는 구간부터 2분간 72℃를 유지하는 구간을 35회 반복하였다. 그런 다음 72℃에서 5분 수행하였다.
증폭한 산물을 30 ng과 ABI PRISM BigDyeTM Terminator Cycle Sequencing Kit (Applied Biosystems)를 사용하여 제조사의 프로토콜에 따라 direct sequencing을 수행하였다. 이때 프라이머는 이전 논문을 참고하여 SLA-i1F과 SLA-i2F를 사용하였다(Choi et al., 2015) (Table 1).
2. 3. 직접염기서열분석 결과를 기반으로 한 유전자 타이핑(Genotyping)
IPD에서 SLA-2 유전자 염기 서열을 다운받아 엑손 2, 3 영역으로 구성된 참조서열(Reference sequences)를 만들었다. 그 후 본 연구를 통해 얻은 SLA-2에 대한 직접염기서열분석(direct sequencing) 결과와 참조서열을 CLC Genomics Workbench 3 (https://digitalinsights.qiagen.com; Qiagen CLC bio, Hilden, Germany)를 이용해 정렬하여, 총 10개 샘플의 SLA-2 유전자형을 판별하였다.
2. 4. Cloning을 통한 유전자형의 결정
서열 정렬 결과 중 참조서열과 일치하지 않는 대립유전자를 가지고 있거나 다수의 이형 접합 부분(Heterozygous site)를 갖고 있어 대립 유전자의 구성을 정확히 결정할 수 없는 개체의 경우, 클로닝(cloning)을 진행하여 유전자형을 결정하였다.
PCR 산물은 QIAquick Gel Extraction Kit(Qiagen, Hamburg, Germany)를 사용하여 정제되었다. 정제된 산물은 pGEM®-T Easy vector Cloning Kit (Promega, Madison, WI, USA)를 사용하여 ligation한 후 Escherichia coli DH5-α cells에 MicroPulser (Bio-Rad, Hercules, CA)를 사용하여 transfection 하였다. Transfection된 박테리아는 100 mg/mL ampicillin이 첨가된 agar plate에서 배양되었다. 그 결과 생성된 집락(Colony)을 한 배양접시 당 8개에서 10개 추출하여 이를 template으로 PCR을 수행하였다. PCR 반응물의 조성은 직접염기서열분석을 위한 PCR의 반응물과 사용한 프라이머까지 모두 같다. PCR 조건은 95℃에서 20초, 56℃에서 40초, 72℃에서 1분, 72℃에서 7분을 수행하였다. 40초간 56℃를 유지하는 구간부터 1분 간 72℃를 유지하는 구간을 30회 반복한 후 72℃에서 10분간 더 수행하였다. 그 후 PCR 산물을 1.5% 아가로스 겔(agarose gel)에 로딩하여 타겟을 확인한 뒤, 이전 과정과 같이 직접염기서열분석과 유전자 typing 과정을 시행하였다. 직접염기서열분석에서 사용한 Taq polymerase는 교정(proofreading) 기능이 없어 에러(error)를 수정할 수 없으므로 일부 염기 서열이 일치하지 않는 경우에는 직접염기서열분석 결과와 비교하여 대립유전자 구성을 찾았다.
Results and discussion
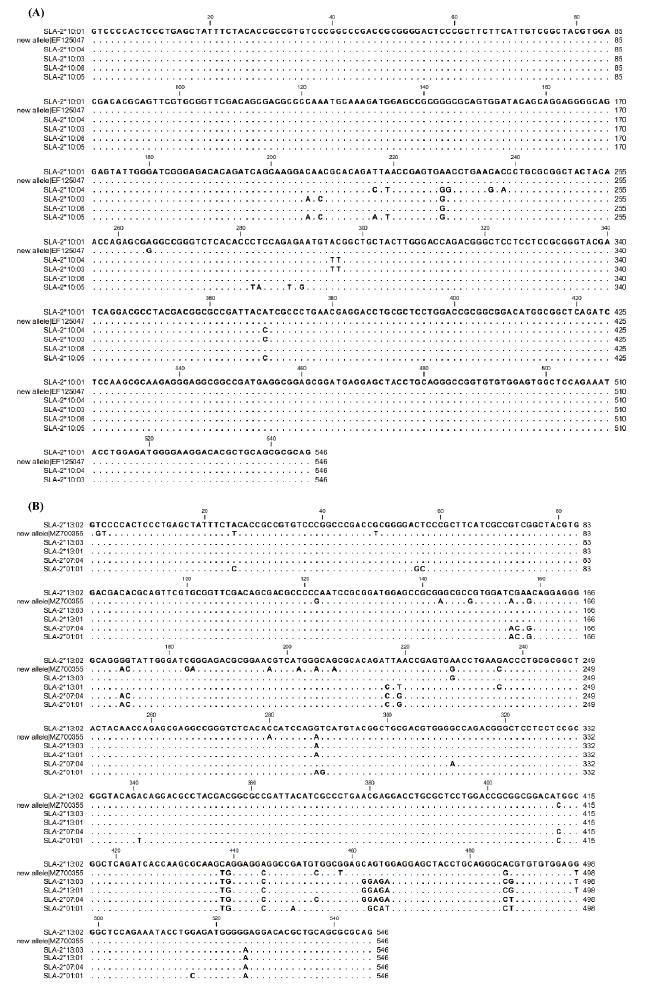
현재 SLA 유전자 명명법에 따르면 SLA class Ⅰ 유전자의 high resolution typing을 위한 최소 요건은 엑손 2와 3의 완전한 뉴클레오타이드(nucleotide) 서열이다(D.M. Smith, 2005; Ho et al., 2009). 그러므로 본 연구에서는 PCR과 직접염기서열분석법을 통해 총 10개체의 SLA-2 유전자의 엑손 2와 3 영역에 대한 typing을 실시하였다(Table 2). 그 결과 IPD에 등록되어 있지 않은 MZ700355과 EF125047 등의 2개의 대립유전자를 새로이 식별하였다. MZ700355는 기존 SLA-2 *13:02 대립유전자와 총 29 bp 염기 차를 보였으며, 본 연구를 통해 National Center for Biotechnology Information (NCBI)에 등록되었다(MZ700355) (Fig. 2). 반면에 EF125047은 기존 SLA-2*10:01과 단지 1 bp 차이가 존재하나 NCBI에 등재된 서열 중 일치하는 서열이 존재하였다. 그러나 IPD에는 등록되어 있지 않아 두 서열 모두 ISAG SLA nomenclature committee에 제출하였다.
계통수 분석에서 EF125047은 기존 SLA-2*10과 클러스터를 이루고 있으며, MZ700355은 SLA-2*13과 유사성이 가장 높았다(Fig. 4). 새롭게 식별된 두 대립유전자 서열을 아미노산으로 번역하여 계통수 분석을 위하여 현재까지 보고된 SLA-2 대립유전자의 다형성 분석이 가능한 90개의 참조 서열과 비교하였다(Fig. 3). MZ700355은 8번째 아미노산 F (Phenylalanine)과 162번째 아미노산 A(Alanine)이, EF125047는 88번째 아미노산인 G(Glycine)이 기존 보고된 대립유전자의 서열과 상이했다.
최근 20여 년 동안 세포나 장기의 이식, 질병 저항성과 면역 반응에 대한 SLA 유전자들의 영향에 대해 많은 연구가 진행되었다(D.M. Smith, 2005). SLA classical classⅠ에 속하는 SLA-1, SLA-2, SLA-3 유전자는 매우 높은 유전적 다형성을 나타내며 지금까지 각각 90, 96, 43 개의 대립유전자형이 보고되었다(IPD, https://www.ebi.ac.uk/ipd/mhc/group/SLA/). 그러나 여전히 SLA의 대립유전자형에 대한 정보는 부족한 실정이다. 이에 본 연구를 통한 새로운 대립유전자 발견과 이전 연구에서 보고한 Typing method에 대한 성공적인 입증은 새로운 SLA 대립유전자 발견을 통한 데이터의 확충뿐만 아니라, SLA 유전자의 다형성 분석의 기반이 될 수 있을 것으로 사료된다.
|
Table 2. Typing results of SLA-2 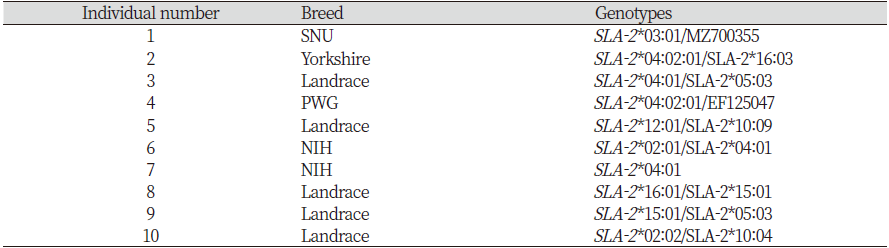
|
|
Note: SNU, Seoul national university miniature pigs; PWG, PWG Micropig. New alleles identified from individuals 1 and 4 were indicated by GenBank accession numbers. |
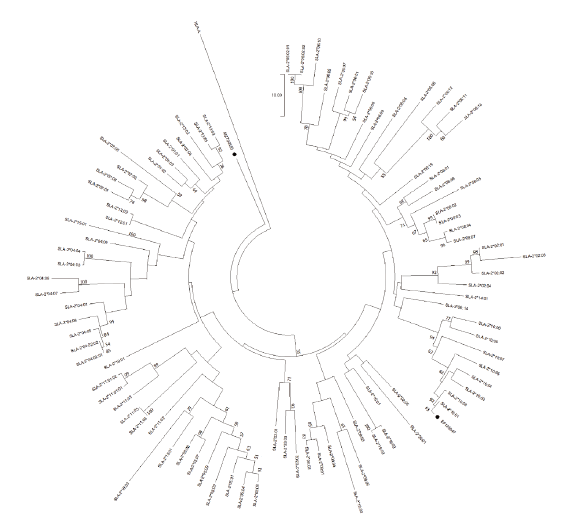
Fig. 4. The results of a phylogenetic analysis using known SLA-2 alleles present in the IPD database together with two new alleles. A phylogenetic tree was constructed using MEGA-X software with neighbor joining and a bootstrap value of 1,000. The numbers on the tree indicate bootstrap probability. New alleles were indicated by “●” symbols with the allele names. HLA-A was used as an outgroup.
Conclusion
지금까지의 연구결과들이 보여주는 SLA 대립유전자형을 규명하는 작업은 돼지의 생물의학적 모델의 측면에서 상당히 중요할 뿐만 아니라, 산업적으로도 동물의약품 개발 및 질병에 강한 돼지의 선발에 분자표지인자로 활용될 수 있음을 시사한다. 본 연구는 기개발한 타이핑 방법을 사용하여 SLA 유전자 다형성 이해를 위한 SLA-2 신규 대립유전자를 식별하여 보고하였다. 본 연구결과는 다양한 품종의 돼지에 대하여 추가적인 SLA 대립유전자의 규명을 위한 확실한 방법론적 기반을 제공할 것이라 사료되며, 새로운 SLA 대립유전자의 규명은 양돈 농가가 직면한 돼지의 강건성과 주조직적합성 복합체 다형성의 상관관계이해에 필요한 기본 자료로 활용될 수 있을 것으로 기대하는 바이다.