서론
칡소는 한우 중에서도 몸 전체에 칡 모양의 무늬를 특징으로 하는 우리나라 재래소이다. 1970년 한우심사표준에서 한우의 모색을 황갈색으로 규정함에 따라 이모색인 칡소의 수가 점점 감소하였지만, 1980년대 중반 이후부터 일부 농가에 의해 보존되어오던 소수의 개체들이 재래가축 복원사업을 계기로 보존, 증식을 통해 점점 개체 수가 증가하게 되었고, 2020년 기준으로 칡소의 개체수가 전국적으로 약 4,000두 정도 사육되고 있다(Seo et al., 2021). 칡소는 국제 식량 농업기구(FAO)에 우리나라 고유 품종으로 등록된 이후, 품종 보존을 위해 모색 관련 유전자 연구(Lee et al., 2002), 모색 분포에 관한 연구(Park et al., 2013), 체적을 통한 외모 특성에 관한 연구(Lee et al., 2016), 성장단계별 연구(Kim et al., 2014) 등 외모에 관한 연구는 활발히 이루어졌지만, 칡소의 개량에 관한 연구는 한우에 비해 미미한 상황이다. 칡소의 개량 연구가 더욱 활발히 이루어져 칡소의 유전능력을 개량하게 된다면, 농가들의 수익이 증가하고, 다양화된 소비 트렌드에 희소품종으로써의 가치가 부각되어 칡소 사육에 대한 관심이 더욱 커질 것으로 사료된다. 한우의 경우에는, 육용으로 육종목표 설정 전까지는 역용우로써 육량이나 육질에 대한 개량이 전혀 이루어지지 않았지만, 1910년대의 한우개량 관련시책을 시행한 이후, 체적이나 육량 및 육질에 대한 관심이 커져 개체 증식 및 연구가 활발하게 이루어졌다. 1961년에는 농협 인공수정소가 설립된 이후, 가축 육종에 인공수정 기법이 도입되어 유전적으로 우수한 씨수소를 선발하여 수정시키는 것이 가능해졌다. 1980년에 가축개량협의회에서 한우 씨수소를 능력검정과 후대검정을 통해 선발하도록 결정하여 검정체계가 확립되었고(Kim et al., 2014), 검정 과정에서 선발이나 계획 교배를 통한 편중을 피할 수 있고, 많은 수의 개체를 아비 개체의 자손 수와 관계없이 각 형질에 대하여 평가할 수 있는 Best Linear Unbiased Prediction (BLUP)방법을 통해 유전능력을 평가하여 고능력의 개체를 선발하게 되었다. 칡소의 유전능력을 개량을 위해서는 이미 오랫동안 연구가 활발히 진행되어온 한우의 개량체계를 이용하는 것이 바람직하지만, 칡소에 당대검정, 후대검정 등 많은 시간과 금액을 투입하는 방안은 어려울 것으로 사료된다. 하지만, 최근에 한우의 개량방법 중 방대한 후대 데이터를 활용한 전통적인 BLUP 방법보다 정확도가 높은, 유전체 정보를 이용하여 육종가를 추정하는 GBLUP 기술이 개발되어 많은 시간과 금액을 투입하지 않고 개량하는 것이 가능해졌다(Lee et al., 2013). 또한, 한우와 칡소에 관한 선행연구를 살펴보면, 한우와 칡소의 유전적 거리가 0.093±0.044, 한우와 홀스타인의 유전적 거리가 0.369±0.064로 한우와 칡소가 유전적으로 유사하다고 보고하였고(Choi, 2009), 다른 선행 연구 결과 보고서에서는 한우, 칡소, 흑우 재래 3품종 간의 FST값을 이용한 계통수를 비교했을 때, 한우와 칡소의 FST 값은 0.0116, 한우와 흑우의 FST값은 0.0614로 한우와 칡소가 유전적으로 더욱 유사하다고 보고하였다(NIAS, 2014-2018). 이러한 결과를 바탕으로 칡소와 한우가 유전적으로 유사하다고 가정할 수 있고, 한우의 유전능력 평가 체계를 칡소에 적용할 수 있을 것으로 사료된다. 또한 2020년 기준, 전국적으로 칡소의 사육 두수는 대략 4,000두 정도 사육되고 있지만, 한우에 비해 경제적인 능력이 미흡하고, 개량을 위한 기초적인 데이터도 아직은 미흡한 실정이다(Yu et al., 2021). 따라서 칡소의 사육 두수가 한우에 비해 적음에도 불구하고, 칡소의 일정 두수 이상 유전체 데이터를 전국에서 추출하여 개량을 위한 분석에 활용하게 된다면, 소규모 집단에서도 유의미한 결과가 나올 수 있을 것으로 사료된다. 본 연구는 칡소의 유전체 정보와 한우의 참조축군을 바탕으로 칡소의 유전능력을 GBLUP으로 평가하고, 칡소의 유전체 육종가 정확도에 대하여 한우의 유전능력을 평가한 선행 연구결과와 비교하여 칡소 분석결과의 유효성을 검증하고자 한다. 이 연구 결과를 바탕으로 소규모 축군인 칡소에서 효율적인 개량체계를 구축하기 위한 기초 연구자료로 활용될 수 있을 것으로 사료된다.
재료 및 방법
1. 공시재료
본 연구에 사용된 평가축군의 자료는 바이오그린21 과제를 통해 전국에서 분석된 총합 칡소 암소 564두 중 출생연도나 혈통이 등록되지 않은 64두를 제외한 칡소 암소 500두의 유전체 정보를 활용하였으며, 이에 대한 출생연도별 개체수 분포를 Table 1에 나타내었다. GBLUP 분석을 위해 구축한 참조축군의 자료는 동일 과제를 통해 전국에서 출하된 한우 거세우 13,000두의 유전체 자료를 사용하였다. 또한, 참조축군 13,000두의 표현형 정보는 축산물품질평가원 홈페이지에서 정보조회를 이용하여 성별, 출생연월일, 도축연월일, 도축장명, 도체중, 등심단면적, 등지방두께, 근내지방도 등을 수집하였고, 분석 전에 표현형 정보에서 이상치 기준에 따라 이상치를 제거하였다(Kim, 2018). 평가축군과 참조축군의 혈통정보는 (사)한국종축개량협회를 통해 KPN, 어미 개체 식별번호 등의 정보를 수집하였다. 유전자형 분석을 위한 시료는 확보된 모근에서 Genome Nucleic Acid Purification kit (MagExtractor, Toyobo CO., LTD. Osaka, Japan)을 이용해 200 ng/ul 이상의 고농도, 고순도의 DNA를 추출하였다.
2. 통계분석
SNP 유전자형 분석은 Illumina Bovine SNP50K Bead Chip version3(Illumina, SanDiego, CA, USA)을 이용하였다(40,757 SNPs). 여기에서 Minor allele frequency (MAF) 5% 미만, Missing Genotype rate 10%이상, Hardy-Weinberg equilibrium (HWE) 10 −6 미만인 데이터를 제외하고 총 40,687개의 SNP를 사용하였다.
(1) 유전체 육종가 추정
유전모수 추정은 REMLF90 (ver1.82) program을 이용해 추정해주었으며, 육종가의 추정은 BLUPF90 program을 이용하였다. 총 4개 형질에 대한 유전모수 추정을 위하여 각 형질간 유전 및 환경분산을 추정한 후, 총 4개 형질에 대한 단형질 분석을 실시하였다. 각 형질에 대한 상가적 유전효과의 유전모수 추정을 위한 선형모형을 행렬식으로 표기하면 다음과 같다.
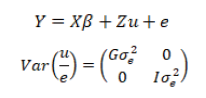
Y: 각 형질의 관측치
X: 고정효과
Z: 상가적 유전효과의 계수행렬
β: 알려지지 않은 고정효과의 추정치
u: 상가적 유전효과
e: 임의 환경효과
G: 유전체혈연관계계수 행렬
I: 대각성분이 1인 단위행렬
여기서 Y는 도체형질 관측치, X는 고정효과에 대한 계수행렬이고, β는 고정효과에 대한 추정치의 벡터, Z는 개체에 대한 임의효과의 벡터, u는 개체에 대한 추정치의 벡터, e는 임의오차 벡터이며 E(y) = Xβ, Var(u) = G = A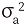 , Cov(u,e) = 0 으로가정하여 Var(y) = V = ZGZ′ + R이 된다. G는 개체간의 GRM이며,
, Cov(u,e) = 0 으로가정하여 Var(y) = V = ZGZ′ + R이 된다. G는 개체간의 GRM이며, 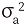 와
와 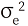 는 각각 상가적 유전분산 및 임의 환경 분산이다. 각 형질에 대한 유전체 육종가는 GRM을 통해 BLUPF90 program을 이용하여 추정하였다.
는 각각 상가적 유전분산 및 임의 환경 분산이다. 각 형질에 대한 유전체 육종가는 GRM을 통해 BLUPF90 program을 이용하여 추정하였다.
(2) 유전력 추정
BLUP방법으로 육종가 추정을 실시하였으며, 추정 육종가의 유전력은 모두 동일한 방법으로 추정하였다. 유전력 추정을 위하여 사용된 유전모수의 값은 BLUPF90 program분석 결과에서 제공되는 REMLF90 (ver1.82) program 값을 이용하여 아래와 같은 공식에 따라 추정하였다.
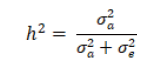
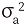 : 유전분산성분
: 유전분산성분
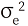 : 환경분산성분
: 환경분산성분
유전력은 전체분산 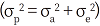 에서 유전분산이 차지하는 비율로 추정하였다.
에서 유전분산이 차지하는 비율로 추정하였다.
(3) 정확도 추정
BLUP방법으로 추전된 육종가의 정확도 값은 모두 동일한 방법으로 추정하였으며, BLUPF90 program 분석 결과 추정되는 육종가의 결과 값 중 육종가의 예측오차분산(prediction error variance, PEV)과 형질별 상가적 유전분산을 활용하여 추정하였다.
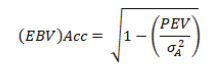
Acc: 추정된 육종가의 정확도
PEV: 추정된 육종가의 예측오차 분산
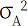 : 상가적 유전분산
: 상가적 유전분산
결과 및 고찰
칡소 500두의 유전체 육종가를 추정하기 위해 사용된 한우 참조축군과 칡소 실험축군의 표현형정보에 대한 기초통계량과 Table 2와 같다. 참조축군 13,000두는 전국에서 출하된 한우 거세우들로 구성하였으며, 도축 개월령은 평균 29개월로 도체중, 등심단면적, 등지방두께, 근내지방도의 평균 및 표준편차는 각각 445.30±46.05 kg, 97.34±11.88 cm2, 13.90±4.14 mm, 6.16±1.68점(score)으로분석되었다.유전체 참조축군의 기초 통계량을 선행 연구와 비교해보면 전국 지역에서 출하된 거세우들로 참조축군을 구성했으며 4개의 형질에 대한 평균 및 표준편차는 각각 440.68±51.46 kg, 95.79±12.02 cm2,14.36±4.85 mm, 6.05±1.84점(score)으로 보고하였다(Kim, 2021). 다른 선행 연구에서는 평균 및 표준편차를 441.21±51.53 ㎏, 95.92±12.10 cm2, 14.41±4.87 mm, 6.10±1.84점(score)으로 보고하였다(Lee, 2021). 두 집단의 수치를 비교해보면 선행연구들과 본 연구에 사용된 참조축군의 기초통계량이 유사한 수치를 나타내었다. 산업축군에서의 선행연구와 비교해보면 2006년부터 2011년까지 축산물품질평가원에서 등급판정된 거세우들의 평균 및 표준편차가 각각 437.08±50.658 kg, 91.67±10.717 cm2,14.99±5.252 mm, 6.09±1.877점(score)으로 보고하였다(Sun, 2012). 다른 선행연구에서는 경남지역 한우 거세우의 참조축군은 평균 및 표준편차가 각각 439.9±51.3 kg, 94.8±12 cm2, 14.25 mm, 6±1.9점(score)으로 보고하였다(Kim, 2020). 본 연구와 두 참조집단의 기초통계량을 비교해보면, 선행연구들과 본 연구의 참조축군 기초통계량이 유사한 수치를 나타내고 있으므로, 본 연구에 사용한 참조축군이 전국의 한우 산업축군을 평가하기에 평균 30개월령 기준의 도축개월령에 따른 도축성적을 기준으로 적합하다고 사료된다.
|
Table 2. Descriptive statistics of traits for test, reference population 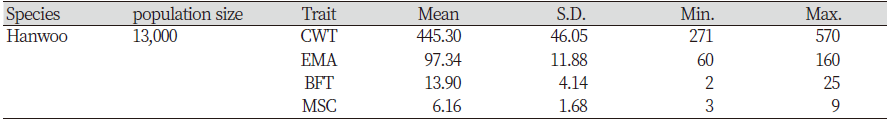
|
|
CWT: Carcass weight, EMA: Eye muscle area, BFT: Backfat thickness, MSC: Marbling score S.D.: Standard deviation |
GBLUP 분석을 위하여 칡소 암소 500두와 참조축군 13,000두의 총 13,500두에 대한 유전체 관계 행렬(GRM)을 생성하고, 추정한 유전모수 값인 유전분산, 잔차분산, 유전력의 추정값을 Table 3에 나타내었다. 유전분산은 도체중, 등심단면적, 등지방두께, 근내지방도가 각각 804.66, 50.66, 6.56, 1.19로 나타났고, 잔차분산은 각각 1148.00, 82.05, 10.73, 1.54로 나타났다. 추정된 유전분산과 잔차분산을 이용하여 유전력을 계산한 결과, 도체중, 등심단면적, 등지방두께 및 근내지방도가 각각 0.41, 0.38, 0.38 및 0.44로 추정되었다. 유전력에 관하여 본 연구와 선행연구를 비교해보면, 당대검정우 및 후대검정우를 대상으로 GBLUP방법으로 추정한 유전력이 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 각각 0.520, 0.413, 0.451, 0.434로 보고하였고(Shin et al., 2018), 다른 선행연구로는 일반 한우 농가에서 사육중인 암소를 대상으로 GBLUP방법을 사용하여 추정한 유전력이 각각 0.368, 0.339, 0.342, 0.424로 보고하였다(Lee, 2019). 본 연구의 추정된 유전력이 중간 수치를 나타낸 이유는 유전체 참조축군을 바이오그린21 과제를 통해 혈통이 관리된 거세우들로 구성하였기 때문에, 일반 한우농가보다 유전력이 높고, 당대검정우 및 후대검정우에 비해 유전력이 낮게 나온 것으로 사료된다.
|
Table 3. Heritability and Variance to GBLUP method of test population 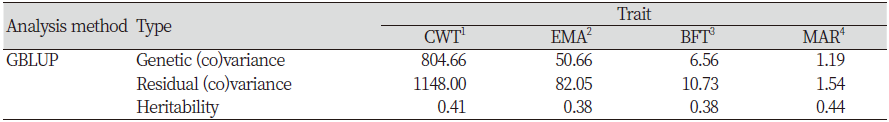
|
|
1Carcass weight, 2Eye muscle area, 3Backfat thickness, 4Marbling score, 5Standard deviation |
칡소 500두에 대한 유전체 육종가의 평균을 출생연도별로 정리하여 Table 4에 나타내었다. 연도별로 정리한 분석결과를 살펴보면 2005~2008년에는 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 육종가 평균이 -19.31, -5.70, 0.49, -0.71로 추정되었다. 2009~2011년에는 -20.62, -5.82, 0.66, -0.70으로 추정 되었으며, 2012~2015년에는 -18.35, -5.64, 0.80, -0.70으로 추정되어 출생연도별 육종가 모두 상당히 낮은 수치의 결과값이 나타났다. 그 이유는 칡소는 현재 종의 보존을 목적으로 모색에 따라 선발과 도태를 시켰기 때문에 유전능력이 개량되지 않아 육종가가 낮게 나온 것으로 사료된다. 한우도 마찬가지로 본격적인 개량체계가 도입되기 전에는 역용우로써 유전능력과 관계없이 선발이 이루어졌기 때문에 도축성적이 낮았다. 선행연구의 연도별 한우 표현형가 변화를 살펴보면 1974년부터 2007년까지 매년 거세 18개월령의 체중이 12 kg씩 증가하여 300 kg에서 600 kg에 육박하는 도축성적의 증가량을 나타냈다(Kim et al., 2014). 따라서, 칡소를 유전체 육종가를 통해 개량하여 한우와 비슷한 개량 효과가 나타날 수 있을 것으로 사료된다.
|
Table 4. Average of breeding value using GBLUP according to birth Year 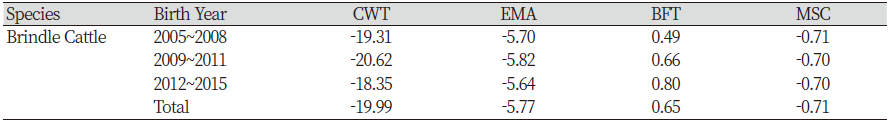
|
|
CWT: Carcass weight, EMA: Eye muscle area, BFT: Backfat thickness, MSC: Marbling score |
Table 5에는 칡소 500두 전체에 대한 도체중, 등심단면적, 등지방두께, 근내지방도의 GBLUP방법으로 추정한 유전체 육종가 평균 및 정확도를 나타냈다. 유전체 육종가 평균은 -19,99, -5.77, 0.65, -0.71로 나타났으며, 정확도의 평균은 0.44, 0.43, 0.42, 0.44로 나타났다. 선행연구와 비교해보면 GBLUP방법의 육종가를 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 2.936, 1.030, -0.165, 0.178으로, 정확도를 0.676, 0.672, 0.679, 0.709로 보고하였다(Kim, 2020). 다른 선행연구에서는 GBLUP 방법의 육종가를 6.09, 2.07, -0.29, 0.26으로, 정확도를 0.68, 0.67, 0.67, 0.70으로 보고하였다(Kim, 2021). 본 연구와 선행연구의 결과값을 비교하여 보았을 때, 선행연구들은 모두 전국에서 출하된 거세우인 산업축군을 참조축군으로 구성하여 한우 암소를 GBLUP방법으로 분석하였기 때문에 비슷한 수치를 나타내고 있었다. 본 연구는 칡소 암소를 한우의 참조축군으로 분석하였기 때문에 육종가는 각 형질에서 최대 26.08까지 차이가 나타났고, 정확도는 각 형질에서 최대 0.289까지 차이가 나타났다.
결론
본 연구결과, 칡소의 유전체 정보를 활용하여 한우 거세우를 참조축군으로 구성하여 GBLUP방법으로 유전체 육종가 및 정확도를 추정하였을 때, 칡소가 선행연구들의 한우에 비해 육종가와 정확도 모두 다소 낮은 수치를 나타내고 있었다. 그 이유는, 현재 칡소는 모색 기준으로 선발해왔으며, 유전능력에 대한 개량은 이루어지지 않고 있기 때문에 육종가가 낮은 수치를 보였으며, 참조축군을 구성할 때, 칡소가 포함되어 있지 않기 때문에 한우를 분석했을 때 보다 정확도가 낮게 나온 것으로 사료된다. 또한, 유전체를 활용한 칡소의 육종가 정확도 수치가 한우의 유전체 육종가 정확도 수치보다는 낮았지만, 한우의 혈통을 이용한 BLUP방법으로 분석하였을 때의 정확도는 선행연구에서 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 0.385, 0.402, 0.409, 0.415로 보고하였으며(Shin,2017), 다른 선행연구에서는 0.441, 0.458, 0.464, 0.472로 보고하였다(Shin et al., 2018). 본 연구와 선행연구들의 분석결과를 비교해보면, 본 연구결과와 비슷한 수치를 나타내고 있다. 따라서, 칡소의 유전체 정보를 활용하여 GBLUP방법으로 분석한 결과값이 한우의 10만 두 이상의 혈통을 가진 참조축군으로 분석한 BLUP방법의 결과값과 유사함을 알 수 있었다. 이러한 결과는 현재 칡소가 한우처럼 개량 체계나 30개월령 사양 프로그램과 같은 정형화된 사육 지표가 없고, 유전 능력이 개량되지 않았기 때문에 도축 성적이 낮아 한우의 유전체 정확도에 비해 낮은 것으로 사료된다. 또한 도축 개월령이 한우처럼 일정한 개월령에 도축을 하지 않기 때문에 정확도가 비교적 낮지만, 유전능력 평가 시스템 자체가 없는 칡소 집단에서 많은 혈통을 가진 참조축군으로 분석한 BLUP 방법의 분석 결과정도의 정확도를 나타내기 때문에, 충분히 칡소의 개량에 활용할 수 있을 것으로 사료된다. 결과적으로, 칡소의 유전체 정보를 활용하여 한우의 참조축군으로 유전능력을 평가할 수 있고, 칡소의 유전체 육종가를 활용한다면, 불필요한 암소 사육두수의 증가를 완화하고, 생산비를 절감할 수 있을 뿐만 아니라, 적은 비용으로 사육 두수가 적은 다른 소규모 농가에서도 충분히 개량의 지표로 활용할 수 있을 것으로 사료된다. 따라서 본 연구 결과는 칡소의 GBLUP방법으로 분석한 결과의 정확도가 한우 2세대 이상의 혈통을 활용한 BLUP방법으로 분석한 결과와 유사하므로, 칡소와 같은 소규모 축군에서 적은 비용으로 단기간에 개량할 수 있는 기초 연구자료로 활용될 수 있을 것으로 사료된다.




